+検索条件
-Structure paper
| タイトル | Cryo-Electron Microscopy Structures of a Campylobacter Multidrug Efflux Pump Reveal a Novel Mechanism of Drug Recognition and Resistance. |
|---|---|
| ジャーナル・号・ページ | Microbiol Spectr, Vol. 11, Issue 4, Page e0119723, Year 2023 |
| 掲載日 | 2023年8月17日 |
 著者 著者 | Zhemin Zhang / Nicholas Lizer / Zuowei Wu / Christopher E Morgan / Yuqi Yan / Qijing Zhang / Edward W Yu /  |
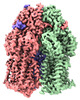
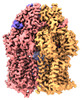
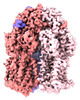
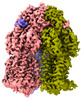
| PubMed 要旨 | Campylobacter jejuni is a bacterium that is commonly present in the intestinal tracts of animals. It is also a major foodborne pathogen that causes gastroenteritis in humans. The most predominant and ...Campylobacter jejuni is a bacterium that is commonly present in the intestinal tracts of animals. It is also a major foodborne pathogen that causes gastroenteritis in humans. The most predominant and clinically important multidrug efflux system in C. jejuni is the CmeABC (Campylobacter multidrug efflux) pump, a tripartite system that includes an inner membrane transporter (CmeB), a periplasmic fusion protein (CmeA), and an outer membrane channel protein (CmeC). This efflux protein machinery mediates resistance to a number of structurally diverse antimicrobial agents. A recently identified CmeB variant, termed resistance enhancing CmeB (RE-CmeB), can increase its multidrug efflux pump activity, likely by influencing antimicrobial recognition and extrusion. Here, we report structures of RE-CmeB in its apo form as well as in the presence of four different drugs by using single-particle cryo-electron microscopy (cryo-EM). Coupled with mutagenesis and functional studies, this structural information allows us to identify critical amino acids that are important for drug resistance. We also report that RE-CmeB utilizes a somewhat unique subset of residues to bind different drugs, thereby optimizing its ability to accommodate different compounds with distinct scaffolds. These findings provide insights into the structure-function relationship of this newly emerged antibiotic efflux transporter variant in Campylobacter. Campylobacter jejuni has emerged as one of the most problematic and highly antibiotic-resistant pathogens, worldwide. The Centers for Disease Control and Prevention have designated antibiotic-resistant C. jejuni as a serious antibiotic resistance threat in the United States. We recently identified a C. jejuni resistance enhancing CmeB (RE-CmeB) variant that can increase its multidrug efflux pump activity and confers an exceedingly high-level of resistance to fluoroquinolones. Here, we report the cryo-EM structures of this prevalent and clinically important C. jejuni RE-CmeB multidrug efflux pump in both the absence and presence of four antibiotics. These structures allow us to understand the action mechanism for multidrug recognition in this pump. Our studies will ultimately inform an era in structure-guided drug design to combat multidrug resistance in these Gram-negative pathogens. |
 リンク リンク |  Microbiol Spectr / Microbiol Spectr /  PubMed:37289051 / PubMed:37289051 /  PubMed Central PubMed Central |
| 手法 | EM (単粒子) |
| 解像度 | 3.08 - 3.44 Å |
| 構造データ | EMDB-40091, PDB-8gjj: EMDB-40092, PDB-8gjk: EMDB-40093, PDB-8gjl: EMDB-40177, PDB-8gk0: EMDB-40179, PDB-8gk4: |
| 化合物 |  ChemComp-ZZ7: 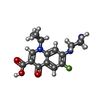 ChemComp-CPF: 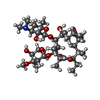 ChemComp-ERY: 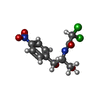 ChemComp-CLM: |
| 由来 |
|
 キーワード キーワード | MEMBRANE PROTEIN / efflux pump |
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について