+検索条件
-Structure paper
| タイトル | Direct observation of backtracking by influenza A and B polymerases upon consecutive incorporation of the nucleoside analog T1106. |
|---|---|
| ジャーナル・号・ページ | Cell Rep, Vol. 42, Issue 1, Page 111901, Year 2023 |
| 掲載日 | 2023年1月31日 |
 著者 著者 | Tomas Kouba / Anna Dubankova / Petra Drncova / Elisa Donati / Pietro Vidossich / Valentina Speranzini / Alex Pflug / Johanna Huchting / Chris Meier / Marco De Vivo / Stephen Cusack /    |
| PubMed 要旨 | The antiviral pseudo-base T705 and its de-fluoro analog T1106 mimic adenine or guanine and can be competitively incorporated into nascent RNA by viral RNA-dependent RNA polymerases. Although ...The antiviral pseudo-base T705 and its de-fluoro analog T1106 mimic adenine or guanine and can be competitively incorporated into nascent RNA by viral RNA-dependent RNA polymerases. Although dispersed, single pseudo-base incorporation is mutagenic, consecutive incorporation causes polymerase stalling and chain termination. Using a template encoding single and then consecutive T1106 incorporation four nucleotides later, we obtained a cryogenic electron microscopy structure of stalled influenza A/H7N9 polymerase. This shows that the entire product-template duplex backtracks by 5 nt, bringing the singly incorporated T1106 to the +1 position, where it forms an unexpected T1106:U wobble base pair. Similar structures show that influenza B polymerase also backtracks after consecutive T1106 incorporation, regardless of whether prior single incorporation has occurred. These results give insight into the unusual mechanism of chain termination by pyrazinecarboxamide base analogs. Consecutive incorporation destabilizes the proximal end of the product-template duplex, promoting irreversible backtracking to a more energetically favorable overall configuration. |
 リンク リンク |  Cell Rep / Cell Rep /  PubMed:36596301 PubMed:36596301 |
| 手法 | EM (単粒子) |
| 解像度 | 2.34 - 3.12 Å |
| 構造データ | EMDB-14144, PDB-7qtl: EMDB-14222, PDB-7r0e: EMDB-14240, PDB-7r1f: EMDB-14857, PDB-7zpl: EMDB-14858, PDB-7zpm: EMDB-15984, PDB-8bdr: EMDB-15996, PDB-8be0: EMDB-16006, PDB-8bek: EMDB-16013, PDB-8bf5: |
| 化合物 |  ChemComp-2TM:  ChemComp-MG: 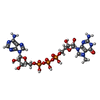 ChemComp-GTA:  ChemComp-HOH: 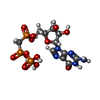 ChemComp-G2P: |
| 由来 |
|
 キーワード キーワード | VIRAL PROTEIN / Influenza / viral RNA-dependent RNA polymerase; cap-dependent transcription / viral RNA-dependent RNA polymerase; antiviral drug; nucleoside analogue; T705 (favipiravir); T1106; cap-dependent transcription; backtracking; / H7N9 / viral RNA-dependent RNA polymerase / viral RNA-dependent RNA polymerase; / nucleoside analogue |
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について











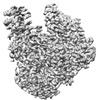

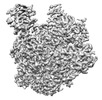

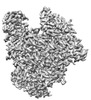



 influenza a virus (a/zhejiang/dtid-zju01/2013(h7n9)) (A型インフルエンザウイルス)
influenza a virus (a/zhejiang/dtid-zju01/2013(h7n9)) (A型インフルエンザウイルス) influenza a virus (A型インフルエンザウイルス)
influenza a virus (A型インフルエンザウイルス)