+検索条件
-Structure paper
| タイトル | Conformational motions and ligand-binding underlying gating and regulation in IPR channel. |
|---|---|
| ジャーナル・号・ページ | Nat Commun, Vol. 13, Issue 1, Page 6942, Year 2022 |
| 掲載日 | 2022年11月14日 |
 著者 著者 | Guizhen Fan / Mariah R Baker / Lara E Terry / Vikas Arige / Muyuan Chen / Alexander B Seryshev / Matthew L Baker / Steven J Ludtke / David I Yule / Irina I Serysheva /  |
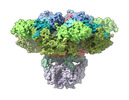
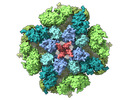
| PubMed 要旨 | Inositol-1,4,5-trisphosphate receptors (IPRs) are activated by IP and Ca and their gating is regulated by various intracellular messengers that finely tune the channel activity. Here, using single ...Inositol-1,4,5-trisphosphate receptors (IPRs) are activated by IP and Ca and their gating is regulated by various intracellular messengers that finely tune the channel activity. Here, using single particle cryo-EM analysis we determined 3D structures of the nanodisc-reconstituted IPR1 channel in two ligand-bound states. These structures provide unprecedented details governing binding of IP, Ca and ATP, revealing conformational changes that couple ligand-binding to channel opening. Using a deep-learning approach and 3D variability analysis we extracted molecular motions of the key protein domains from cryo-EM density data. We find that IP binding relies upon intrinsic flexibility of the ARM2 domain in the tetrameric channel. Our results highlight a key role of dynamic side chains in regulating gating behavior of IPR channels. This work represents a stepping-stone to developing mechanistic understanding of conformational pathways underlying ligand-binding, activation and regulation of the channel. |
 リンク リンク |  Nat Commun / Nat Commun /  PubMed:36376291 / PubMed:36376291 /  PubMed Central PubMed Central |
| 手法 | EM (単粒子) |
| 解像度 | 3.26 - 3.5 Å |
| 構造データ | EMDB-27982, PDB-8eaq: EMDB-27983, PDB-8ear: |
| 化合物 |  ChemComp-ZN:  ChemComp-CA:  ChemComp-PLX:  ChemComp-ATP: 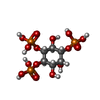 ChemComp-I3P: |
| 由来 |
|
 キーワード キーワード | MEMBRANE PROTEIN / Calcium channel / lipid nanodisc |
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について