+検索条件
-Structure paper
| タイトル | Structural basis of lipopolysaccharide maturation by the O-antigen ligase. |
|---|---|
| ジャーナル・号・ページ | Nature, Vol. 604, Issue 7905, Page 371-376, Year 2022 |
| 掲載日 | 2022年4月6日 |
 著者 著者 | Khuram U Ashraf / Rie Nygaard / Owen N Vickery / Satchal K Erramilli / Carmen M Herrera / Thomas H McConville / Vasileios I Petrou / Sabrina I Giacometti / Meagan Belcher Dufrisne / Kamil Nosol / Allen P Zinkle / Chris L B Graham / Michael Loukeris / Brian Kloss / Karolina Skorupinska-Tudek / Ewa Swiezewska / David I Roper / Oliver B Clarke / Anne-Catrin Uhlemann / Anthony A Kossiakoff / M Stephen Trent / Phillip J Stansfeld / Filippo Mancia /    |
| PubMed 要旨 | The outer membrane of Gram-negative bacteria has an external leaflet that is largely composed of lipopolysaccharide, which provides a selective permeation barrier, particularly against antimicrobials. ...The outer membrane of Gram-negative bacteria has an external leaflet that is largely composed of lipopolysaccharide, which provides a selective permeation barrier, particularly against antimicrobials. The final and crucial step in the biosynthesis of lipopolysaccharide is the addition of a species-dependent O-antigen to the lipid A core oligosaccharide, which is catalysed by the O-antigen ligase WaaL. Here we present structures of WaaL from Cupriavidus metallidurans, both in the apo state and in complex with its lipid carrier undecaprenyl pyrophosphate, determined by single-particle cryo-electron microscopy. The structures reveal that WaaL comprises 12 transmembrane helices and a predominantly α-helical periplasmic region, which we show contains many of the conserved residues that are required for catalysis. We observe a conserved fold within the GT-C family of glycosyltransferases and hypothesize that they have a common mechanism for shuttling the undecaprenyl-based carrier to and from the active site. The structures, combined with genetic, biochemical, bioinformatics and molecular dynamics simulation experiments, offer molecular details on how the ligands come in apposition, and allows us to propose a mechanistic model for catalysis. Together, our work provides a structural basis for lipopolysaccharide maturation in a member of the GT-C superfamily of glycosyltransferases. |
 リンク リンク |  Nature / Nature /  PubMed:35388216 / PubMed:35388216 /  PubMed Central PubMed Central |
| 手法 | EM (単粒子) |
| 解像度 | 3.23 - 3.46 Å |
| 構造データ | EMDB-26054, PDB-7tpg: EMDB-26057, PDB-7tpj: |
| 化合物 | 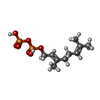 ChemComp-GPP: |
| 由来 |
|
 キーワード キーワード | MEMBRANE PROTEIN / Lipopolysaccharide / single-particle cryo-electron microscopy / molecular dynamics simulations / structural biology / undecaprenyl pyrophosphate / WaaL Ligase / lipid A / O-antigen / membrane proteins / transglycosylation / glycosyltransferase |
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について



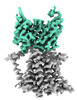

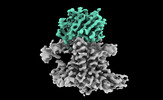

 cupriavidus metallidurans (バクテリア)
cupriavidus metallidurans (バクテリア) homo sapiens (ヒト)
homo sapiens (ヒト)