+検索条件
-Structure paper
| タイトル | Structural basis for dual specificity of yeast N-terminal amidase in the N-end rule pathway. |
|---|---|
| ジャーナル・号・ページ | Proc. Natl. Acad. Sci. U.S.A., Vol. 113, Page 12438-12443, Year 2016 |
| 掲載日 | 2016年5月24日 (構造データの登録日) |
 著者 著者 | Kim, M.K. / Oh, S.J. / Lee, B.G. / Song, H.K. |
 リンク リンク |  Proc. Natl. Acad. Sci. U.S.A. / Proc. Natl. Acad. Sci. U.S.A. /  PubMed:27791147 PubMed:27791147 |
| 手法 | X線回折 |
| 解像度 | 1.899 - 3.042 Å |
| 構造データ |  PDB-5b62:  PDB-5hyy:  PDB-5k5u:  PDB-5k5v:  PDB-5k60:  PDB-5k61:  PDB-5k62:  PDB-5k63:  PDB-5k66: |
| 化合物 |  ChemComp-HOH: 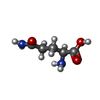 ChemComp-GLN: 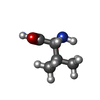 ChemComp-VAL: 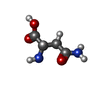 ChemComp-ASN:  ChemComp-GLY:  ChemComp-GLU: |
| 由来 |
|
 キーワード キーワード | HYDROLASE / N-end rule / Nitrilase superfamily / Nta1 |
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について




