+検索条件
-Structure paper
| タイトル | Mechanisms for activation and antagonism of an AMPA-sensitive glutamate receptor: crystal structures of the GluR2 ligand binding core. |
|---|---|
| ジャーナル・号・ページ | Neuron, Vol. 28, Page 165-181, Year 2000 |
| 掲載日 | 2000年9月12日 (構造データの登録日) |
 著者 著者 | Armstrong, N. / Gouaux, E. |
 リンク リンク |  Neuron / Neuron /  PubMed:11086992 PubMed:11086992 |
| 手法 | X線回折 |
| 解像度 | 1.6 - 2 Å |
| 構造データ |  PDB-1ftj:  PDB-1ftk:  PDB-1ftl:  PDB-1ftm:  PDB-1fto:  PDB-1fw0: |
| 化合物 |  ChemComp-ZN:  ChemComp-GLU:  ChemComp-HOH:  ChemComp-KAI: 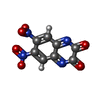 ChemComp-DNQ:  ChemComp-SO4: 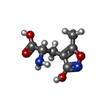 ChemComp-AMQ: |
| 由来 |
|
 キーワード キーワード | MEMBRANE PROTEIN / Ionotropic Glutamate Receptor Ligand Binding Core S1S2 Full Agonist Complex / GluR2 / S1S2 / ligand binding domain / kainate / partial agonist / ionotropic glutamate receptor / Glutamate receptor / ligand binding core / antagonist / DNQX / AMPA Receptor / ligand-binding core / agonist / Unligandeded / Apo |
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について




