+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Bombyx mori R2 retrotransposon initiating target-primed reverse transcription | |||||||||
 マップデータ マップデータ | Sharpened map of Bombyx mori R2 retrotransposon initiating target-primed reverse transcription | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | retrotransposon / LINE / reverse transcriptase / endonuclease / RNA BINDING PROTEIN / RNA BINDING PROTEIN-RNA-DNA complex | |||||||||
| 機能・相同性 | Zinc finger C2H2 type domain profile. / Zinc finger C2H2 type domain signature. / Zinc finger C2H2-type / Reverse transcriptase domain / Reverse transcriptase (RT) catalytic domain profile. / Reverse transcriptase (RNA-dependent DNA polymerase) / RNA-directed DNA polymerase activity / DNA/RNA polymerase superfamily / Reverse transcriptase-like protein 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  | |||||||||
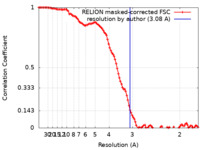
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.08 Å | |||||||||
 データ登録者 データ登録者 | Wilkinson ME / Zhang F | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Science / 年: 2023 ジャーナル: Science / 年: 2023タイトル: Structure of the R2 non-LTR retrotransposon initiating target-primed reverse transcription. 著者: Max E Wilkinson / Chris J Frangieh / Rhiannon K Macrae / Feng Zhang /  要旨: Non-long terminal repeat (non-LTR) retrotransposons, or long interspersed nuclear elements (LINEs), are an abundant class of eukaryotic transposons that insert into genomes by target-primed reverse ...Non-long terminal repeat (non-LTR) retrotransposons, or long interspersed nuclear elements (LINEs), are an abundant class of eukaryotic transposons that insert into genomes by target-primed reverse transcription (TPRT). During TPRT, a target DNA sequence is nicked and primes reverse transcription of the retrotransposon RNA. Here, we report the cryo-electron microscopy structure of the R2 non-LTR retrotransposon initiating TPRT at its ribosomal DNA target. The target DNA sequence is unwound at the insertion site and recognized by an upstream motif. An extension of the reverse transcriptase (RT) domain recognizes the retrotransposon RNA and guides the 3' end into the RT active site to template reverse transcription. We used Cas9 to retarget R2 in vitro to non-native sequences, suggesting future use as a reprogrammable RNA-based gene-insertion tool. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_40033.map.gz emd_40033.map.gz | 96.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-40033-v30.xml emd-40033-v30.xml emd-40033.xml emd-40033.xml | 23.1 KB 23.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_40033_fsc.xml emd_40033_fsc.xml | 10.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_40033.png emd_40033.png | 112.1 KB | ||
| マスクデータ |  emd_40033_msk_1.map emd_40033_msk_1.map | 103 MB |  マスクマップ マスクマップ | |
| その他 |  emd_40033_half_map_1.map.gz emd_40033_half_map_1.map.gz emd_40033_half_map_2.map.gz emd_40033_half_map_2.map.gz | 80.8 MB 80.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-40033 http://ftp.pdbj.org/pub/emdb/structures/EMD-40033 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-40033 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-40033 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_40033_validation.pdf.gz emd_40033_validation.pdf.gz | 1.1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_40033_full_validation.pdf.gz emd_40033_full_validation.pdf.gz | 1.1 MB | 表示 | |
| XML形式データ |  emd_40033_validation.xml.gz emd_40033_validation.xml.gz | 17.8 KB | 表示 | |
| CIF形式データ |  emd_40033_validation.cif.gz emd_40033_validation.cif.gz | 23.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-40033 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-40033 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-40033 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-40033 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8gh6MC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_40033.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_40033.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Sharpened map of Bombyx mori R2 retrotransposon initiating target-primed reverse transcription | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.884 Å | ||||||||||||||||||||||||||||||||||||
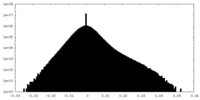
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_40033_msk_1.map emd_40033_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
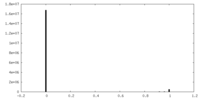
| 密度ヒストグラム |
-ハーフマップ: Half map one
| ファイル | emd_40033_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map one | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
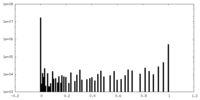
| 密度ヒストグラム |
-ハーフマップ: Half map two
| ファイル | emd_40033_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map two | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
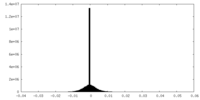
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Bombyx mori R2 retrotransposon initiating target-primed reverse t...
| 全体 | 名称: Bombyx mori R2 retrotransposon initiating target-primed reverse transcription |
|---|---|
| 要素 |
|
-超分子 #1: Bombyx mori R2 retrotransposon initiating target-primed reverse t...
| 超分子 | 名称: Bombyx mori R2 retrotransposon initiating target-primed reverse transcription タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#5 |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 230 KDa |
-分子 #1: Reverse transcriptase-like protein
| 分子 | 名称: Reverse transcriptase-like protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 123.358352 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MMASTALSLM GRCNPDGCTR GKHVTAAPMD GPRGPSSLAG TFGWGLAIPA GEPCGRVCSP ATVGFFPVAK KSNKENRPEA SGLPLESER TGDNPTVRGS AGADPVGQDA PGWTCQFCER TFSTNRGLGV HKRRAHPVET NTDAAPMMVK RRWHGEEIDL L ARTEARLL ...文字列: MMASTALSLM GRCNPDGCTR GKHVTAAPMD GPRGPSSLAG TFGWGLAIPA GEPCGRVCSP ATVGFFPVAK KSNKENRPEA SGLPLESER TGDNPTVRGS AGADPVGQDA PGWTCQFCER TFSTNRGLGV HKRRAHPVET NTDAAPMMVK RRWHGEEIDL L ARTEARLL AERGQCSGGD LFGALPGFGR TLEAIKGQRR REPYRALVQA HLARFGSQPG PSSGGCSAEP DFRRASGAEE AG EERCAED AAAYDPSAVG QMSPDAARVL SELLEGAGRR RACRAMRPKT AGRRNDLHDD RTASAHKTSR QKRRAEYARV QEL YKKCRS RAAAEVIDGA CGGVGHSLEE METYWRPILE RVSDAPGPTP EALHALGRAE WHGGNRDYTQ LWKPISVEEI KASR FDWRT SPGPDGIRSG QWRAVPVHLK AEMFNAWMAR GEIPEILRQC RTVFVPKVER PGGPGEYRPI SIASIPLRHF HSILA RRLL ACCPPDARQR GFICADGTLE NSAVLDAVLG DSRKKLRECH VAVLDFAKAF DTVSHEALVE LLRLRGMPEQ FCGYIA HLY DTASTTLAVN NEMSSPVKVG RGVRQGDPLS PILFNVVMDL ILASLPERVG YRLEMELVSA LAYADDLVLL AGSKVGM QE SISAVDCVGR QMGLRLNCRK SAVLSMIPDG HRKKHHYLTE RTFNIGGKPL RQVSCVERWR YLGVDFEASG CVTLEHSI S SALNNISRAP LKPQQRLEIL RAHLIPRFQH GFVLGNISDD RLRMLDVQIR KAVGQWLRLP ADVPKAYYHA AVQDGGLAI PSVRATIPDL IVRRFGGLDS SPWSVARAAA KSDKIRKKLR WAWKQLRRFS RVDSTTQRPS VRLFWREHLH ASVDGRELRE STRTPTSTK WIRERCAQIT GRDFVQFVHT HINALPSRIR GSRGRRGGGE SSLTCRAGCK VRETTAHILQ QCHRTHGGRI L RHNKIVSF VAKAMEENKW TVELEPRLRT SVGLRKPDII ASRDGVGVIV DVQVVSGQRS LDELHREKRN KYGNHGELVE LV AGRLGLP KAECVRATSC TISWRGVWSL TSYKELRSII GLREPTLQIV PILALRGSHM NWTRFNQMTS VMGGGVG UniProtKB: Reverse transcriptase-like protein |
-分子 #2: 28S DNA bottom strand, 3' side
| 分子 | 名称: 28S DNA bottom strand, 3' side / タイプ: dna / ID: 2 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 21.507758 KDa |
| 配列 | 文字列: (DT)(DT)(DA)(DG)(DA)(DT)(DG)(DA)(DC)(DG) (DA)(DG)(DG)(DC)(DA)(DT)(DT)(DT)(DG)(DG) (DC)(DT)(DA)(DC)(DC)(DT)(DT)(DA)(DA) (DG)(DA)(DG)(DA)(DG)(DT)(DC)(DA)(DT)(DA) (DG) (DT)(DT)(DA)(DC)(DT) ...文字列: (DT)(DT)(DA)(DG)(DA)(DT)(DG)(DA)(DC)(DG) (DA)(DG)(DG)(DC)(DA)(DT)(DT)(DT)(DG)(DG) (DC)(DT)(DA)(DC)(DC)(DT)(DT)(DA)(DA) (DG)(DA)(DG)(DA)(DG)(DT)(DC)(DA)(DT)(DA) (DG) (DT)(DT)(DA)(DC)(DT)(DC)(DC)(DC) (DG)(DC)(DC)(DG)(DT)(DT)(DT)(DA)(DC)(DC) (DC)(DG) (DC)(DG)(DC)(DT)(DT)(DC)(DA) (DC)(DA)(DG) |
-分子 #3: 28S DNA bottom strand, 5' side (priming strand)
| 分子 | 名称: 28S DNA bottom strand, 5' side (priming strand) / タイプ: dna / ID: 3 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 7.439812 KDa |
| 配列 | 文字列: (DT)(DT)(DA)(DG)(DA)(DT)(DG)(DA)(DC)(DG) (DA)(DG)(DG)(DC)(DA)(DT)(DT)(DT)(DG)(DG) (DC)(DT)(DA)(DT) |
-分子 #5: 28S DNA top strand
| 分子 | 名称: 28S DNA top strand / タイプ: dna / ID: 5 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 21.654875 KDa |
| 配列 | 文字列: (DC)(DT)(DG)(DT)(DG)(DA)(DA)(DG)(DC)(DG) (DC)(DG)(DG)(DG)(DT)(DA)(DA)(DA)(DC)(DG) (DG)(DC)(DG)(DG)(DG)(DA)(DG)(DT)(DA) (DA)(DC)(DT)(DA)(DT)(DG)(DA)(DC)(DT)(DC) (DT) (DC)(DT)(DT)(DA)(DA) ...文字列: (DC)(DT)(DG)(DT)(DG)(DA)(DA)(DG)(DC)(DG) (DC)(DG)(DG)(DG)(DT)(DA)(DA)(DA)(DC)(DG) (DG)(DC)(DG)(DG)(DG)(DA)(DG)(DT)(DA) (DA)(DC)(DT)(DA)(DT)(DG)(DA)(DC)(DT)(DC) (DT) (DC)(DT)(DT)(DA)(DA)(DG)(DG)(DT) (DA)(DG)(DC)(DC)(DA)(DA)(DA)(DT)(DG)(DC) (DC)(DT) (DC)(DG)(DT)(DC)(DA)(DT)(DC) (DT)(DA)(DA) |
-分子 #4: R2Bm 3'UTR RNA
| 分子 | 名称: R2Bm 3'UTR RNA / タイプ: rna / ID: 4 / コピー数: 1 |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 81.21393 KDa |
| 配列 | 文字列: GCCUUGCACA GUAGUCCAGC GGUAAGGGUG UAGAUCAGGC CCGUCUGUUU CUCCCCCGGA GCUCGCUCCC UUGGCUUCCC UUAUAUAUU UUAACAUCAG AAACAGACAU UAAACAUCUA CUGAUCCAAU UUCGCCGGCG UACGGCCACG AUCGGGAGGG U GGGAAUCU ...文字列: GCCUUGCACA GUAGUCCAGC GGUAAGGGUG UAGAUCAGGC CCGUCUGUUU CUCCCCCGGA GCUCGCUCCC UUGGCUUCCC UUAUAUAUU UUAACAUCAG AAACAGACAU UAAACAUCUA CUGAUCCAAU UUCGCCGGCG UACGGCCACG AUCGGGAGGG U GGGAAUCU CGGGGGUCUU CCGAUCCUAA UCCAUGAUGA UUACGACCUG AGUCACUAAA GACGAUGGCA UGAUGAUCCG GC GAUGAAA AUAGCC |
-分子 #6: ZINC ION
| 分子 | 名称: ZINC ION / タイプ: ligand / ID: 6 / コピー数: 2 / 式: ZN |
|---|---|
| 分子量 | 理論値: 65.409 Da |
-分子 #7: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 7 / コピー数: 1 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-分子 #8: THYMIDINE-5'-TRIPHOSPHATE
| 分子 | 名称: THYMIDINE-5'-TRIPHOSPHATE / タイプ: ligand / ID: 8 / コピー数: 1 / 式: TTP |
|---|---|
| 分子量 | 理論値: 482.168 Da |
| Chemical component information |  ChemComp-TTP: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.9 詳細: 20 mM HEPES-KOH pH 7.9, 500 mM potassium acetate, 5 mM magnesium acetate, 1 mM TCEP |
|---|---|
| グリッド | モデル: Quantifoil R2/2 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: CONTINUOUS / 支持フィルム - Film thickness: 2 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 12 sec. / 前処理 - 雰囲気: AIR |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 285 K / 装置: FEI VITROBOT MARK IV |
| 詳細 | A260 = 3 |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 撮影したグリッド数: 1 / 実像数: 16551 / 平均露光時間: 0.69 sec. / 平均電子線量: 42.2 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.5 µm / 倍率(公称値): 130000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | プロトコル: AB INITIO MODEL |
|---|---|
| 得られたモデル |  PDB-8gh6: |
 ムービー
ムービー コントローラー
コントローラー








 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)