[English] 日本語
 Yorodumi
Yorodumi- EMDB-26656: SARS-Cov2 S protein structure in complex with neutralizing monocl... -
+ Open data
Open data
- Basic information
Basic information
| Entry |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
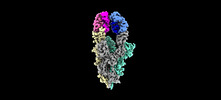
| Title | SARS-Cov2 S protein structure in complex with neutralizing monoclonal antibody 034_32 | |||||||||
 Map data Map data | Overall_map | |||||||||
 Sample Sample |
| |||||||||
 Keywords Keywords | SARS-Cov2 6P spike protein / immune complex / VIRAL PROTEIN | |||||||||
| Function / homology |  Function and homology information Function and homology informationsymbiont-mediated disruption of host tissue / Maturation of spike protein / Translation of Structural Proteins / Virion Assembly and Release / host cell surface / host extracellular space / viral translation / symbiont-mediated-mediated suppression of host tetherin activity / Induction of Cell-Cell Fusion / structural constituent of virion ...symbiont-mediated disruption of host tissue / Maturation of spike protein / Translation of Structural Proteins / Virion Assembly and Release / host cell surface / host extracellular space / viral translation / symbiont-mediated-mediated suppression of host tetherin activity / Induction of Cell-Cell Fusion / structural constituent of virion / membrane fusion / entry receptor-mediated virion attachment to host cell / Attachment and Entry / host cell endoplasmic reticulum-Golgi intermediate compartment membrane / positive regulation of viral entry into host cell / receptor-mediated virion attachment to host cell / host cell surface receptor binding / symbiont-mediated suppression of host innate immune response / receptor ligand activity / endocytosis involved in viral entry into host cell / fusion of virus membrane with host plasma membrane / fusion of virus membrane with host endosome membrane / viral envelope / symbiont entry into host cell / virion attachment to host cell / SARS-CoV-2 activates/modulates innate and adaptive immune responses / host cell plasma membrane / virion membrane / identical protein binding / membrane / plasma membrane Similarity search - Function | |||||||||
| Biological species |   Homo sapiens (human) / Homo sapiens (human) /  Severe acute respiratory syndrome coronavirus Severe acute respiratory syndrome coronavirus | |||||||||
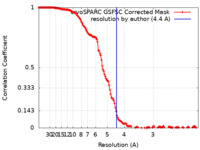
| Method | single particle reconstruction / cryo EM / Resolution: 4.4 Å | |||||||||
 Authors Authors | Patel A / Ortlund E | |||||||||
| Funding support |  United States, 1 items United States, 1 items
| |||||||||
 Citation Citation |  Journal: Structure / Year: 2023 Journal: Structure / Year: 2023Title: Molecular basis of SARS-CoV-2 Omicron variant evasion from shared neutralizing antibody response. Authors: Anamika Patel / Sanjeev Kumar / Lilin Lai / Chennareddy Chakravarthy / Rajesh Valanparambil / Elluri Seetharami Reddy / Kamalvishnu Gottimukkala / Prashant Bajpai / Dinesh Ravindra Raju / ...Authors: Anamika Patel / Sanjeev Kumar / Lilin Lai / Chennareddy Chakravarthy / Rajesh Valanparambil / Elluri Seetharami Reddy / Kamalvishnu Gottimukkala / Prashant Bajpai / Dinesh Ravindra Raju / Venkata Viswanadh Edara / Meredith E Davis-Gardner / Susanne Linderman / Kritika Dixit / Pragati Sharma / Grace Mantus / Narayanaiah Cheedarla / Hans P Verkerke / Filipp Frank / Andrew S Neish / John D Roback / Carl W Davis / Jens Wrammert / Rafi Ahmed / Mehul S Suthar / Amit Sharma / Kaja Murali-Krishna / Anmol Chandele / Eric A Ortlund /   Abstract: Understanding the molecular features of neutralizing epitopes is important for developing vaccines/therapeutics against emerging SARS-CoV-2 variants. We describe three monoclonal antibodies (mAbs) ...Understanding the molecular features of neutralizing epitopes is important for developing vaccines/therapeutics against emerging SARS-CoV-2 variants. We describe three monoclonal antibodies (mAbs) generated from COVID-19 recovered individuals during the first wave of the pandemic in India. These mAbs had publicly shared near germline gene usage and potently neutralized Alpha and Delta, poorly neutralized Beta, and failed to neutralize Omicron BA.1 SARS-CoV-2 variants. Structural analysis of these mAbs in complex with trimeric spike protein showed that all three mAbs bivalently bind spike with two mAbs targeting class 1 and one targeting a class 4 receptor binding domain epitope. The immunogenetic makeup, structure, and function of these mAbs revealed specific molecular interactions associated with the potent multi-variant binding/neutralization efficacy. This knowledge shows how mutational combinations can affect the binding or neutralization of an antibody, which in turn relates to the efficacy of immune responses to emerging SARS-CoV-2 escape variants. | |||||||||
| History |
|
- Structure visualization
Structure visualization
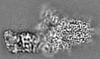
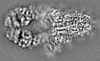
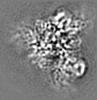
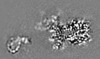
| Supplemental images |
|---|
- Downloads & links
Downloads & links
-EMDB archive
| Map data |  emd_26656.map.gz emd_26656.map.gz | 258.8 MB |  EMDB map data format EMDB map data format | |
|---|---|---|---|---|
| Header (meta data) |  emd-26656-v30.xml emd-26656-v30.xml emd-26656.xml emd-26656.xml | 30.4 KB 30.4 KB | Display Display |  EMDB header EMDB header |
| FSC (resolution estimation) |  emd_26656_fsc.xml emd_26656_fsc.xml | 15.7 KB | Display |  FSC data file FSC data file |
| Images |  emd_26656.png emd_26656.png | 20.1 KB | ||
| Masks |  emd_26656_msk_1.map emd_26656_msk_1.map | 274.6 MB |  Mask map Mask map | |
| Filedesc metadata |  emd-26656.cif.gz emd-26656.cif.gz | 8 KB | ||
| Others |  emd_26656_additional_1.map.gz emd_26656_additional_1.map.gz emd_26656_additional_2.map.gz emd_26656_additional_2.map.gz emd_26656_additional_3.map.gz emd_26656_additional_3.map.gz emd_26656_half_map_1.map.gz emd_26656_half_map_1.map.gz emd_26656_half_map_2.map.gz emd_26656_half_map_2.map.gz | 255.3 MB 17.3 MB 255.7 MB 254.5 MB 254.5 MB | ||
| Archive directory |  http://ftp.pdbj.org/pub/emdb/structures/EMD-26656 http://ftp.pdbj.org/pub/emdb/structures/EMD-26656 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-26656 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-26656 | HTTPS FTP |
-Validation report
| Summary document |  emd_26656_validation.pdf.gz emd_26656_validation.pdf.gz | 950.4 KB | Display |  EMDB validaton report EMDB validaton report |
|---|---|---|---|---|
| Full document |  emd_26656_full_validation.pdf.gz emd_26656_full_validation.pdf.gz | 950 KB | Display | |
| Data in XML |  emd_26656_validation.xml.gz emd_26656_validation.xml.gz | 22.9 KB | Display | |
| Data in CIF |  emd_26656_validation.cif.gz emd_26656_validation.cif.gz | 29.7 KB | Display | |
| Arichive directory |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26656 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26656 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26656 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26656 | HTTPS FTP |
-Related structure data
| Related structure data |  7uowMC  7u0qC  7u0xC M: atomic model generated by this map C: citing same article ( |
|---|---|
| Similar structure data | Similarity search - Function & homology  F&H Search F&H Search |
- Links
Links
| EMDB pages |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| Related items in Molecule of the Month |
- Map
Map
| File |  Download / File: emd_26656.map.gz / Format: CCP4 / Size: 274.6 MB / Type: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) Download / File: emd_26656.map.gz / Format: CCP4 / Size: 274.6 MB / Type: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Annotation | Overall_map | ||||||||||||||||||||||||||||||||||||
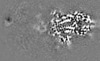
| Projections & slices | Image control
Images are generated by Spider. | ||||||||||||||||||||||||||||||||||||
| Voxel size | X=Y=Z: 1.0691 Å | ||||||||||||||||||||||||||||||||||||
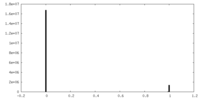
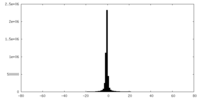
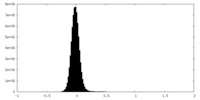
| Density |
| ||||||||||||||||||||||||||||||||||||
| Symmetry | Space group: 1 | ||||||||||||||||||||||||||||||||||||
| Details | EMDB XML:
|
-Supplemental data
-Mask #1
| File |  emd_26656_msk_1.map emd_26656_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
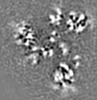
| Projections & Slices |
| ||||||||||||
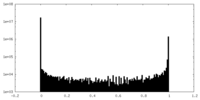
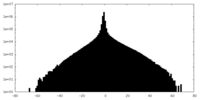
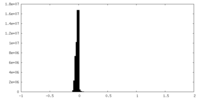
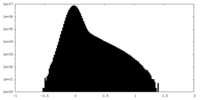
| Density Histograms |
-Additional map: Local refine RBD A
| File | emd_26656_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Annotation | Local_refine_RBD_A | ||||||||||||
| Projections & Slices |
| ||||||||||||
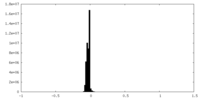
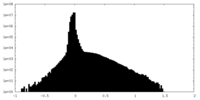
| Density Histograms |
-Additional map: Composit focused map
| File | emd_26656_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Annotation | Composit_focused_map | ||||||||||||
| Projections & Slices |
| ||||||||||||
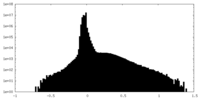
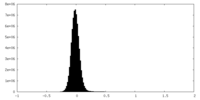
| Density Histograms |
-Additional map: Local refine RBD C
| File | emd_26656_additional_3.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Annotation | Local_refine_RBD_C | ||||||||||||
| Projections & Slices |
| ||||||||||||
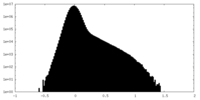
| Density Histograms |
-Half map: Half map A
| File | emd_26656_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Annotation | Half_map_A | ||||||||||||
| Projections & Slices |
| ||||||||||||
| Density Histograms |
-Half map: Half map B
| File | emd_26656_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Annotation | Half_map_B | ||||||||||||
| Projections & Slices |
| ||||||||||||
| Density Histograms |
- Sample components
Sample components
-Entire : SARS-Cov2 Spike trimer in complex with monoclonal antibody
| Entire | Name: SARS-Cov2 Spike trimer in complex with monoclonal antibody |
|---|---|
| Components |
|
-Supramolecule #1: SARS-Cov2 Spike trimer in complex with monoclonal antibody
| Supramolecule | Name: SARS-Cov2 Spike trimer in complex with monoclonal antibody type: complex / ID: 1 / Parent: 0 / Macromolecule list: #1-#3 |
|---|
-Supramolecule #2: Spike glycoprotein
| Supramolecule | Name: Spike glycoprotein / type: complex / ID: 2 / Parent: 1 / Macromolecule list: #1 |
|---|---|
| Source (natural) | Organism:  |
-Supramolecule #3: mAb 034-32 Heavy chain, mAb 034-32 Light chain
| Supramolecule | Name: mAb 034-32 Heavy chain, mAb 034-32 Light chain / type: complex / ID: 3 / Parent: 1 / Macromolecule list: #2-#3 |
|---|---|
| Source (natural) | Organism:  Homo sapiens (human) Homo sapiens (human) |
-Macromolecule #1: Spike glycoprotein
| Macromolecule | Name: Spike glycoprotein / type: protein_or_peptide / ID: 1 / Number of copies: 3 / Enantiomer: LEVO |
|---|---|
| Source (natural) | Organism:  Severe acute respiratory syndrome coronavirus Severe acute respiratory syndrome coronavirus |
| Molecular weight | Theoretical: 133.781312 KDa |
| Recombinant expression | Organism:  Homo sapiens (human) Homo sapiens (human) |
| Sequence | String: MFVFLVLLPL VSSQCVNLTT RTQLPPAYTN SFTRGVYYPD KVFRSSVLHS TQDLFLPFFS NVTWFHAIHV SGTNGTKRFD NPVLPFNDG VYFASTEKSN IIRGWIFGTT LDSKTQSLLI VNNATNVVIK VCEFQFCNDP FLGVYYHKNN KSWMESEFRV Y SSANNCTF ...String: MFVFLVLLPL VSSQCVNLTT RTQLPPAYTN SFTRGVYYPD KVFRSSVLHS TQDLFLPFFS NVTWFHAIHV SGTNGTKRFD NPVLPFNDG VYFASTEKSN IIRGWIFGTT LDSKTQSLLI VNNATNVVIK VCEFQFCNDP FLGVYYHKNN KSWMESEFRV Y SSANNCTF EYVSQPFLMD LEGKQGNFKN LREFVFKNID GYFKIYSKHT PINLVRDLPQ GFSALEPLVD LPIGINITRF QT LLALHRS YLTPGDSSSG WTAGAAAYYV GYLQPRTFLL KYNENGTITD AVDCALDPLS ETKCTLKSFT VEKGIYQTSN FRV QPTESI VRFPNITNLC PFGEVFNATR FASVYAWNRK RISNCVADYS VLYNSASFST FKCYGVSPTK LNDLCFTNVY ADSF VIRGD EVRQIAPGQT GKIADYNYKL PDDFTGCVIA WNSNNLDSKV GGNYNYLYRL FRKSNLKPFE RDISTEIYQA GSTPC NGVE GFNCYFPLQS YGFQPTNGVG YQPYRVVVLS FELLHAPATV CGPKKSTNLV KNKCVNFNFN GLTGTGVLTE SNKKFL PFQ QFGRDIADTT DAVRDPQTLE ILDITPCSFG GVSVITPGTN TSNQVAVLYQ DVNCTEVPVA IHADQLTPTW RVYSTGS NV FQTRAGCLIG AEHVNNSYEC DIPIGAGICA SYQTQTNSPG SASSVASQSI IAYTMSLGAE NSVAYSNNSI AIPTNFTI S VTTEILPVSM TKTSVDCTMY ICGDSTECSN LLLQYGSFCT QLNRALTGIA VEQDKNTQEV FAQVKQIYKT PPIKDFGGF NFSQILPDPS KPSKRSPIED LLFNKVTLAD AGFIKQYGDC LGDIAARDLI CAQKFNGLTV LPPLLTDEMI AQYTSALLAG TITSGWTFG AGPALQIPFP MQMAYRFNGI GVTQNVLYEN QKLIANQFNS AIGKIQDSLS STPSALGKLQ DVVNQNAQAL N TLVKQLSS NFGAISSVLN DILSRLDPPE AEVQIDRLIT GRLQSLQTYV TQQLIRAAEI RASANLAATK MSECVLGQSK RV DFCGKGY HLMSFPQSAP HGVVFLHVTY VPAQEKNFTT APAICHDGKA HFPREGVFVS NGTHWFVTQR NFYEPQIITT DNT FVSGNC DVVIGIVNNT VYDPLQPELD SFKEELDKYF KNHTSPDVDL GDISGINASV VNIQKEIDRL NEVAKNLNES LIDL QELGK YEQ UniProtKB: Spike glycoprotein |
-Macromolecule #2: Monoclonal antibody 034_32 heavy chain
| Macromolecule | Name: Monoclonal antibody 034_32 heavy chain / type: protein_or_peptide / ID: 2 / Number of copies: 2 / Enantiomer: LEVO |
|---|---|
| Source (natural) | Organism:  Homo sapiens (human) Homo sapiens (human) |
| Molecular weight | Theoretical: 48.869992 KDa |
| Recombinant expression | Organism:  Homo sapiens (human) Homo sapiens (human) |
| Sequence | String: EVQLVESGGG LIQPGGSLRL SCAASGITVS SNYMSWVRQA PGKGLEWVSV IYAGGSTFYA DSVKGRFTIS RDNSKNTLYL QMSSLRVED TAVYYCARDL DYYGMDVWGR GTTVTVSSAS TKGPSVFPLA PSSKSTSGGT AALGCLVKDY FPEPVTVSWN S GALTSGVH ...String: EVQLVESGGG LIQPGGSLRL SCAASGITVS SNYMSWVRQA PGKGLEWVSV IYAGGSTFYA DSVKGRFTIS RDNSKNTLYL QMSSLRVED TAVYYCARDL DYYGMDVWGR GTTVTVSSAS TKGPSVFPLA PSSKSTSGGT AALGCLVKDY FPEPVTVSWN S GALTSGVH TFPAVLQSSG LYSLSSVVTV PSSSLGTQTY ICNVNHKPSN TKVDKRVEPK SCDKTHTCPP CPAPELLGGP SV FLFPPKP KDTLMISRTP EVTCVVVDVS HEDPEVKFNW YVDGVEVHNA KTKPREEQYN STYRVVSVLT VLHQDWLNGK EYK CKVSNK ALPAPIEKTI SKAKGQPREP QVYTLPPSRE EMTKNQVSLT CLVKGFYPSD IAVEWESNGQ PENNYKTTPP VLDS DGSFF LYSKLTVDKS RWQQGNVFSC SVMHEALHNH YTQKSLSLSP GK |
-Macromolecule #3: Monoclonal antibody 034_32 light chain
| Macromolecule | Name: Monoclonal antibody 034_32 light chain / type: protein_or_peptide / ID: 3 / Number of copies: 2 / Enantiomer: LEVO |
|---|---|
| Source (natural) | Organism:  Homo sapiens (human) Homo sapiens (human) |
| Molecular weight | Theoretical: 23.225838 KDa |
| Recombinant expression | Organism:  Homo sapiens (human) Homo sapiens (human) |
| Sequence | String: AIQLTQSPSS LSASVGDRVT ITCRASQGIS TYLAWYQQKP GKAPKLLIYG ASTLQSGVPS RFSGSGSGTD FTLTISSLQP EDFAAYYCQ QVNSYPPITF GPGTKVDIKR TVAAPSVFIF PPSDEQLKSG TASVVCLLNN FYPREAKVQW KVDNALQSGN S QESVTEQD ...String: AIQLTQSPSS LSASVGDRVT ITCRASQGIS TYLAWYQQKP GKAPKLLIYG ASTLQSGVPS RFSGSGSGTD FTLTISSLQP EDFAAYYCQ QVNSYPPITF GPGTKVDIKR TVAAPSVFIF PPSDEQLKSG TASVVCLLNN FYPREAKVQW KVDNALQSGN S QESVTEQD SKDSTYSLSS TLTLSKADYE KHKVYACEVT HQGLSSPVTK SFNRGEC |
-Macromolecule #5: 2-acetamido-2-deoxy-beta-D-glucopyranose
| Macromolecule | Name: 2-acetamido-2-deoxy-beta-D-glucopyranose / type: ligand / ID: 5 / Number of copies: 23 / Formula: NAG |
|---|---|
| Molecular weight | Theoretical: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-Experimental details
-Structure determination
| Method | cryo EM |
|---|---|
 Processing Processing | single particle reconstruction |
| Aggregation state | particle |
- Sample preparation
Sample preparation
| Concentration | 0.7 mg/mL | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Buffer | pH: 7.4 Component:
| |||||||||||||||
| Grid | Model: C-flat-1.2/1.3 / Material: COPPER / Mesh: 400 / Support film - Material: CARBON / Support film - topology: HOLEY / Pretreatment - Type: GLOW DISCHARGE / Pretreatment - Time: 30 sec. / Pretreatment - Atmosphere: AIR | |||||||||||||||
| Vitrification | Cryogen name: ETHANE / Chamber humidity: 100 % / Chamber temperature: 297 K / Instrument: FEI VITROBOT MARK IV / Details: Wait time 20 seconds and blot time 3 seconds. |
- Electron microscopy
Electron microscopy
| Microscope | FEI TALOS ARCTICA |
|---|---|
| Specialist optics | Energy filter - Slit width: 30 eV |
| Image recording | Film or detector model: GATAN K3 (6k x 4k) / Number grids imaged: 1 / Number real images: 4186 / Average electron dose: 63.81 e/Å2 |
| Electron beam | Acceleration voltage: 300 kV / Electron source:  FIELD EMISSION GUN FIELD EMISSION GUN |
| Electron optics | Illumination mode: FLOOD BEAM / Imaging mode: BRIGHT FIELD / Cs: 2.7 mm / Nominal defocus max: 2.6 µm / Nominal defocus min: 0.6 µm / Nominal magnification: 81000 |
| Sample stage | Cooling holder cryogen: NITROGEN |
| Experimental equipment |  Model: Talos Arctica / Image courtesy: FEI Company |
+ Image processing
Image processing
-Atomic model buiding 1
| Refinement | Space: REAL / Protocol: RIGID BODY FIT / Overall B value: 102 / Target criteria: Correlation coefficient |
|---|---|
| Output model |  PDB-7uow: |
 Movie
Movie Controller
Controller








 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)