+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | CHAPSO treated partial catalytic component (comprising only AnfD & AnfK, lacking AnfG and FeFeco) of iron nitrogenase from Rhodobacter capsulatus | |||||||||
 マップデータ マップデータ | Core complex of Fe-nitrogenase comprising only AnfD and AnfK. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | nitrogen fixation / Fe nitrogenase / OXIDOREDUCTASE | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報nitrogenase / : / nitrogenase activity / nitrogen fixation / iron-sulfur cluster binding / ATP binding / metal ion binding 類似検索 - 分子機能 | |||||||||
| 生物種 |  Rhodobacter capsulatus SB 1003 (バクテリア) Rhodobacter capsulatus SB 1003 (バクテリア) | |||||||||
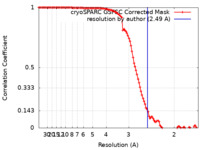
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.49 Å | |||||||||
 データ登録者 データ登録者 | Schmidt FV / Schulz L / Zarzycki J / Prinz S / Erb TJ / Rebelein JG | |||||||||
| 資金援助 |  ドイツ, 2件 ドイツ, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2024 ジャーナル: Nat Struct Mol Biol / 年: 2024タイトル: Structural insights into the iron nitrogenase complex. 著者: Frederik V Schmidt / Luca Schulz / Jan Zarzycki / Simone Prinz / Niels N Oehlmann / Tobias J Erb / Johannes G Rebelein /  要旨: Nitrogenases are best known for catalyzing the reduction of dinitrogen to ammonia at a complex metallic cofactor. Recently, nitrogenases were shown to reduce carbon dioxide (CO) and carbon monoxide ...Nitrogenases are best known for catalyzing the reduction of dinitrogen to ammonia at a complex metallic cofactor. Recently, nitrogenases were shown to reduce carbon dioxide (CO) and carbon monoxide to hydrocarbons, offering a pathway to recycle carbon waste into hydrocarbon products. Among the three nitrogenase isozymes, the iron nitrogenase has the highest wild-type activity for the reduction of CO, but the molecular architecture facilitating these activities has remained unknown. Here, we report a 2.35-Å cryogenic electron microscopy structure of the ADP·AlF-stabilized iron nitrogenase complex from Rhodobacter capsulatus, revealing an [FeSC-(R)-homocitrate] cluster in the active site. The enzyme complex suggests that the iron nitrogenase G subunit is involved in cluster stabilization and substrate channeling and confers specificity between nitrogenase reductase and catalytic component proteins. Moreover, the structure highlights a different interface between the two catalytic halves of the iron and the molybdenum nitrogenase, potentially influencing the intrasubunit 'communication' and thus the nitrogenase mechanism. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_17583.map.gz emd_17583.map.gz | 122.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-17583-v30.xml emd-17583-v30.xml emd-17583.xml emd-17583.xml | 18.5 KB 18.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_17583_fsc.xml emd_17583_fsc.xml | 10.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_17583.png emd_17583.png | 59.8 KB | ||
| Filedesc metadata |  emd-17583.cif.gz emd-17583.cif.gz | 6.3 KB | ||
| その他 |  emd_17583_half_map_1.map.gz emd_17583_half_map_1.map.gz emd_17583_half_map_2.map.gz emd_17583_half_map_2.map.gz | 120.1 MB 120.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-17583 http://ftp.pdbj.org/pub/emdb/structures/EMD-17583 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-17583 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-17583 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_17583_validation.pdf.gz emd_17583_validation.pdf.gz | 891.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_17583_full_validation.pdf.gz emd_17583_full_validation.pdf.gz | 891 KB | 表示 | |
| XML形式データ |  emd_17583_validation.xml.gz emd_17583_validation.xml.gz | 19.3 KB | 表示 | |
| CIF形式データ |  emd_17583_validation.cif.gz emd_17583_validation.cif.gz | 24.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17583 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17583 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17583 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17583 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8pbbMC  8oieC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_17583.map.gz / 形式: CCP4 / 大きさ: 129.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_17583.map.gz / 形式: CCP4 / 大きさ: 129.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Core complex of Fe-nitrogenase comprising only AnfD and AnfK. | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.837 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: half-map A
| ファイル | emd_17583_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half-map A | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half-map B
| ファイル | emd_17583_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half-map B | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : CHAPSO treated partial catalytic component (comprising only AnfD ...
| 全体 | 名称: CHAPSO treated partial catalytic component (comprising only AnfD & AnfK, lacking AnfG and FeFeco) of iron nitrogenase from Rhodobacter capsulatus |
|---|---|
| 要素 |
|
-超分子 #1: CHAPSO treated partial catalytic component (comprising only AnfD ...
| 超分子 | 名称: CHAPSO treated partial catalytic component (comprising only AnfD & AnfK, lacking AnfG and FeFeco) of iron nitrogenase from Rhodobacter capsulatus タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 |
|---|---|
| 由来(天然) | 生物種:  Rhodobacter capsulatus SB 1003 (バクテリア) Rhodobacter capsulatus SB 1003 (バクテリア) |
-分子 #1: Nitrogenase protein alpha chain
| 分子 | 名称: Nitrogenase protein alpha chain / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Rhodobacter capsulatus SB 1003 (バクテリア) Rhodobacter capsulatus SB 1003 (バクテリア) |
| 分子量 | 理論値: 60.221117 KDa |
| 組換発現 | 生物種:  Rhodobacter capsulatus SB 1003 (バクテリア) Rhodobacter capsulatus SB 1003 (バクテリア) |
| 配列 | 文字列: MPYHEFEVSK CIPERREHAV MKAAGEDLTS CLPKGYLNTI PGTISERGCA YCGAKHVIGT PMKDVIHISH GPNGCTYDTW QTKRYISDN DNFQLKYTFA TDVKEKHVVF GAEGLLKKSM HEAFDAFPNI KRMTVYQTCT TALIGDDVDA IAKEVMEERG D VDVFVCNS ...文字列: MPYHEFEVSK CIPERREHAV MKAAGEDLTS CLPKGYLNTI PGTISERGCA YCGAKHVIGT PMKDVIHISH GPNGCTYDTW QTKRYISDN DNFQLKYTFA TDVKEKHVVF GAEGLLKKSM HEAFDAFPNI KRMTVYQTCT TALIGDDVDA IAKEVMEERG D VDVFVCNS PGFAGPSQSG GHHKINIAWL NQKVGTVEPD YLGEHVINYV GEYNIQGDQE VMIDYFNRMG IQVLSTFTGN GS YDSLRMM HRAHLNVLEC ARSAEYICDE LRARYGIPRL DIDGFGFEPL ANSLRKVALF FGIEDKAEAI IAEEYAKWKP QLD WYKERL KGKKVCLWPG GSKLWHWAHA IEEEMGLKVV SVYTKFGHQG DMEKGVSRCG EGALAIDDPN ELESVEAIEM LKPD IIFTG KRPGEFVKKH GVPYLNAHAY HNGPYKGFEG WVRFARDIYN AIYSPMRQLA ALDISAPDAA ITSGFRTAKM NADLT VSDE VKFSEVLHEY TGKYDSIAEI RARNQAYAAE QKALRDAVQP AAEWSHPQFE K UniProtKB: Nitrogenase protein alpha chain |
-分子 #2: Nitrogenase iron-iron protein, beta subunit
| 分子 | 名称: Nitrogenase iron-iron protein, beta subunit / タイプ: protein_or_peptide / ID: 2 / コピー数: 2 / 光学異性体: LEVO / EC番号: nitrogenase |
|---|---|
| 由来(天然) | 生物種:  Rhodobacter capsulatus SB 1003 (バクテリア) Rhodobacter capsulatus SB 1003 (バクテリア) |
| 分子量 | 理論値: 50.776398 KDa |
| 組換発現 | 生物種:  Rhodobacter capsulatus SB 1003 (バクテリア) Rhodobacter capsulatus SB 1003 (バクテリア) |
| 配列 | 文字列: MTCQVTQKAR EGTINPIFTC QPAGAQFASI GIKDCIGIVH GGQGCVMFVR LLISQHMKES FEIASSSVHE DGAVFGALDR VETAVEVLL TRYPDVKVVP IITTCSTEII GDDVDGLLSK LEDELLPTKF PGREVHLLTV HCPSFVGSMI TGYDKAVHDF V KKFATKDE ...文字列: MTCQVTQKAR EGTINPIFTC QPAGAQFASI GIKDCIGIVH GGQGCVMFVR LLISQHMKES FEIASSSVHE DGAVFGALDR VETAVEVLL TRYPDVKVVP IITTCSTEII GDDVDGLLSK LEDELLPTKF PGREVHLLTV HCPSFVGSMI TGYDKAVHDF V KKFATKDE PSDKINLITG WVNPGDVKEL KHLLEVMEVK ANVLFEVESF DSPLMPDLEH HSHGSTTIED LRDTANAKGT IA LNRYEGM KAADYLKKKF KVPAVIGPTP VGIRNTDAFL KAVSEMTGQP IPAQLVKERG LALDAIADIG HMFLADKRVA IYA NPDLAI GLTEFCLDLE MKPKLLLLGD DNSGYVKDPR VLALQENAPD LEIVTNADFW DLESRIQQGL ELDLILGHSK GRFI SIDYK VPMVRVGFPT YDRAGMYRHP VLGYGGAMFL AETMANTLFA DMEAKKNKEW ILNVW UniProtKB: Nitrogenase iron-iron protein, beta subunit |
-分子 #3: FE(8)-S(7) CLUSTER
| 分子 | 名称: FE(8)-S(7) CLUSTER / タイプ: ligand / ID: 3 / コピー数: 2 / 式: CLF |
|---|---|
| 分子量 | 理論値: 671.215 Da |
| Chemical component information | 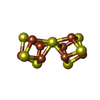 ChemComp-CLF: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.75 mg/mL |
|---|---|
| 緩衝液 | pH: 7.8 詳細: 37.5 mM TRIS (pH = 7.8) 150 mM NaCl 3.75 mM sodium dithionite 0.4 % (m/V) CHAPSO |
| グリッド | モデル: Quantifoil R2/1 / 材質: COPPER / メッシュ: 300 / 前処理 - タイプ: GLOW DISCHARGE |
| 凍結 | 凍結剤: ETHANE-PROPANE / チャンバー内湿度: 90 % / チャンバー内温度: 281 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.4 µm 最小 デフォーカス(公称値): 1.4000000000000001 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー





 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)