+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-8075 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of the cellulose synthase complex of Gluconacetobacter hansenii at 23.4 Angstrom resolution | |||||||||
 マップデータ マップデータ | None | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |  Gluconacetobacter hansenii ATCC 23769 (バクテリア) / Gluconacetobacter hansenii ATCC 23769 (バクテリア) /  Komagataeibacter hansenii (バクテリア) Komagataeibacter hansenii (バクテリア) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  ネガティブ染色法 / 解像度: 23.4 Å ネガティブ染色法 / 解像度: 23.4 Å | |||||||||
 データ登録者 データ登録者 | Nixon BT / Du J | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
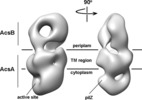
 引用 引用 |  ジャーナル: PLoS One / 年: 2016 ジャーナル: PLoS One / 年: 2016タイトル: Structure of the Cellulose Synthase Complex of Gluconacetobacter hansenii at 23.4 Å Resolution. 著者: Juan Du / Venkata Vepachedu / Sung Hyun Cho / Manish Kumar / B Tracy Nixon /  要旨: Bacterial crystalline cellulose is used in biomedical and industrial applications, but the molecular mechanisms of synthesis are unclear. Unlike most bacteria, which make non-crystalline cellulose, ...Bacterial crystalline cellulose is used in biomedical and industrial applications, but the molecular mechanisms of synthesis are unclear. Unlike most bacteria, which make non-crystalline cellulose, Gluconacetobacter hansenii extrudes profuse amounts of crystalline cellulose. Its cellulose synthase (AcsA) exists as a complex with accessory protein AcsB, forming a 'terminal complex' (TC) that has been visualized by freeze-fracture TEM at the base of ribbons of crystalline cellulose. The catalytic AcsAB complex is embedded in the cytoplasmic membrane. The C-terminal portion of AcsC is predicted to form a translocation channel in the outer membrane, with the rest of AcsC possibly interacting with AcsD in the periplasm. It is thus believed that synthesis from an organized array of TCs coordinated with extrusion by AcsC and AcsD enable this bacterium to make crystalline cellulose. The only structural data that exist for this system are the above mentioned freeze-fracture TEM images, fluorescence microscopy images revealing that TCs align in a row, a crystal structure of AcsD bound to cellopentaose, and a crystal structure of PilZ domain of AcsA. Here we advance our understanding of the structural basis for crystalline cellulose production by bacterial cellulose synthase by determining a negative stain structure resolved to 23.4 Å for highly purified AcsAB complex that catalyzed incorporation of UDP-glucose into β-1,4-glucan chains, and responded to the presence of allosteric activator cyclic diguanylate. Although the AcsAB complex was functional in vitro, the synthesized cellulose was not visible in TEM. The negative stain structure revealed that AcsAB is very similar to that of the BcsAB synthase of Rhodobacter sphaeroides, a non-crystalline cellulose producing bacterium. The results indicate that the crystalline cellulose producing and non-crystalline cellulose producing bacteria share conserved catalytic and membrane translocation components, and support the hypothesis that it is the extrusion mechanism and order in linearly arrayed TCs that enables production of crystalline cellulose. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_8075.map.gz emd_8075.map.gz | 31.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-8075-v30.xml emd-8075-v30.xml emd-8075.xml emd-8075.xml | 10.3 KB 10.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_8075.png emd_8075.png | 67.8 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-8075 http://ftp.pdbj.org/pub/emdb/structures/EMD-8075 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8075 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8075 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_8075.map.gz / 形式: CCP4 / 大きさ: 34.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_8075.map.gz / 形式: CCP4 / 大きさ: 34.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | None | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.45 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : membrane protein AcsAB
| 全体 | 名称: membrane protein AcsAB 生体膜 生体膜 |
|---|---|
| 要素 |
|
-超分子 #1: membrane protein AcsAB
| 超分子 | 名称: membrane protein AcsAB / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Gluconacetobacter hansenii ATCC 23769 (バクテリア) Gluconacetobacter hansenii ATCC 23769 (バクテリア) |
| 組換発現 | 生物種:  Komagataeibacter hansenii ATCC 23769 (バクテリア) Komagataeibacter hansenii ATCC 23769 (バクテリア)組換プラスミド: pUCD2CDHisAcsAB |
| 分子量 | 理論値: 170 KDa |
-分子 #1: AcsAB Cellulose Synthase of Gluconacetobacter hansenii
| 分子 | 名称: AcsAB Cellulose Synthase of Gluconacetobacter hansenii タイプ: protein_or_peptide / ID: 1 詳細: Heterodimer of AcsA and AcsB; membrane protein expressed as a fusion protein and then proteolytically processed. 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Komagataeibacter hansenii (バクテリア) Komagataeibacter hansenii (バクテリア) |
| 組換発現 | 生物種:  Komagataeibacter hansenii (バクテリア) Komagataeibacter hansenii (バクテリア) |
| 配列 | 文字列: MPEVRSSTQS ESGMSQWMGK ILSIRGAGLI IGVFGLCALI AATSVTLPPE QQLIVAFVCV VIFFIVGHKP SRRSQIFLEV LSGLVSLRYL TWRLTETLSF DTWLQGLLGT MLLVAELYAL MMLFLSYFQT IAPLHRAPLP LPPNPDEWPT VDIFVPTYNE ELSIVRLTVL ...文字列: MPEVRSSTQS ESGMSQWMGK ILSIRGAGLI IGVFGLCALI AATSVTLPPE QQLIVAFVCV VIFFIVGHKP SRRSQIFLEV LSGLVSLRYL TWRLTETLSF DTWLQGLLGT MLLVAELYAL MMLFLSYFQT IAPLHRAPLP LPPNPDEWPT VDIFVPTYNE ELSIVRLTVL GSLGIDWPPE KVRVHILDDG RRPEFAAFAA ECGANYIARP TNEHAKAGNL NYAIGHTDGD YILIFDCDHV PTRAFLQLTM GWMVEDPKIA LMQTPHHFYS PDPFQRNLSA GYRTPPEGNL FYGVVQDGND FWDATFFCGS CAILRRTAIE QIGGFATQTV TEDAHTALKM QRLGWSTAYL RIPLAGGLAT ERLILHIGQR VRWARGMLQI FRIDNPLFGR GLSWGQRLCY LSAMTSFLFA VPRVIFLSSP LAFLFFGQNI IAASPLALLA YAIPHMFHAV GTASKINKGW RYSFWSEVYE TTMALFLVRV TIVTLLSPSR GKFNVTDKGG LLEKGYFDLG AVYPNIILGL IMFGGLARGV YELSFGHLDQ IAERAYLLNS AWAMLSLIII LAAIAVGRET QQKRNSHRIP ATIPVEVANA DGSIIVTGVT EDLSMGGAAV KMSWPAKLSG PTPVYIRTVL DGEELILPAR IIRAGNGRGI FIWTIDNLQQ EFSVIRLVFG RADAWVDWGN YKADRPLLSL MDMVLSVKGL FRSSGDIVHR SSPTKPSAGN ALSDDTNNPS RKERVLKGTV KMVSLLALLT FASSAQAASA PRAVAAKAPA HQPEASDLPP LPALLPATSG AAQAGSGDAG ANGPGSPTGQ PLAADSADAL VENAENTSDT ATVHNYTLKD LGAAGSITMR GLAPLQGIEF GIPSDQLVTS ARLVLSGSMS PNLRPETNSV TMTLNEQYIG TLRPDPAHPT FGPMSFEINP IFFVSGNRLN FNFASGSKGC SDITNDTLWA TISQNSQLQI TTIALPPRRL LSRLPQPFYD KNVRQHVTVP MVLAQTYDPQ ILKSAGILAS WFGKQTDFLG VTFPVSSTIP QSGNAILIGV ADELPTSLGR PQVNGPAVLE LPNPSDANAT ILVVTGRDRD EVITASKGIA FASAPLPTDS HMDVAPVDIA PRKPNDAPSF IAMDHPVRFG DLVTASKLQG TGFTSGVLSV PFRIPPDLYT WRNRPYKMQV RFRSPAGEAK DVEKSRLDVG INEVYLHSYP LRETHGLIGA VLQGVGLARP ASGMQVHDLD VPPWTVFGQD QLNFYFDAMP LARGICQSGA ANNAFHLGLD PDSTIDFSRA HHIAQMPNLA YMATVGFPFT TYADLSQTAV VLPEHPNAAT VGAYLDLMGF MGAATWYPVA GVDIVSADHV SDVADRNLLV ISTLATSGEI APLLSRSSYE VADGHLRTVS HASALDNAIK AVDDPLTAFR DRDSKPQDVD TPLTGGVGAM IEAESPLTAG RTVLALLSSD GAGLNNLLQM LGERKKQANI QGDLVVAHGE DLSSYRTSPV YTIGTLPLWL WPDWYMHNRP VRVLLVGLLG CILIVSVLAR ALARHATRRF KQLEDERRKS |
-実験情報
-構造解析
| 手法 |  ネガティブ染色法 ネガティブ染色法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.2 |
|---|---|
| 染色 | タイプ: NEGATIVE / 材質: uranyl formate |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI SPIRIT |
|---|---|
| 電子線 | 加速電圧: 120 kV / 電子線源: TUNGSTEN HAIRPIN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy Bright-field microscopy |
| 撮影 | フィルム・検出器のモデル: FEI EAGLE (4k x 4k) / 平均電子線量: 20.0 e/Å2 |
| 実験機器 |  モデル: Tecnai Spirit / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期 角度割当 | タイプ: NOT APPLICABLE |
|---|---|
| 最終 角度割当 | タイプ: NOT APPLICABLE |
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 23.4 Å / 解像度の算出法: FSC 0.5 CUT-OFF / 使用した粒子像数: 3168 |
 ムービー
ムービー コントローラー
コントローラー