+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 8qg0 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Archaeoglobus fulgidus AfAgo complex with AfAgo-N protein (fAfAgo) bound with 17 nt RNA guide and 17 nt DNA target | |||||||||
 要素 要素 |
| |||||||||
 キーワード キーワード |  DNA BINDING PROTEIN (DNA結合タンパク質) / DNA BINDING PROTEIN (DNA結合タンパク質) /  ARGONAUTE (アルゴノート (タンパク質)) / ARGONAUTE (アルゴノート (タンパク質)) /  PIWI DOMAIN / PIWI DOMAIN /  PROTEIN-DNA COMPLEX (デオキシリボ核酸) PROTEIN-DNA COMPLEX (デオキシリボ核酸) | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |    Archaeoglobus fulgidus (アルカエオグロブス属) Archaeoglobus fulgidus (アルカエオグロブス属)  Escherichia coli (大腸菌) Escherichia coli (大腸菌) | |||||||||
| 手法 |  電子顕微鏡法 / 電子顕微鏡法 /  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.43 Å クライオ電子顕微鏡法 / 解像度: 3.43 Å | |||||||||
 データ登録者 データ登録者 | Manakova, E.N. / Zaremba, M. / Pocevicuite, R. / Golovinas, E. / Zagorskaite, E. / Silanskas, A. | |||||||||
| 資金援助 | リトアニア, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nucleic Acids Res / 年: 2024 ジャーナル: Nucleic Acids Res / 年: 2024タイトル: The missing part: the Archaeoglobus fulgidus Argonaute forms a functional heterodimer with an N-L1-L2 domain protein. 著者: Elena Manakova / Edvardas Golovinas / Reda Pocevičiūtė / Giedrius Sasnauskas / Arunas Silanskas / Danielis Rutkauskas / Marija Jankunec / Evelina Zagorskaitė / Edvinas Jurgelaitis / ...著者: Elena Manakova / Edvardas Golovinas / Reda Pocevičiūtė / Giedrius Sasnauskas / Arunas Silanskas / Danielis Rutkauskas / Marija Jankunec / Evelina Zagorskaitė / Edvinas Jurgelaitis / Algirdas Grybauskas / Česlovas Venclovas / Mindaugas Zaremba 要旨: Argonaute (Ago) proteins are present in all three domains of life (bacteria, archaea and eukaryotes). They use small (15-30 nucleotides) oligonucleotide guides to bind complementary nucleic acid ...Argonaute (Ago) proteins are present in all three domains of life (bacteria, archaea and eukaryotes). They use small (15-30 nucleotides) oligonucleotide guides to bind complementary nucleic acid targets and are responsible for gene expression regulation, mobile genome element silencing, and defence against viruses or plasmids. According to their domain organization, Agos are divided into long and short Agos. Long Agos found in prokaryotes (long-A and long-B pAgos) and eukaryotes (eAgos) comprise four major functional domains (N, PAZ, MID and PIWI) and two structural linker domains L1 and L2. The majority (∼60%) of pAgos are short pAgos, containing only the MID and inactive PIWI domains. Here we focus on the prokaryotic Argonaute AfAgo from Archaeoglobus fulgidus DSM4304. Although phylogenetically classified as a long-B pAgo, AfAgo contains only MID and catalytically inactive PIWI domains, akin to short pAgos. We show that AfAgo forms a heterodimeric complex with a protein encoded upstream in the same operon, which is a structural equivalent of the N-L1-L2 domains of long pAgos. This complex, structurally equivalent to a long PAZ-less pAgo, outperforms standalone AfAgo in guide RNA-mediated target DNA binding. Our findings provide a missing piece to one of the first and the most studied pAgos. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  8qg0.cif.gz 8qg0.cif.gz | 151.4 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb8qg0.ent.gz pdb8qg0.ent.gz | 111 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  8qg0.json.gz 8qg0.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/qg/8qg0 https://data.pdbj.org/pub/pdb/validation_reports/qg/8qg0 ftp://data.pdbj.org/pub/pdb/validation_reports/qg/8qg0 ftp://data.pdbj.org/pub/pdb/validation_reports/qg/8qg0 | HTTPS FTP |
|---|
-関連構造データ
| 関連構造データ | 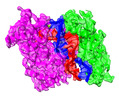 18386MC  8ok9C  8oldC  8oljC  8pvvC C: 同じ文献を引用 ( M: このデータのモデリングに利用したマップデータ |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
| その他のデータベース |
|
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
| #1: タンパク質 |  分子量: 49302.434 Da / 分子数: 1 / 由来タイプ: 組換発現 由来: (組換発現)    Archaeoglobus fulgidus (アルカエオグロブス属) Archaeoglobus fulgidus (アルカエオグロブス属)株: DSM4304 / 遺伝子: XD48_2091 / プラスミド: pBAD / 発現宿主:   Escherichia coli BL21(DE3) (大腸菌) / 参照: UniProt: A0A101DYI0 Escherichia coli BL21(DE3) (大腸菌) / 参照: UniProt: A0A101DYI0 |
|---|---|
| #2: タンパク質 | 分子量: 31356.576 Da / 分子数: 1 / Mutation: N-terminal His-tag / 由来タイプ: 組換発現 由来: (組換発現)    Archaeoglobus fulgidus (アルカエオグロブス属) Archaeoglobus fulgidus (アルカエオグロブス属)株: DSM8774 / 遺伝子: AFULGI_00014290 / プラスミド: pBAD / 発現宿主:   Escherichia coli BL21(DE3) (大腸菌) / 参照: UniProt: A0A075WKW4 Escherichia coli BL21(DE3) (大腸菌) / 参照: UniProt: A0A075WKW4 |
| #3: DNA鎖 | 分子量: 5202.384 Da / 分子数: 1 / 由来タイプ: 合成 / 由来: (合成)   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| #4: RNA鎖 | 分子量: 5427.286 Da / 分子数: 1 / 由来タイプ: 合成 / 由来: (合成)   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
-実験情報
-実験
| 実験 | 手法:  電子顕微鏡法 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法:  単粒子再構成法 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 |
| ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 分子量 | 値: 0.1 MDa / 実験値: YES | ||||||||||||||||||||||||||||||||||||
| 由来(天然) |
| ||||||||||||||||||||||||||||||||||||
| 由来(組換発現) |
| ||||||||||||||||||||||||||||||||||||
| 緩衝液 | pH: 8.5 | ||||||||||||||||||||||||||||||||||||
| 緩衝液成分 |
| ||||||||||||||||||||||||||||||||||||
| 試料 | 濃度: 1 mg/ml / 包埋: NO / シャドウイング: NO / 染色 : NO / 凍結 : NO / 凍結 : YES : YES詳細: fAfAgo complex with 17/17 guide-target heteroduplexes was mixed and applied on grid | ||||||||||||||||||||||||||||||||||||
| 試料支持 | グリッドの材料: COPPER / グリッドのサイズ: 300 divisions/in. / グリッドのタイプ: Quantifoil R1.2/1.3 | ||||||||||||||||||||||||||||||||||||
急速凍結 | 装置: FEI VITROBOT MARK IV / 凍結剤: ETHANE / 湿度: 95 % / 凍結前の試料温度: 277 K |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 顕微鏡 | モデル: TFS GLACIOS |
|---|---|
| 電子銃 | 電子線源 : :  FIELD EMISSION GUN / 加速電圧: 200 kV / 照射モード: FLOOD BEAM FIELD EMISSION GUN / 加速電圧: 200 kV / 照射モード: FLOOD BEAM |
| 電子レンズ | モード: BRIGHT FIELD Bright-field microscopy / 倍率(公称値): 92000 X / 最大 デフォーカス(公称値): 2000 nm / 最小 デフォーカス(公称値): 1000 nm / Cs Bright-field microscopy / 倍率(公称値): 92000 X / 最大 デフォーカス(公称値): 2000 nm / 最小 デフォーカス(公称値): 1000 nm / Cs : 2.7 mm / C2レンズ絞り径: 100 µm / アライメント法: BASIC : 2.7 mm / C2レンズ絞り径: 100 µm / アライメント法: BASIC |
| 試料ホルダ | 凍結剤: NITROGEN / 試料ホルダーモデル: OTHER |
| 撮影 | 平均露光時間: 46.33 sec. / 電子線照射量: 31 e/Å2 / 検出モード: COUNTING フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 撮影したグリッド数: 1 / 実像数: 2152 |
| 画像スキャン | 横: 4000 / 縦: 4000 |
- 解析
解析
| EMソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
CTF補正 | タイプ: NONE | ||||||||||||||||||||||||||||||||||||||||||||||||
| 粒子像の選択 | 選択した粒子像数: 1480423 / 詳細: Blob particle picking | ||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 点対称性 : C1 (非対称) : C1 (非対称) | ||||||||||||||||||||||||||||||||||||||||||||||||
3次元再構成 | 解像度: 3.43 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 364005 / 対称性のタイプ: POINT | ||||||||||||||||||||||||||||||||||||||||||||||||
| 原子モデル構築 | B value: 48 / プロトコル: FLEXIBLE FIT / 空間: REAL | ||||||||||||||||||||||||||||||||||||||||||||||||
| 原子モデル構築 | 3D fitting-ID: 1 / Accession code: 8ok9 / 詳細: fAfAgo complex was used as initial model / Initial refinement model-ID: 1 / PDB-ID: 8ok9 / Source name: PDB / タイプ: experimental model
| ||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー




 PDBj
PDBj































































