+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 8q84 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
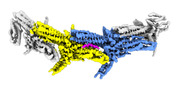
| タイトル | Outer kinetochore Dam1 protomer dimer Ndc80-Nuf2 coiled-coil complex | |||||||||
 要素 要素 |
| |||||||||
 キーワード キーワード |  CELL CYCLE (細胞周期) / CELL CYCLE (細胞周期) /  Kinetochore (動原体) / Kinetochore (動原体) /  microtubule (微小管) / error correction / microtubule (微小管) / error correction /  chromosome segregation chromosome segregation | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報mitotic spindle polar microtubule /  Ndc80 complex / DASH complex / protein transport along microtubule to mitotic spindle pole body / mitotic sister chromatid biorientation / kinetochore organization / positive regulation of attachment of spindle microtubules to kinetochore / mitotic spindle pole body / meiotic chromosome segregation / mitotic spindle midzone ...mitotic spindle polar microtubule / Ndc80 complex / DASH complex / protein transport along microtubule to mitotic spindle pole body / mitotic sister chromatid biorientation / kinetochore organization / positive regulation of attachment of spindle microtubules to kinetochore / mitotic spindle pole body / meiotic chromosome segregation / mitotic spindle midzone ...mitotic spindle polar microtubule /  Ndc80 complex / DASH complex / protein transport along microtubule to mitotic spindle pole body / mitotic sister chromatid biorientation / kinetochore organization / positive regulation of attachment of spindle microtubules to kinetochore / mitotic spindle pole body / meiotic chromosome segregation / mitotic spindle midzone / attachment of spindle microtubules to kinetochore / attachment of mitotic spindle microtubules to kinetochore / condensed chromosome, centromeric region / Ndc80 complex / DASH complex / protein transport along microtubule to mitotic spindle pole body / mitotic sister chromatid biorientation / kinetochore organization / positive regulation of attachment of spindle microtubules to kinetochore / mitotic spindle pole body / meiotic chromosome segregation / mitotic spindle midzone / attachment of spindle microtubules to kinetochore / attachment of mitotic spindle microtubules to kinetochore / condensed chromosome, centromeric region /  spindle pole body / protein localization to kinetochore / positive regulation of microtubule polymerization / mitotic spindle organization / spindle pole body / protein localization to kinetochore / positive regulation of microtubule polymerization / mitotic spindle organization /  chromosome segregation / spindle microtubule / chromosome segregation / spindle microtubule /  紡錘体 / 紡錘体 /  動原体 / 動原体 /  紡錘体 / 紡錘体 /  微小管 / 微小管 /  細胞分裂 / protein-containing complex binding / identical protein binding / 細胞分裂 / protein-containing complex binding / identical protein binding /  細胞核 / 細胞核 /  細胞質 細胞質類似検索 - 分子機能 | |||||||||
| 生物種 |   Saccharomyces cerevisiae (パン酵母) Saccharomyces cerevisiae (パン酵母) | |||||||||
| 手法 |  電子顕微鏡法 / 電子顕微鏡法 /  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.15 Å クライオ電子顕微鏡法 / 解像度: 3.15 Å | |||||||||
 データ登録者 データ登録者 | Muir, K.W. / Barford, D. | |||||||||
| 資金援助 |  英国, 2件 英国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Science / 年: 2023 ジャーナル: Science / 年: 2023タイトル: Structural mechanism of outer kinetochore Dam1-Ndc80 complex assembly on microtubules. 著者: Kyle W Muir / Christopher Batters / Tom Dendooven / Jing Yang / Ziguo Zhang / Alister Burt / David Barford /  要旨: Kinetochores couple chromosomes to the mitotic spindle to segregate the genome during cell division. An error correction mechanism drives the turnover of kinetochore-microtubule attachments until ...Kinetochores couple chromosomes to the mitotic spindle to segregate the genome during cell division. An error correction mechanism drives the turnover of kinetochore-microtubule attachments until biorientation is achieved. The structural basis for how kinetochore-mediated chromosome segregation is accomplished and regulated remains an outstanding question. In this work, we describe the cryo-electron microscopy structure of the budding yeast outer kinetochore Ndc80 and Dam1 ring complexes assembled onto microtubules. Complex assembly occurs through multiple interfaces, and a staple within Dam1 aids ring assembly. Perturbation of key interfaces suppresses yeast viability. Force-rupture assays indicated that this is a consequence of impaired kinetochore-microtubule attachment. The presence of error correction phosphorylation sites at Ndc80-Dam1 ring complex interfaces and the Dam1 staple explains how kinetochore-microtubule attachments are destabilized and reset. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  8q84.cif.gz 8q84.cif.gz | 608.8 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb8q84.ent.gz pdb8q84.ent.gz | 表示 |  PDB形式 PDB形式 | |
| PDBx/mmJSON形式 |  8q84.json.gz 8q84.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/q8/8q84 https://data.pdbj.org/pub/pdb/validation_reports/q8/8q84 ftp://data.pdbj.org/pub/pdb/validation_reports/q8/8q84 ftp://data.pdbj.org/pub/pdb/validation_reports/q8/8q84 | HTTPS FTP |
|---|
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
-Kinetochore protein ... , 2種, 4分子 AFBG
| #1: タンパク質 |  動原体 動原体分子量: 80609.375 Da / 分子数: 2 / 由来タイプ: 組換発現 由来: (組換発現)   Saccharomyces cerevisiae (パン酵母) Saccharomyces cerevisiae (パン酵母)遺伝子: NDC80 / 発現宿主:   Trichoplusia ni (イラクサキンウワバ) / 参照: UniProt: P40460 Trichoplusia ni (イラクサキンウワバ) / 参照: UniProt: P40460#2: タンパク質 |  動原体 動原体分子量: 53025.949 Da / 分子数: 2 / 由来タイプ: 組換発現 由来: (組換発現)   Saccharomyces cerevisiae (パン酵母) Saccharomyces cerevisiae (パン酵母)遺伝子: NUF2 / 発現宿主:   Trichoplusia ni (イラクサキンウワバ) / 参照: UniProt: P33895 Trichoplusia ni (イラクサキンウワバ) / 参照: UniProt: P33895 |
|---|
-DASH complex subunit ... , 10種, 21分子 IUeJVKWLXMYNZOaPbQcRd
| #3: タンパク質 | 分子量: 38477.871 Da / 分子数: 3 / 由来タイプ: 組換発現 由来: (組換発現)   Saccharomyces cerevisiae (パン酵母) Saccharomyces cerevisiae (パン酵母)遺伝子: DAM1 / 発現宿主:   Trichoplusia ni (イラクサキンウワバ) / 参照: UniProt: P53267 Trichoplusia ni (イラクサキンウワバ) / 参照: UniProt: P53267#4: タンパク質 | 分子量: 27515.008 Da / 分子数: 2 / 由来タイプ: 組換発現 由来: (組換発現)   Saccharomyces cerevisiae (パン酵母) Saccharomyces cerevisiae (パン酵母)遺伝子: DUO1 / 発現宿主:   Trichoplusia ni (イラクサキンウワバ) / 参照: UniProt: P53168 Trichoplusia ni (イラクサキンウワバ) / 参照: UniProt: P53168#5: タンパク質 | 分子量: 15084.501 Da / 分子数: 2 / 由来タイプ: 組換発現 由来: (組換発現)   Saccharomyces cerevisiae (パン酵母) Saccharomyces cerevisiae (パン酵母)遺伝子: DAD2 / 発現宿主:   Trichoplusia ni (イラクサキンウワバ) / 参照: UniProt: P36162 Trichoplusia ni (イラクサキンウワバ) / 参照: UniProt: P36162#6: タンパク質 | 分子量: 10522.616 Da / 分子数: 2 / 由来タイプ: 組換発現 由来: (組換発現)   Saccharomyces cerevisiae (パン酵母) Saccharomyces cerevisiae (パン酵母)遺伝子: DAD1 / 発現宿主:   Trichoplusia ni (イラクサキンウワバ) / 参照: UniProt: Q12248 Trichoplusia ni (イラクサキンウワバ) / 参照: UniProt: Q12248#7: タンパク質 | 分子量: 8164.255 Da / 分子数: 2 / 由来タイプ: 組換発現 由来: (組換発現)   Saccharomyces cerevisiae (パン酵母) Saccharomyces cerevisiae (パン酵母)遺伝子: DAD4 / 発現宿主:   Trichoplusia ni (イラクサキンウワバ) / 参照: UniProt: P69851 Trichoplusia ni (イラクサキンウワバ) / 参照: UniProt: P69851#8: タンパク質 | 分子量: 10862.234 Da / 分子数: 2 / 由来タイプ: 組換発現 由来: (組換発現)   Saccharomyces cerevisiae (パン酵母) Saccharomyces cerevisiae (パン酵母)遺伝子: DAD3 / 発現宿主:   Trichoplusia ni (イラクサキンウワバ) / 参照: UniProt: P69850 Trichoplusia ni (イラクサキンウワバ) / 参照: UniProt: P69850#9: タンパク質 | 分子量: 34131.812 Da / 分子数: 2 / 由来タイプ: 組換発現 由来: (組換発現)   Saccharomyces cerevisiae (パン酵母) Saccharomyces cerevisiae (パン酵母)遺伝子: SPC34 / 発現宿主:   Trichoplusia ni (イラクサキンウワバ) / 参照: UniProt: P36131 Trichoplusia ni (イラクサキンウワバ) / 参照: UniProt: P36131#10: タンパク質 | 分子量: 32106.631 Da / 分子数: 2 / 由来タイプ: 組換発現 由来: (組換発現)   Saccharomyces cerevisiae (パン酵母) Saccharomyces cerevisiae (パン酵母)遺伝子: ASK1 / 発現宿主:   Trichoplusia ni (イラクサキンウワバ) / 参照: UniProt: P35734 Trichoplusia ni (イラクサキンウワバ) / 参照: UniProt: P35734#11: タンパク質 | 分子量: 8097.205 Da / 分子数: 2 / 由来タイプ: 組換発現 由来: (組換発現)   Saccharomyces cerevisiae (パン酵母) Saccharomyces cerevisiae (パン酵母)遺伝子: HSK3 / 発現宿主:   Trichoplusia ni (イラクサキンウワバ) / 参照: UniProt: P69852 Trichoplusia ni (イラクサキンウワバ) / 参照: UniProt: P69852#12: タンパク質 | 分子量: 18935.559 Da / 分子数: 2 / 由来タイプ: 組換発現 由来: (組換発現)   Saccharomyces cerevisiae (パン酵母) Saccharomyces cerevisiae (パン酵母)遺伝子: SPC19 / 発現宿主:   Trichoplusia ni (イラクサキンウワバ) / 参照: UniProt: Q03954 Trichoplusia ni (イラクサキンウワバ) / 参照: UniProt: Q03954 |
|---|
-実験情報
-実験
| 実験 | 手法:  電子顕微鏡法 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法:  単粒子再構成法 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 | 名称: Outer kinetochore Dam1 protomer dimer with staple and Ndc80-Nuf2 coiled-coils タイプ: COMPLEX / Entity ID: all / 由来: RECOMBINANT |
|---|---|
| 分子量 | 値: 673772.36 / 実験値: NO |
| 由来(天然) | 生物種:   Saccharomyces cerevisiae (パン酵母) Saccharomyces cerevisiae (パン酵母) |
| 由来(組換発現) | 生物種:   Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 緩衝液 | pH: 6.8 |
| 試料 | 包埋: NO / シャドウイング: NO / 染色 : NO / 凍結 : NO / 凍結 : YES : YES |
急速凍結 | 凍結剤: ETHANE |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: TFS KRIOS |
| 電子銃 | 電子線源 : :  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM |
| 電子レンズ | モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 3000 nm / 最小 デフォーカス(公称値): 1200 nm Bright-field microscopy / 最大 デフォーカス(公称値): 3000 nm / 最小 デフォーカス(公称値): 1200 nm |
| 撮影 | 電子線照射量: 50 e/Å2 フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) |
- 解析
解析
| EMソフトウェア |
| ||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
CTF補正 | タイプ: PHASE FLIPPING AND AMPLITUDE CORRECTION | ||||||||||||||||||||||||||||
3次元再構成 | 解像度: 3.15 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 248732 / 対称性のタイプ: POINT | ||||||||||||||||||||||||||||
| 原子モデル構築 | B value: 540.98 / プロトコル: OTHER / 空間: REAL 詳細: Initial rigid body fitting was performed in chimera, with manual correction in coot and real-space refinement in PHENIX | ||||||||||||||||||||||||||||
| 原子モデル構築 | Source name: AlphaFold / タイプ: in silico model | ||||||||||||||||||||||||||||
| 精密化 | 交差検証法: NONE 立体化学のターゲット値: GeoStd + Monomer Library + CDL v1.2 | ||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 540.98 Å2 | ||||||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー





 PDBj
PDBj

