+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 7yu4 | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | Human Lysophosphatidic Acid Receptor 1-Gi complex bound to ONO-0740556, focused on receptor | ||||||
 要素 要素 | Lysophosphatidic acid receptor 1 | ||||||
 キーワード キーワード |  MEMBRANE PROTEIN (膜タンパク質) / MEMBRANE PROTEIN (膜タンパク質) /  GPCR (Gタンパク質共役受容体) GPCR (Gタンパク質共役受容体) | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報cellular response to 1-oleoyl-sn-glycerol 3-phosphate /  lysophosphatidic acid receptor activity / positive regulation of smooth muscle cell chemotaxis / calmodulin dependent kinase signaling pathway / Lysosphingolipid and LPA receptors / lysophosphatidic acid receptor activity / positive regulation of smooth muscle cell chemotaxis / calmodulin dependent kinase signaling pathway / Lysosphingolipid and LPA receptors /  lysophosphatidic acid binding / negative regulation of cilium assembly / regulation of synaptic vesicle cycle / cellular response to oxygen levels / oligodendrocyte development ...cellular response to 1-oleoyl-sn-glycerol 3-phosphate / lysophosphatidic acid binding / negative regulation of cilium assembly / regulation of synaptic vesicle cycle / cellular response to oxygen levels / oligodendrocyte development ...cellular response to 1-oleoyl-sn-glycerol 3-phosphate /  lysophosphatidic acid receptor activity / positive regulation of smooth muscle cell chemotaxis / calmodulin dependent kinase signaling pathway / Lysosphingolipid and LPA receptors / lysophosphatidic acid receptor activity / positive regulation of smooth muscle cell chemotaxis / calmodulin dependent kinase signaling pathway / Lysosphingolipid and LPA receptors /  lysophosphatidic acid binding / negative regulation of cilium assembly / regulation of synaptic vesicle cycle / cellular response to oxygen levels / oligodendrocyte development / corpus callosum development / bleb assembly / lysophosphatidic acid binding / negative regulation of cilium assembly / regulation of synaptic vesicle cycle / cellular response to oxygen levels / oligodendrocyte development / corpus callosum development / bleb assembly /  regulation of metabolic process / optic nerve development / negative regulation of cAMP-mediated signaling / regulation of postsynaptic neurotransmitter receptor internalization / positive regulation of Rho protein signal transduction / positive regulation of dendritic spine development / activation of phospholipase C activity / G-protein alpha-subunit binding / GABA-ergic synapse / positive regulation of stress fiber assembly / adenylate cyclase-inhibiting G protein-coupled receptor signaling pathway / regulation of metabolic process / optic nerve development / negative regulation of cAMP-mediated signaling / regulation of postsynaptic neurotransmitter receptor internalization / positive regulation of Rho protein signal transduction / positive regulation of dendritic spine development / activation of phospholipase C activity / G-protein alpha-subunit binding / GABA-ergic synapse / positive regulation of stress fiber assembly / adenylate cyclase-inhibiting G protein-coupled receptor signaling pathway /  髄鞘 / 髄鞘 /  神経発生 / cerebellum development / cell chemotaxis / dendritic shaft / 神経発生 / cerebellum development / cell chemotaxis / dendritic shaft /  PDZ domain binding / G protein-coupled receptor activity / adenylate cyclase-activating G protein-coupled receptor signaling pathway / negative regulation of neuron projection development / PDZ domain binding / G protein-coupled receptor activity / adenylate cyclase-activating G protein-coupled receptor signaling pathway / negative regulation of neuron projection development /  presynaptic membrane / phospholipase C-activating G protein-coupled receptor signaling pathway / regulation of cell shape / G alpha (i) signalling events / positive regulation of cytosolic calcium ion concentration / G alpha (q) signalling events / presynaptic membrane / phospholipase C-activating G protein-coupled receptor signaling pathway / regulation of cell shape / G alpha (i) signalling events / positive regulation of cytosolic calcium ion concentration / G alpha (q) signalling events /  postsynaptic membrane / positive regulation of canonical NF-kappaB signal transduction / postsynaptic membrane / positive regulation of canonical NF-kappaB signal transduction /  樹状突起スパイン / positive regulation of MAPK cascade / 樹状突起スパイン / positive regulation of MAPK cascade /  エンドソーム / positive regulation of apoptotic process / G protein-coupled receptor signaling pathway / neuronal cell body / glutamatergic synapse / エンドソーム / positive regulation of apoptotic process / G protein-coupled receptor signaling pathway / neuronal cell body / glutamatergic synapse /  細胞膜 / 細胞膜 /  細胞膜 / 細胞膜 /  細胞質 細胞質類似検索 - 分子機能 | ||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||
| 手法 |  電子顕微鏡法 / 電子顕微鏡法 /  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.7 Å クライオ電子顕微鏡法 / 解像度: 3.7 Å | ||||||
 データ登録者 データ登録者 | Akasaka, H. / Shihoya, W. / Nureki, O. | ||||||
| 資金援助 |  日本, 1件 日本, 1件
| ||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2022 ジャーナル: Nat Commun / 年: 2022タイトル: Structure of the active G-coupled human lysophosphatidic acid receptor 1 complexed with a potent agonist. 著者: Hiroaki Akasaka / Tatsuki Tanaka / Fumiya K Sano / Yuma Matsuzaki / Wataru Shihoya / Osamu Nureki /  要旨: Lysophosphatidic acid receptor 1 (LPA) is one of the six G protein-coupled receptors activated by the bioactive lipid, lysophosphatidic acid (LPA). LPA is a drug target for various diseases, ...Lysophosphatidic acid receptor 1 (LPA) is one of the six G protein-coupled receptors activated by the bioactive lipid, lysophosphatidic acid (LPA). LPA is a drug target for various diseases, including cancer, inflammation, and neuropathic pain. Notably, LPA agonists have potential therapeutic value for obesity and urinary incontinence. Here, we report a cryo-electron microscopy structure of the active human LPA-G complex bound to ONO-0740556, an LPA analog with more potent activity against LPA. Our structure elucidated the details of the agonist binding mode and receptor activation mechanism mediated by rearrangements of transmembrane segment 7 and the central hydrophobic core. A structural comparison of LPA and other phylogenetically-related lipid-sensing GPCRs identified the structural determinants for lipid preference of LPA. Moreover, we characterized the structural polymorphisms at the receptor-G-protein interface, which potentially reflect the G-protein dissociation process. Our study provides insights into the detailed mechanism of LPA binding to agonists and paves the way toward the design of drug-like agonists targeting LPA. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  7yu4.cif.gz 7yu4.cif.gz | 63.8 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb7yu4.ent.gz pdb7yu4.ent.gz | 45.6 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  7yu4.json.gz 7yu4.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/yu/7yu4 https://data.pdbj.org/pub/pdb/validation_reports/yu/7yu4 ftp://data.pdbj.org/pub/pdb/validation_reports/yu/7yu4 ftp://data.pdbj.org/pub/pdb/validation_reports/yu/7yu4 | HTTPS FTP |
|---|
-関連構造データ
| 関連構造データ |  34098MC  7yu3C  7yu5C  7yu6C  7yu7C  7yu8C M: このデータのモデリングに利用したマップデータ C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
| #1: タンパク質 |  / LPA receptor 1 / LPA-1 / Lysophosphatidic acid receptor Edg-2 / LPA receptor 1 / LPA-1 / Lysophosphatidic acid receptor Edg-2分子量: 42936.004 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)   Homo sapiens (ヒト) / 遺伝子: LPAR1, EDG2, LPA1 Homo sapiens (ヒト) / 遺伝子: LPAR1, EDG2, LPA1発現宿主:   Spodoptera frugiperda (ツマジロクサヨトウ) Spodoptera frugiperda (ツマジロクサヨトウ)参照: UniProt: Q92633 |
|---|---|
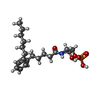
| #2: 化合物 | ChemComp-K6L / [( |
| 研究の焦点であるリガンドがあるか | Y |
-実験情報
-実験
| 実験 | 手法:  電子顕微鏡法 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法:  単粒子再構成法 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 | 名称: Human Lysophosphatidic Acid Receptor 1 bound to ONO-0740556 タイプ: COMPLEX / Entity ID: #1 / 由来: NATURAL |
|---|---|
| 分子量 | 実験値: NO |
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 由来(組換発現) | 生物種:   Spodoptera frugiperda (ツマジロクサヨトウ) Spodoptera frugiperda (ツマジロクサヨトウ) |
| 緩衝液 | pH: 8 |
| 試料 | 濃度: 7 mg/ml / 包埋: NO / シャドウイング: NO / 染色 : NO / 凍結 : NO / 凍結 : YES : YES |
急速凍結 | 凍結剤: ETHANE |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: FEI TITAN KRIOS |
| 電子銃 | 電子線源 : :  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM |
| 電子レンズ | モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 2500 nm / 最小 デフォーカス(公称値): 500 nm Bright-field microscopy / 最大 デフォーカス(公称値): 2500 nm / 最小 デフォーカス(公称値): 500 nm |
| 撮影 | 電子線照射量: 50 e/Å2 / フィルム・検出器のモデル: GATAN K3 (6k x 4k) |
- 解析
解析
| ソフトウェア | 名称: REFMAC / バージョン: 5.8.0267 / 分類: 精密化 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
CTF補正 | タイプ: PHASE FLIPPING AND AMPLITUDE CORRECTION | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
3次元再構成 | 解像度: 3.7 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 181071 / 対称性のタイプ: POINT | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 | 解像度: 3.3→88.44 Å / Cor.coef. Fo:Fc: 0.803 / SU B: 32.341 / SU ML: 0.472 / ESU R: 0.592 立体化学のターゲット値: MAXIMUM LIKELIHOOD WITH PHASES 詳細: HYDROGENS HAVE BEEN ADDED IN THE RIDING POSITIONS
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | 溶媒モデル: PARAMETERS FOR MASK CACLULATION | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 99.316 Å2 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: 1 / 合計: 2251 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー








 PDBj
PDBj