+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 5vnw | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Crystal structure of Nb.b201 bound to human serum albumin | |||||||||
 要素 要素 |
| |||||||||
 キーワード キーワード | TRANSPORT PROTEIN/DE NOVO PROTEIN /  nanobody (ナノボディ) / synthetic protein / nanobody (ナノボディ) / synthetic protein /  llama (リャマ) / llama (リャマ) /  camelid (ラクダ科) / TRANSPORT PROTEIN-DE NOVO PROTEIN complex camelid (ラクダ科) / TRANSPORT PROTEIN-DE NOVO PROTEIN complex | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報cellular response to calcium ion starvation /  exogenous protein binding / Ciprofloxacin ADME / exogenous protein binding / Ciprofloxacin ADME /  enterobactin binding / enterobactin binding /  ヘム / HDL remodeling / negative regulation of mitochondrial depolarization / Prednisone ADME / Heme degradation / Aspirin ADME ...cellular response to calcium ion starvation / ヘム / HDL remodeling / negative regulation of mitochondrial depolarization / Prednisone ADME / Heme degradation / Aspirin ADME ...cellular response to calcium ion starvation /  exogenous protein binding / Ciprofloxacin ADME / exogenous protein binding / Ciprofloxacin ADME /  enterobactin binding / enterobactin binding /  ヘム / HDL remodeling / negative regulation of mitochondrial depolarization / Prednisone ADME / Heme degradation / Aspirin ADME / ヘム / HDL remodeling / negative regulation of mitochondrial depolarization / Prednisone ADME / Heme degradation / Aspirin ADME /  antioxidant activity / antioxidant activity /  toxic substance binding / Scavenging of heme from plasma / Recycling of bile acids and salts / cellular response to starvation / platelet alpha granule lumen / toxic substance binding / Scavenging of heme from plasma / Recycling of bile acids and salts / cellular response to starvation / platelet alpha granule lumen /  fatty acid binding / fatty acid binding /  Post-translational protein phosphorylation / Cytoprotection by HMOX1 / Regulation of Insulin-like Growth Factor (IGF) transport and uptake by Insulin-like Growth Factor Binding Proteins (IGFBPs) / Post-translational protein phosphorylation / Cytoprotection by HMOX1 / Regulation of Insulin-like Growth Factor (IGF) transport and uptake by Insulin-like Growth Factor Binding Proteins (IGFBPs) /  pyridoxal phosphate binding / Platelet degranulation / protein-folding chaperone binding / blood microparticle / copper ion binding / pyridoxal phosphate binding / Platelet degranulation / protein-folding chaperone binding / blood microparticle / copper ion binding /  小胞体 / 小胞体 /  ゴルジ体 / ゴルジ体 /  小胞体 / protein-containing complex / 小胞体 / protein-containing complex /  DNA binding / DNA binding /  extracellular space / extracellular exosome / extracellular region / identical protein binding / extracellular space / extracellular exosome / extracellular region / identical protein binding /  細胞核 / 細胞核 /  細胞質 細胞質類似検索 - 分子機能 | |||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト)synthetic construct (人工物) | |||||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2.6 Å 分子置換 / 解像度: 2.6 Å | |||||||||
 データ登録者 データ登録者 | McMahon, C. / Kruse, A.C. | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nat. Struct. Mol. Biol. / 年: 2018 ジャーナル: Nat. Struct. Mol. Biol. / 年: 2018タイトル: Yeast surface display platform for rapid discovery of conformationally selective nanobodies. 著者: McMahon, C. / Baier, A.S. / Pascolutti, R. / Wegrecki, M. / Zheng, S. / Ong, J.X. / Erlandson, S.C. / Hilger, D. / Rasmussen, S.G.F. / Ring, A.M. / Manglik, A. / Kruse, A.C. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  5vnw.cif.gz 5vnw.cif.gz | 572.3 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb5vnw.ent.gz pdb5vnw.ent.gz | 479.4 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  5vnw.json.gz 5vnw.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/vn/5vnw https://data.pdbj.org/pub/pdb/validation_reports/vn/5vnw ftp://data.pdbj.org/pub/pdb/validation_reports/vn/5vnw ftp://data.pdbj.org/pub/pdb/validation_reports/vn/5vnw | HTTPS FTP |
|---|
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||
| 2 | 
| ||||||||
| 単位格子 |
|
- 要素
要素
-タンパク質 / 抗体 , 2種, 4分子 ABCD
| #1: タンパク質 |  分子量: 66571.219 Da / 分子数: 2 / 由来タイプ: 組換発現 / 由来: (組換発現)   Homo sapiens (ヒト) Homo sapiens (ヒト)遺伝子: ALB, GIG20, GIG42, PRO0903, PRO1708, PRO2044, PRO2619, PRO2675, UNQ696/PRO1341 発現宿主:   Saccharomyces cerevisiae (パン酵母) / 参照: UniProt: P02768 Saccharomyces cerevisiae (パン酵母) / 参照: UniProt: P02768#2: 抗体 | 分子量: 13349.759 Da / 分子数: 2 / 由来タイプ: 組換発現 / 由来: (組換発現) synthetic construct (人工物) / 発現宿主:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
|---|
-非ポリマー , 5種, 77分子 

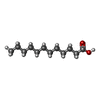






| #3: 化合物 | ChemComp-GOL /  グリセリン グリセリン#4: 化合物 |  パルミチン酸 パルミチン酸#5: 化合物 | ChemComp-DAO / |  ラウリン酸 ラウリン酸#6: 化合物 | ChemComp-BUA / |  酪酸 酪酸#7: 水 | ChemComp-HOH / |  水 水 |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 3 Å3/Da / 溶媒含有率: 59.02 % |
|---|---|
結晶化 | 温度: 293 K / 手法: 蒸気拡散法, シッティングドロップ法 / pH: 8.5 詳細: 1,6-hexanediol, 1-butanol, 1,2-propanediol, 2-propanol, 1,4-butanediol, 1,3-propanediol, Tris(base)/BICINE pH 8.5, PEG 500 MME, PEG 20,0000, sodium chloride, HEPES pH 7.5 |
-データ収集
| 回折 | 平均測定温度: 80 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  APS APS  / ビームライン: 23-ID-D / 波長: 1.033 Å / ビームライン: 23-ID-D / 波長: 1.033 Å |
| 検出器 | タイプ: DECTRIS PILATUS3 6M / 検出器: PIXEL / 日付: 2016年11月4日 |
| 放射 | モノクロメーター: Si(111) / プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長 : 1.033 Å / 相対比: 1 : 1.033 Å / 相対比: 1 |
| 反射 | 解像度: 2.6→46.78 Å / Num. obs: 55905 / % possible obs: 97.7 % / 冗長度: 3.5 % / CC1/2: 0.998 / Rsym value: 0.073 / Net I/σ(I): 8.99 |
| 反射 シェル | 解像度: 2.6→2.76 Å / 冗長度: 3.3 % / Mean I/σ(I) obs: 0.75 / Num. unique all: 8732 / CC1/2: 0.49 / Rsym value: 1.17 / % possible all: 94.4 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法 : :  分子置換 分子置換開始モデル: 5ID7 解像度: 2.6→39.6 Å / SU ML: 0.55 / 交差検証法: FREE R-VALUE / σ(F): 1.96 / 位相誤差: 34.69
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | 減衰半径: 0.9 Å / VDWプローブ半径: 1.11 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 2.6→39.6 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLS | 手法: refined / Refine-ID: X-RAY DIFFRACTION
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLSグループ |
|
 ムービー
ムービー コントローラー
コントローラー













 PDBj
PDBj





















