+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-6340 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Electron cryo-microscopy of a BRCA1-RNAPII transcriptional complex | |||||||||
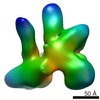
 マップデータ マップデータ | Reconstruction of BRCA1-RNAPII transcriptional complexes derived from human breast cancer cells | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード |  transcription (転写 (生物学)) / transcription (転写 (生物学)) /  DNA repair (DNA修復) / DNA repair (DNA修復) /  cancer (悪性腫瘍) / cancer (悪性腫瘍) /  mRNA (伝令RNA) / mRNA (伝令RNA) /  tumor suppressor (がん抑制遺伝子) / tumor suppressor (がん抑制遺伝子) /  ubiquitin ligase (ユビキチンリガーゼ) ubiquitin ligase (ユビキチンリガーゼ) | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報negative regulation of mRNA 3'-end processing / Defective DNA double strand break response due to BRCA1 loss of function / Defective DNA double strand break response due to BARD1 loss of function / : /  microfibril binding / BRCA1-BARD1 complex / BRCA1-C complex / BRCA1-B complex / BRCA1-A complex / random inactivation of X chromosome ...negative regulation of mRNA 3'-end processing / Defective DNA double strand break response due to BRCA1 loss of function / Defective DNA double strand break response due to BARD1 loss of function / : / microfibril binding / BRCA1-BARD1 complex / BRCA1-C complex / BRCA1-B complex / BRCA1-A complex / random inactivation of X chromosome ...negative regulation of mRNA 3'-end processing / Defective DNA double strand break response due to BRCA1 loss of function / Defective DNA double strand break response due to BARD1 loss of function / : /  microfibril binding / BRCA1-BARD1 complex / BRCA1-C complex / BRCA1-B complex / BRCA1-A complex / random inactivation of X chromosome / negative regulation of centriole replication / negative regulation of intracellular estrogen receptor signaling pathway / gamma-tubulin ring complex / nuclear ubiquitin ligase complex / DNA strand resection involved in replication fork processing / chordate embryonic development / negative regulation of fatty acid biosynthetic process / cellular response to indole-3-methanol / microfibril binding / BRCA1-BARD1 complex / BRCA1-C complex / BRCA1-B complex / BRCA1-A complex / random inactivation of X chromosome / negative regulation of centriole replication / negative regulation of intracellular estrogen receptor signaling pathway / gamma-tubulin ring complex / nuclear ubiquitin ligase complex / DNA strand resection involved in replication fork processing / chordate embryonic development / negative regulation of fatty acid biosynthetic process / cellular response to indole-3-methanol /  相同組換え / lateral element / tissue homeostasis / XY body / protein K6-linked ubiquitination / 相同組換え / lateral element / tissue homeostasis / XY body / protein K6-linked ubiquitination /  regulation of DNA damage checkpoint / regulation of DNA damage checkpoint /  regulation of phosphorylation / dosage compensation by inactivation of X chromosome / negative regulation of gene expression via chromosomal CpG island methylation / Impaired BRCA2 binding to PALB2 / : / mitotic G2/M transition checkpoint / negative regulation of protein export from nucleus / regulation of phosphorylation / dosage compensation by inactivation of X chromosome / negative regulation of gene expression via chromosomal CpG island methylation / Impaired BRCA2 binding to PALB2 / : / mitotic G2/M transition checkpoint / negative regulation of protein export from nucleus /  postreplication repair / postreplication repair /  DNA repair complex / Abortive elongation of HIV-1 transcript in the absence of Tat / FGFR2 alternative splicing / DNA repair complex / Abortive elongation of HIV-1 transcript in the absence of Tat / FGFR2 alternative splicing /  RNA polymerase binding / RNA polymerase binding /  centrosome cycle / Defective homologous recombination repair (HRR) due to BRCA1 loss of function / Defective HDR through Homologous Recombination Repair (HRR) due to PALB2 loss of BRCA1 binding function / Defective HDR through Homologous Recombination Repair (HRR) due to PALB2 loss of BRCA2/RAD51/RAD51C binding function / Homologous DNA Pairing and Strand Exchange / Resolution of D-loop Structures through Synthesis-Dependent Strand Annealing (SDSA) / MicroRNA (miRNA) biogenesis / Resolution of D-loop Structures through Holliday Junction Intermediates / Viral Messenger RNA Synthesis / HDR through Single Strand Annealing (SSA) / Impaired BRCA2 binding to RAD51 / Signaling by FGFR2 IIIa TM / response to ionizing radiation / DNA-binding transcription activator activity / intracellular non-membrane-bounded organelle / RNA Pol II CTD phosphorylation and interaction with CE during HIV infection / RNA Pol II CTD phosphorylation and interaction with CE / Formation of the Early Elongation Complex / Formation of the HIV-1 Early Elongation Complex / mRNA Capping / PIWI-interacting RNA (piRNA) biogenesis / mRNA Splicing - Minor Pathway / HIV Transcription Initiation / RNA Polymerase II HIV Promoter Escape / Transcription of the HIV genome / RNA Polymerase II Promoter Escape / RNA Polymerase II Transcription Pre-Initiation And Promoter Opening / RNA Polymerase II Transcription Initiation / RNA Polymerase II Transcription Initiation And Promoter Clearance / Transcriptional Regulation by E2F6 / mitotic G2 DNA damage checkpoint signaling / Presynaptic phase of homologous DNA pairing and strand exchange / Processing of Capped Intron-Containing Pre-mRNA / Pausing and recovery of Tat-mediated HIV elongation / Tat-mediated HIV elongation arrest and recovery / centrosome cycle / Defective homologous recombination repair (HRR) due to BRCA1 loss of function / Defective HDR through Homologous Recombination Repair (HRR) due to PALB2 loss of BRCA1 binding function / Defective HDR through Homologous Recombination Repair (HRR) due to PALB2 loss of BRCA2/RAD51/RAD51C binding function / Homologous DNA Pairing and Strand Exchange / Resolution of D-loop Structures through Synthesis-Dependent Strand Annealing (SDSA) / MicroRNA (miRNA) biogenesis / Resolution of D-loop Structures through Holliday Junction Intermediates / Viral Messenger RNA Synthesis / HDR through Single Strand Annealing (SSA) / Impaired BRCA2 binding to RAD51 / Signaling by FGFR2 IIIa TM / response to ionizing radiation / DNA-binding transcription activator activity / intracellular non-membrane-bounded organelle / RNA Pol II CTD phosphorylation and interaction with CE during HIV infection / RNA Pol II CTD phosphorylation and interaction with CE / Formation of the Early Elongation Complex / Formation of the HIV-1 Early Elongation Complex / mRNA Capping / PIWI-interacting RNA (piRNA) biogenesis / mRNA Splicing - Minor Pathway / HIV Transcription Initiation / RNA Polymerase II HIV Promoter Escape / Transcription of the HIV genome / RNA Polymerase II Promoter Escape / RNA Polymerase II Transcription Pre-Initiation And Promoter Opening / RNA Polymerase II Transcription Initiation / RNA Polymerase II Transcription Initiation And Promoter Clearance / Transcriptional Regulation by E2F6 / mitotic G2 DNA damage checkpoint signaling / Presynaptic phase of homologous DNA pairing and strand exchange / Processing of Capped Intron-Containing Pre-mRNA / Pausing and recovery of Tat-mediated HIV elongation / Tat-mediated HIV elongation arrest and recovery /  RNA polymerase II activity / RNA polymerase II transcribes snRNA genes / HIV elongation arrest and recovery / Pausing and recovery of HIV elongation / negative regulation of cell cycle / positive regulation of vascular endothelial growth factor production / negative regulation of reactive oxygen species metabolic process / Tat-mediated elongation of the HIV-1 transcript / Formation of HIV-1 elongation complex containing HIV-1 Tat / localization / RNA polymerase II activity / RNA polymerase II transcribes snRNA genes / HIV elongation arrest and recovery / Pausing and recovery of HIV elongation / negative regulation of cell cycle / positive regulation of vascular endothelial growth factor production / negative regulation of reactive oxygen species metabolic process / Tat-mediated elongation of the HIV-1 transcript / Formation of HIV-1 elongation complex containing HIV-1 Tat / localization /  regulation of DNA repair / protein autoubiquitination / Formation of HIV elongation complex in the absence of HIV Tat / regulation of DNA repair / protein autoubiquitination / Formation of HIV elongation complex in the absence of HIV Tat /  RNA polymerase II, core complex / RNA polymerase II, core complex /  ubiquitin ligase complex / SUMOylation of DNA damage response and repair proteins / RNA Polymerase II Transcription Elongation / Formation of RNA Pol II elongation complex / negative regulation of extrinsic apoptotic signaling pathway via death domain receptors / Maturation of protein E / Maturation of protein E / ER Quality Control Compartment (ERQC) / Myoclonic epilepsy of Lafora / FLT3 signaling by CBL mutants / Prevention of phagosomal-lysosomal fusion / IRAK2 mediated activation of TAK1 complex / Alpha-protein kinase 1 signaling pathway / ubiquitin ligase complex / SUMOylation of DNA damage response and repair proteins / RNA Polymerase II Transcription Elongation / Formation of RNA Pol II elongation complex / negative regulation of extrinsic apoptotic signaling pathway via death domain receptors / Maturation of protein E / Maturation of protein E / ER Quality Control Compartment (ERQC) / Myoclonic epilepsy of Lafora / FLT3 signaling by CBL mutants / Prevention of phagosomal-lysosomal fusion / IRAK2 mediated activation of TAK1 complex / Alpha-protein kinase 1 signaling pathway /  グリコーゲン合成 / IRAK1 recruits IKK complex グリコーゲン合成 / IRAK1 recruits IKK complex類似検索 - 分子機能 | |||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / クライオ電子顕微鏡法 /  ネガティブ染色法 / 解像度: 22.0 Å ネガティブ染色法 / 解像度: 22.0 Å | |||||||||
 データ登録者 データ登録者 | Gilmore BL / Winton CE / Demmert AC / Tanner JR / Bowman S / Karageorge V / Patel K / Sheng Z / Kelly DF | |||||||||
 引用 引用 | ジャーナル: Nat Struct Mol Biol / 年: 2012 タイトル: Architecture of the RNA polymerase II preinitiation complex and mechanism of ATP-dependent promoter opening. 著者: Sebastian Grünberg / Linda Warfield / Steven Hahn /  要旨: Yeast RNA polymerase II (Pol II) general transcription factor TFIIE and the TFIIH subunit Ssl2 (yeast ortholog of mammalian XPB) function in the transition of the preinitiation complex (PIC) to the ...Yeast RNA polymerase II (Pol II) general transcription factor TFIIE and the TFIIH subunit Ssl2 (yeast ortholog of mammalian XPB) function in the transition of the preinitiation complex (PIC) to the open complex. We show that the three TFIIE winged-helix (WH) domains form a heterodimer, with the Tfa1 (TFIIEα) WH binding the Pol II clamp and the Tfa2 (TFIIEβ) tandem WH domain encircling promoter DNA that becomes single-stranded in the open complex. Ssl2 lies adjacent to TFIIE, enclosing downstream promoter DNA. Unlike previous proposals, comparison of the PIC and open-complex models strongly suggests that Ssl2 promotes DNA opening by functioning as a double-stranded-DNA translocase, feeding 15 base pairs into the Pol II cleft. Right-handed threading of DNA through the Ssl2 binding groove, combined with the fixed position of upstream promoter DNA, leads to DNA unwinding and the open state. #5:  ジャーナル: Journal of Scientific Reports ジャーナル: Journal of Scientific Reportsタイトル: A Molecular Toolkit to Visualize Native Protein Assemblies in the Context of the Human Disease 著者: Gilmore BL / Winton CE / Demmert AC / Tanner JR / Bowman S / Karageorge V / Patel K / Sheng Z / Kelly DF | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_6340.map.gz emd_6340.map.gz | 283.4 KB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-6340-v30.xml emd-6340-v30.xml emd-6340.xml emd-6340.xml | 23.9 KB 23.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  400_6340.gif 400_6340.gif 80_6340.gif 80_6340.gif | 54.3 KB 4.2 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-6340 http://ftp.pdbj.org/pub/emdb/structures/EMD-6340 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-6340 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-6340 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_6340.map.gz / 形式: CCP4 / 大きさ: 825.2 KB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_6340.map.gz / 形式: CCP4 / 大きさ: 825.2 KB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Reconstruction of BRCA1-RNAPII transcriptional complexes derived from human breast cancer cells | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 5.5 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : BRCA1-RNAP II transcriptional assemblies isolated from hereditary...
| 全体 | 名称: BRCA1-RNAP II transcriptional assemblies isolated from hereditary breast cancer cells |
|---|---|
| 要素 |
|
-超分子 #1000: BRCA1-RNAP II transcriptional assemblies isolated from hereditary...
| 超分子 | 名称: BRCA1-RNAP II transcriptional assemblies isolated from hereditary breast cancer cells タイプ: sample / ID: 1000 集合状態: one BRCA1 molecule binds to one polymerase complex Number unique components: 5 |
|---|---|
| 分子量 | 理論値: 800 KDa |
-分子 #1: RNA Polyermase II core
| 分子 | 名称: RNA Polyermase II core / タイプ: protein_or_peptide / ID: 1 / Name.synonym: Pol2 / コピー数: 1 / 組換発現: No / データベース: NCBI |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) / 別称: human / 組織: breast / 細胞: tumor / Organelle: nucleus / 細胞中の位置: nucleus Homo sapiens (ヒト) / 別称: human / 組織: breast / 細胞: tumor / Organelle: nucleus / 細胞中の位置: nucleus |
| 分子量 | 理論値: 500 KDa |
| 配列 | UniProtKB:  DNA-directed RNA polymerase II subunit RPB1 DNA-directed RNA polymerase II subunit RPB1 |
-分子 #2: BRCA1
| 分子 | 名称: BRCA1 / タイプ: protein_or_peptide / ID: 2 詳細: BRCA1 was attached to tunable microchips using antibodies against the BRCA1 C-terminal domain コピー数: 1 / 集合状態: monomer / 組換発現: No / データベース: NCBI |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) / 別称: human / 組織: breast / 細胞: tumor / Organelle: nucleus / 細胞中の位置: nucleus Homo sapiens (ヒト) / 別称: human / 組織: breast / 細胞: tumor / Organelle: nucleus / 細胞中の位置: nucleus |
| 分子量 | 理論値: 200 KDa |
| 配列 | UniProtKB: Breast cancer type 1 susceptibility protein |
-分子 #3: BARD1
| 分子 | 名称: BARD1 / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 集合状態: monomer / 組換発現: No / データベース: NCBI |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) / 別称: human / 組織: breast / 細胞: tumor / Organelle: nucleus / 細胞中の位置: nucleus Homo sapiens (ヒト) / 別称: human / 組織: breast / 細胞: tumor / Organelle: nucleus / 細胞中の位置: nucleus |
| 分子量 | 理論値: 87 KDa |
| 配列 | UniProtKB: BRCA1-associated RING domain protein 1 |
-分子 #4: Ubiqutin
| 分子 | 名称: Ubiqutin / タイプ: protein_or_peptide / ID: 4 / コピー数: 2 / 集合状態: monomer / 組換発現: No / データベース: NCBI |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) / 別称: human / 組織: breast / 細胞: tumor / Organelle: nucleus / 細胞中の位置: nucleus Homo sapiens (ヒト) / 別称: human / 組織: breast / 細胞: tumor / Organelle: nucleus / 細胞中の位置: nucleus |
| 分子量 | 理論値: 8 KDa |
| 配列 | UniProtKB: Polyubiquitin-C |
-分子 #5: DNA
| 分子 | 名称: DNA / タイプ: dna / ID: 5 / 分類: DNA / Structure: DOUBLE HELIX / Synthetic?: No |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) / 別称: human Homo sapiens (ヒト) / 別称: human |
-実験情報
-構造解析
| 手法 |  ネガティブ染色法, ネガティブ染色法,  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 / 詳細: 50 mM HEPES, 150 mM NaCl, 10 mM MgCl2, 10 mM CaCl2 |
|---|---|
| 染色 | タイプ: NEGATIVE 詳細: Protein complexes were adsorbed to antibody-decorated microchips for 2 minutes and stained with 1% uranyl formate for 1 minute. |
| グリッド | 詳細: SiN microchips with TEM windows coated with 25% Ni-NTA functionalized lipid monolayers |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 90 % / チャンバー内温度: 93 K / 装置: GATAN CRYOPLUNGE 3 手法: Microchips were blotted for ~8 seconds using one-sided blotting. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI SPIRIT |
|---|---|
| 電子線 | 加速電圧: 120 kV / 電子線源: LAB6 |
| 電子光学系 | 倍率(補正後): 54500 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2 mm / 最大 デフォーカス(公称値): -3.0 µm / 最小 デフォーカス(公称値): -1.0 µm / 倍率(公称値): 50000 Bright-field microscopy / Cs: 2 mm / 最大 デフォーカス(公称値): -3.0 µm / 最小 デフォーカス(公称値): -1.0 µm / 倍率(公称値): 50000 |
| 試料ステージ | 試料ホルダーモデル: GATAN LIQUID NITROGEN |
| 温度 | 最低: 83 K |
| アライメント法 | Legacy - 非点収差: Objective lens astigmatism was corrected at high magnification. |
| 詳細 | low-dose illumination |
| 日付 | 2013年3月8日 |
| 撮影 | カテゴリ: CCD / フィルム・検出器のモデル: FEI EAGLE (2k x 2k) / デジタル化 - サンプリング間隔: 30 µm / 実像数: 50 / 平均電子線量: 5 e/Å2 |
| 実験機器 |  モデル: Tecnai Spirit / 画像提供: FEI Company |
- 画像解析
画像解析
| CTF補正 | 詳細: Each particle |
|---|---|
| 最終 2次元分類 | クラス数: 5 |
| 最終 再構成 | アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 22.0 Å / 解像度の算出法: OTHER / ソフトウェア - 名称: RELION / 使用した粒子像数: 22000 |
| 詳細 | The particles were selected using an automatic selection program. |
-原子モデル構築 1
| 初期モデル | PDB ID: Chain - #0 - Chain ID: A / Chain - #1 - Chain ID: B / Chain - #2 - Chain ID: C / Chain - #3 - Chain ID: D / Chain - #4 - Chain ID: E / Chain - #5 - Chain ID: F / Chain - #6 - Chain ID: G / Chain - #7 - Chain ID: H / Chain - #8 - Chain ID: I / Chain - #9 - Chain ID: J / Chain - #10 - Chain ID: K / Chain - #11 - Chain ID: L |
|---|---|
| ソフトウェア | 名称:  Chimera Chimera |
| 詳細 | The domains were separately fitted by manual docking using the Chimera software package. |
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT |
-原子モデル構築 2
| 初期モデル | PDB ID: Chain - Chain ID: A |
|---|---|
| ソフトウェア | 名称:  Chimera Chimera |
| 詳細 | The domains were separately fitted by manual docking using the Chimera software package. |
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT |
-原子モデル構築 3
| 初期モデル | PDB ID: Chain - Chain ID: A |
|---|---|
| ソフトウェア | 名称:  Chimera Chimera |
| 詳細 | The domains were separately fitted by manual docking using the Chimera software package. |
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT |
-原子モデル構築 4
| 初期モデル | PDB ID: Chain - #0 - Chain ID: A / Chain - #1 - Chain ID: B |
|---|---|
| ソフトウェア | 名称:  Chimera Chimera |
| 詳細 | The domains were separately fitted by manual docking using the Chimera software package. |
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT |
 ムービー
ムービー コントローラー
コントローラー