+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-6178 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | CryoEM map of Mycobacterium tuberculosis 50S ribosome | |||||||||
 マップデータ マップデータ | Mycobacterium tuberculosis 50S ribosome | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード |  Mycobacterium tuberculosis (結核菌) / Mycobacterium tuberculosis (結核菌) /  ribosome (リボソーム) / 50S ribosome (リボソーム) / 50S | |||||||||
| 生物種 |   Mycobacterium tuberculosis (結核菌) Mycobacterium tuberculosis (結核菌) | |||||||||
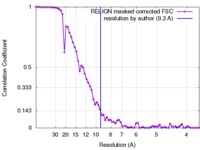
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 9.3 Å クライオ電子顕微鏡法 / 解像度: 9.3 Å | |||||||||
 データ登録者 データ登録者 | Yang K / Zhang J | |||||||||
 引用 引用 |  ジャーナル: Structure / 年: 2015 ジャーナル: Structure / 年: 2015タイトル: Structure of Ribosomal Silencing Factor Bound to Mycobacterium tuberculosis Ribosome. 著者: Xiaojun Li / Qingan Sun / Cai Jiang / Kailu Yang / Li-Wei Hung / Junjie Zhang / James C Sacchettini /  要旨: The ribosomal silencing factor RsfS slows cell growth by inhibiting protein synthesis during periods of diminished nutrient availability. The crystal structure of Mycobacterium tuberculosis (Mtb) ...The ribosomal silencing factor RsfS slows cell growth by inhibiting protein synthesis during periods of diminished nutrient availability. The crystal structure of Mycobacterium tuberculosis (Mtb) RsfS, together with the cryo-electron microscopy (EM) structure of the large subunit 50S of Mtb ribosome, reveals how inhibition of protein synthesis by RsfS occurs. RsfS binds to the 50S at L14, which, when occupied, blocks the association of the small subunit 30S. Although Mtb RsfS is a dimer in solution, only a single subunit binds to 50S. The overlap between the dimer interface and the L14 binding interface confirms that the RsfS dimer must first dissociate to a monomer in order to bind to L14. RsfS interacts primarily through electrostatic and hydrogen bonding to L14. The EM structure shows extended rRNA density that it is not found in the Escherichia coli ribosome, the most striking of these being the extended RNA helix of H54a. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_6178.map.gz emd_6178.map.gz | 8.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-6178-v30.xml emd-6178-v30.xml emd-6178.xml emd-6178.xml | 9.1 KB 9.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_6178_fsc.xml emd_6178_fsc.xml | 7.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  400_6178.gif 400_6178.gif 80_6178.gif 80_6178.gif | 55.7 KB 4.1 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-6178 http://ftp.pdbj.org/pub/emdb/structures/EMD-6178 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-6178 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-6178 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_6178.map.gz / 形式: CCP4 / 大きさ: 26.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_6178.map.gz / 形式: CCP4 / 大きさ: 26.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Mycobacterium tuberculosis 50S ribosome | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.85 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Mycobacterium tuberculosis ribosome 50S
| 全体 | 名称: Mycobacterium tuberculosis ribosome 50S |
|---|---|
| 要素 |
|
-超分子 #1000: Mycobacterium tuberculosis ribosome 50S
| 超分子 | 名称: Mycobacterium tuberculosis ribosome 50S / タイプ: sample / ID: 1000 / 詳細: The sample was monodisperse / 集合状態: 1 / Number unique components: 1 |
|---|---|
| 分子量 | 実験値: 1.6 MDa / 理論値: 1.6 MDa |
-超分子 #1: 50S ribosome
| 超分子 | 名称: 50S ribosome / タイプ: complex / ID: 1 / 組換発現: No / データベース: NCBI Ribosome-details: ribosome-prokaryote: LSU 50S, LSU RNA 23S, LSU RNA 5S |
|---|---|
| 由来(天然) | 生物種:   Mycobacterium tuberculosis (結核菌) / 株: MC_2 7000 Mycobacterium tuberculosis (結核菌) / 株: MC_2 7000 |
| 分子量 | 理論値: 1.6 MDa |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.6 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 詳細: 5 mM HEPES sodium, pH 7.5, 10 mM NH4Cl, 50 mM KCl, 10 mM MgCl2 |
| グリッド | 詳細: 200 mesh R2/2 Quantifoil grid, glow-discharged |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / 装置: FEI VITROBOT MARK I |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI F20 |
|---|---|
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 81081 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 62000 Bright-field microscopy / Cs: 2 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 62000 |
| 試料ステージ | 試料ホルダー: 626 holder / 試料ホルダーモデル: GATAN LIQUID NITROGEN |
| 温度 | 平均: 100 K |
| 日付 | 2013年8月6日 |
| 撮影 | カテゴリ: CCD フィルム・検出器のモデル: GATAN ULTRASCAN 4000 (4k x 4k) 実像数: 165 / 平均電子線量: 20 e/Å2 |
| 実験機器 |  モデル: Tecnai F20 / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー