+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of the human MRS2 magnesium channel under Mg2+ condition | |||||||||
 マップデータ マップデータ | local resolution filtered map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード |  Magnesium (マグネシウム) / Magnesium (マグネシウム) /  Ion channel (イオンチャネル) / Ion channel (イオンチャネル) /  Membrane protein (膜タンパク質) / METAL TRANSPORT / Ion Translocation / Divalent Ion / Membrane protein (膜タンパク質) / METAL TRANSPORT / Ion Translocation / Divalent Ion /  Pentamer Pentamer | |||||||||
| 機能・相同性 | mitochondrial magnesium ion transmembrane transport / Magnesium transporter MRS2-like / Miscellaneous transport and binding events / magnesium ion transmembrane transporter activity / lactate metabolic process / transmembrane transport /  ミトコンドリア内膜 / ミトコンドリア内膜 /  ミトコンドリア / Magnesium transporter MRS2 homolog, mitochondrial ミトコンドリア / Magnesium transporter MRS2 homolog, mitochondrial 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 2.8 Å クライオ電子顕微鏡法 / 解像度: 2.8 Å | |||||||||
 データ登録者 データ登録者 | Lai LTF / Balaraman J / Zhou F / Matthies D | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2023 ジャーナル: Nat Commun / 年: 2023タイトル: Cryo-EM structures of human magnesium channel MRS2 reveal gating and regulatory mechanisms. 著者: Louis Tung Faat Lai / Jayashree Balaraman / Fei Zhou / Doreen Matthies /  要旨: Magnesium ions (Mg) play an essential role in cellular physiology. In mitochondria, protein and ATP synthesis and various metabolic pathways are directly regulated by Mg. MRS2, a magnesium channel ...Magnesium ions (Mg) play an essential role in cellular physiology. In mitochondria, protein and ATP synthesis and various metabolic pathways are directly regulated by Mg. MRS2, a magnesium channel located in the inner mitochondrial membrane, mediates the influx of Mg into the mitochondrial matrix and regulates Mg homeostasis. Knockdown of MRS2 in human cells leads to reduced uptake of Mg into mitochondria and disruption of the mitochondrial metabolism. Despite the importance of MRS2, the Mg translocation and regulation mechanisms of MRS2 are still unclear. Here, using cryo-EM we report the structures of human MRS2 in the presence and absence of Mg at 2.8 Å and 3.3 Å, respectively. From the homo-pentameric structures, we identify R332 and M336 as major gating residues, which are then tested using mutagenesis and two cellular divalent ion uptake assays. A network of hydrogen bonds is found connecting the gating residue R332 to the soluble domain, potentially regulating the gate. Two Mg-binding sites are identified in the MRS2 soluble domain, distinct from the two sites previously reported in CorA, a homolog of MRS2 in prokaryotes. Altogether, this study provides the molecular basis for understanding the Mg translocation and regulatory mechanisms of MRS2. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_41624.map.gz emd_41624.map.gz | 4.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-41624-v30.xml emd-41624-v30.xml emd-41624.xml emd-41624.xml | 29 KB 29 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_41624_fsc.xml emd_41624_fsc.xml | 10.5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_41624.png emd_41624.png | 83.4 KB | ||
| マスクデータ |  emd_41624_msk_1.map emd_41624_msk_1.map | 125 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-41624.cif.gz emd-41624.cif.gz | 7.9 KB | ||
| その他 |  emd_41624_additional_1.map.gz emd_41624_additional_1.map.gz emd_41624_additional_2.map.gz emd_41624_additional_2.map.gz emd_41624_half_map_1.map.gz emd_41624_half_map_1.map.gz emd_41624_half_map_2.map.gz emd_41624_half_map_2.map.gz | 62 MB 117.8 MB 116.1 MB 116.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-41624 http://ftp.pdbj.org/pub/emdb/structures/EMD-41624 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-41624 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-41624 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_41624.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_41624.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | local resolution filtered map | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.83 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_41624_msk_1.map emd_41624_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
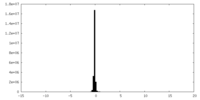
| 密度ヒストグラム |
-追加マップ: unsharpened map
| ファイル | emd_41624_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | unsharpened map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
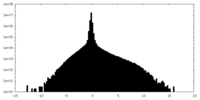
| 密度ヒストグラム |
-追加マップ: sharpened map
| ファイル | emd_41624_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | sharpened map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half map A
| ファイル | emd_41624_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map A | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
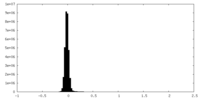
| 密度ヒストグラム |
-ハーフマップ: half map B
| ファイル | emd_41624_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map B | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
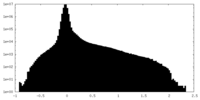
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Cryo-EM structure of the pentameric human MRS2 magnesium channel ...
| 全体 | 名称: Cryo-EM structure of the pentameric human MRS2 magnesium channel under Mg2+ condition at an average resolution of 2.8 A, filtered to local resolution, C5 |
|---|---|
| 要素 |
|
-超分子 #1: Cryo-EM structure of the pentameric human MRS2 magnesium channel ...
| 超分子 | 名称: Cryo-EM structure of the pentameric human MRS2 magnesium channel under Mg2+ condition at an average resolution of 2.8 A, filtered to local resolution, C5 タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 219 KDa |
-分子 #1: Magnesium transporter MRS2 homolog, mitochondrial
| 分子 | 名称: Magnesium transporter MRS2 homolog, mitochondrial / タイプ: protein_or_peptide / ID: 1 / コピー数: 5 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 51.373516 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MECLRSLPCL LPRAMRLPRR TLCALALDVT SVGPPVAACG RRANLIGRSR AAQLCGPDRL RVAGEVHRFR TSDVSQATLA SVAPVFTVT KFDKQGNVTS FERKKTELYQ ELGLQARDLR FQHVMSITVR NNRIIMRMEY LKAVITPECL LILDYRNLNL E QWLFRELP ...文字列: MECLRSLPCL LPRAMRLPRR TLCALALDVT SVGPPVAACG RRANLIGRSR AAQLCGPDRL RVAGEVHRFR TSDVSQATLA SVAPVFTVT KFDKQGNVTS FERKKTELYQ ELGLQARDLR FQHVMSITVR NNRIIMRMEY LKAVITPECL LILDYRNLNL E QWLFRELP SQLSGEGQLV TYPLPFEFRA IEALLQYWIN TLQGKLSILQ PLILETLDAL VDPKHSSVDR SKLHILLQNG KS LSELETD IKIFKESILE ILDEEELLEE LCVSKWSDPQ VFEKSSAGID HAEEMELLLE NYYRLADDLS NAARELRVLI DDS QSIIFI NLDSHRNVMM RLNLQLTMGT FSLSLFGLMG VAFGMNLESS LEEDHRIFWL ITGIMFMGSG LIWRRLLSFL GRQL EAPLP PMMASLPKKT LLADRSMELK NSLRLDGLGS GRSILTNRDY KDDDDK UniProtKB: Magnesium transporter MRS2 homolog, mitochondrial |
-分子 #2: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 2 / コピー数: 14 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-分子 #3: water
| 分子 | 名称: water / タイプ: ligand / ID: 3 / コピー数: 281 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.5 mg/mL | ||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.3 構成要素:
詳細: 20 mM HEPES, 150 mM NaCl, 40 mM MgCl2, 0.003% LMNG, pH 7.3 | ||||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 400 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 60 sec. / 前処理 - 雰囲気: AIR | ||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / チャンバー内温度: 277 K / 装置: LEICA EM GP 詳細: 400-mesh R1.2/1.3 Cu grids (Quantifoil) were made hydrophilic by glow discharging for 60 seconds with a current of 15 mA in a PELCO easiGlow system. The cryo grids were produced using a Leica ...詳細: 400-mesh R1.2/1.3 Cu grids (Quantifoil) were made hydrophilic by glow discharging for 60 seconds with a current of 15 mA in a PELCO easiGlow system. The cryo grids were produced using a Leica EM GP2 (Leica). The chamber was kept at 4 C and set to 95% humidity. 3 microliter sample at 0.5 mg/ml was applied to a glow-discharged holey grid, blotted for 6 s, and plunge frozen into liquid ethane and stored in liquid nitrogen.. | ||||||||||
| 詳細 | This sample was monodisperse |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.0 µm Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.0 µm最小 デフォーカス(公称値): 0.7000000000000001 µm 倍率(公称値): 105000 |
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV 詳細: Cryo-EM datasets were acquired with SerialEM using a Titan Krios (FEI, now ThermoFisher Scientific) operated at 300 keV and equipped with an energy filter and K3 camera (Gatan Inc.). Movies ...詳細: Cryo-EM datasets were acquired with SerialEM using a Titan Krios (FEI, now ThermoFisher Scientific) operated at 300 keV and equipped with an energy filter and K3 camera (Gatan Inc.). Movies of 50 frames with a dose of 1 e-/A2 per frame (50 e-/A2 total dose) were recorded at a nominal magnification of 105,000x, corresponding to a physical pixel size of 0.83 A/px (super-resolution pixel size 0.415 A/px) in CDS mode at a dose rate of 10 e-/px/s and a defocus range of -0.7 to -2.0 um. In total, 3,991 and 9,656 movies were collected. |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 撮影したグリッド数: 1 / 実像数: 3991 / 平均露光時間: 3.462 sec. / 平均電子線量: 50.0 e/Å2 詳細: Cryo-EM datasets were acquired with SerialEM using a Titan Krios (FEI, now ThermoFisher Scientific) operated at 300 keV and equipped with an energy filter and K3 camera (Gatan Inc.). Movies ...詳細: Cryo-EM datasets were acquired with SerialEM using a Titan Krios (FEI, now ThermoFisher Scientific) operated at 300 keV and equipped with an energy filter and K3 camera (Gatan Inc.). Movies of 50 frames with a dose of 1 e-/A2 per frame (50 e-/A2 total dose) were recorded at a nominal magnification of 105,000x, corresponding to a physical pixel size of 0.83 A/px (super-resolution pixel size 0.415 A/px) in CDS mode at a dose rate of 10 e-/px/s and a defocus range of -0.7 to -2.0 um. In total, 3,991 and 9,656 movies were collected. |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | PDB ID: Chain - Source name: AlphaFold / Chain - Initial model type: in silico model |
|---|---|
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT / 温度因子: 141 |
| 得られたモデル |  PDB-8tul: |
-原子モデル構築 2
| 初期モデル | PDB ID: Chain - Source name: AlphaFold / Chain - Initial model type: in silico model |
|---|---|
| 詳細 | The model was then manually rebuilt in COOT v.0.9.7 using the local resolution filtered map, which was generated from refinement with C5 applied. The Mg2+ ions assigned in the pore regions were confirmed in the C1 map. Loop regions (residues 174-181, 273-287) were built with the unsharpened map. Iterative rounds of manual refinement in COOT and real-space refinement in Phenix v.1.20.1-4487 were performed. |
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT / 温度因子: 141 |
| 得られたモデル |  PDB-8tul: |
 ムービー
ムービー コントローラー
コントローラー









 Z
Z Y
Y X
X