+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | CryoEM map of the locally refined interfaces-1,2,3 of soluble OPA1 from the apo bound helical assembly on a lipid membrane | |||||||||
 マップデータ マップデータ | CryoEM map of the locally refined interfaces-1,2,3 of soluble OPA1 from the apo bound helical assembly on a lipid membrane | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード |  GTPase (GTPアーゼ) / Dynamin-family protein / GTPase (GTPアーゼ) / Dynamin-family protein /  mitochondrial fusion protein / mitochondrial fusion protein /  mitochondria (ミトコンドリア) / mitochondria (ミトコンドリア) /  Optic Atrophy / LIPID BINDING PROTEIN Optic Atrophy / LIPID BINDING PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 Regulation of Apoptosis / membrane tubulation / inner mitochondrial membrane organization / Regulation of Apoptosis / membrane tubulation / inner mitochondrial membrane organization /  dynamin GTPase / dynamin GTPase /  cardiolipin binding / cardiolipin binding /  mitochondrial genome maintenance / mitochondrial genome maintenance /  phosphatidic acid binding / phosphatidic acid binding /  mitochondrial fission / GTP metabolic process / mitochondrial fission / GTP metabolic process /  mitochondrial fusion ... mitochondrial fusion ... Regulation of Apoptosis / membrane tubulation / inner mitochondrial membrane organization / Regulation of Apoptosis / membrane tubulation / inner mitochondrial membrane organization /  dynamin GTPase / dynamin GTPase /  cardiolipin binding / cardiolipin binding /  mitochondrial genome maintenance / mitochondrial genome maintenance /  phosphatidic acid binding / phosphatidic acid binding /  mitochondrial fission / GTP metabolic process / mitochondrial fission / GTP metabolic process /  mitochondrial fusion / axonal transport of mitochondrion / negative regulation of release of cytochrome c from mitochondria / mitochondrial crista / negative regulation of endoplasmic reticulum stress-induced intrinsic apoptotic signaling pathway / axon cytoplasm / mitochondrial fusion / axonal transport of mitochondrion / negative regulation of release of cytochrome c from mitochondria / mitochondrial crista / negative regulation of endoplasmic reticulum stress-induced intrinsic apoptotic signaling pathway / axon cytoplasm /  視覚 / mitochondrion organization / neural tube closure / 視覚 / mitochondrion organization / neural tube closure /  ミトコンドリア / ミトコンドリア /  ミトコンドリア / ミトコンドリア /  細胞老化 / protein complex oligomerization / 細胞老化 / protein complex oligomerization /  microtubule binding / microtubule binding /  ミトコンドリア内膜 / mitochondrial outer membrane / ミトコンドリア内膜 / mitochondrial outer membrane /  微小管 / 微小管 /  GTPase activity / GTPase activity /  樹状突起 / apoptotic process / GTP binding / negative regulation of apoptotic process / magnesium ion binding / 樹状突起 / apoptotic process / GTP binding / negative regulation of apoptotic process / magnesium ion binding /  ミトコンドリア / ミトコンドリア /  核質 / 核質 /  生体膜 / 生体膜 /  細胞質基質 / 細胞質基質 /  細胞質 細胞質類似検索 - 分子機能 | |||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | らせん対称体再構成法 /  クライオ電子顕微鏡法 / 解像度: 5.51 Å クライオ電子顕微鏡法 / 解像度: 5.51 Å | |||||||||
 データ登録者 データ登録者 | Nyenhuis SB / Wu X / Strub MP / Yim YI / Stanton AE / Baena V / Syed ZA / Canagarajah B / Hammer JA / Hinshaw JE | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2023 ジャーナル: Nature / 年: 2023タイトル: OPA1 helical structures give perspective to mitochondrial dysfunction. 著者: Sarah B Nyenhuis / Xufeng Wu / Marie-Paule Strub / Yang-In Yim / Abigail E Stanton / Valentina Baena / Zulfeqhar A Syed / Bertram Canagarajah / John A Hammer / Jenny E Hinshaw /  要旨: Dominant optic atrophy is one of the leading causes of childhood blindness. Around 60-80% of cases are caused by mutations of the gene that encodes optic atrophy protein 1 (OPA1), a protein that has ...Dominant optic atrophy is one of the leading causes of childhood blindness. Around 60-80% of cases are caused by mutations of the gene that encodes optic atrophy protein 1 (OPA1), a protein that has a key role in inner mitochondrial membrane fusion and remodelling of cristae and is crucial for the dynamic organization and regulation of mitochondria. Mutations in OPA1 result in the dysregulation of the GTPase-mediated fusion process of the mitochondrial inner and outer membranes. Here we used cryo-electron microscopy methods to solve helical structures of OPA1 assembled on lipid membrane tubes, in the presence and absence of nucleotide. These helical assemblies organize into densely packed protein rungs with minimal inter-rung connectivity, and exhibit nucleotide-dependent dimerization of the GTPase domains-a hallmark of the dynamin superfamily of proteins. OPA1 also contains several unique secondary structures in the paddle domain that strengthen its membrane association, including membrane-inserting helices. The structural features identified in this study shed light on the effects of pathogenic point mutations on protein folding, inter-protein assembly and membrane interactions. Furthermore, mutations that disrupt the assembly interfaces and membrane binding of OPA1 cause mitochondrial fragmentation in cell-based assays, providing evidence of the biological relevance of these interactions. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_40204.map.gz emd_40204.map.gz | 208.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-40204-v30.xml emd-40204-v30.xml emd-40204.xml emd-40204.xml | 20.5 KB 20.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_40204_fsc.xml emd_40204_fsc.xml | 16 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_40204.png emd_40204.png | 62.6 KB | ||
| マスクデータ |  emd_40204_msk_1.map emd_40204_msk_1.map | 421.9 MB |  マスクマップ マスクマップ | |
| その他 |  emd_40204_additional_1.map.gz emd_40204_additional_1.map.gz emd_40204_additional_2.map.gz emd_40204_additional_2.map.gz emd_40204_half_map_1.map.gz emd_40204_half_map_1.map.gz emd_40204_half_map_2.map.gz emd_40204_half_map_2.map.gz | 352.2 MB 398.3 MB 391 MB 391 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-40204 http://ftp.pdbj.org/pub/emdb/structures/EMD-40204 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-40204 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-40204 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_40204.map.gz / 形式: CCP4 / 大きさ: 421.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_40204.map.gz / 形式: CCP4 / 大きさ: 421.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | CryoEM map of the locally refined interfaces-1,2,3 of soluble OPA1 from the apo bound helical assembly on a lipid membrane | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.2518 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_40204_msk_1.map emd_40204_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
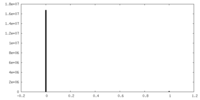
| 投影像・断面図 |
| ||||||||||||
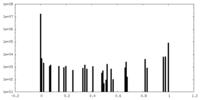
| 密度ヒストグラム |
-追加マップ: CryoEM sharpened map 1 of the locally refined...
| ファイル | emd_40204_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | CryoEM sharpened map 1 of the locally refined interfaces-1,2,3 of soluble OPA1 from the apo bound helical assembly on a lipid membrane | ||||||||||||
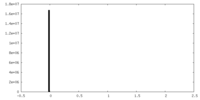
| 投影像・断面図 |
| ||||||||||||
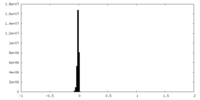
| 密度ヒストグラム |
-追加マップ: CryoEM sharpened map 2 of the locally refined...
| ファイル | emd_40204_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | CryoEM sharpened map 2 of the locally refined interfaces-1,2,3 of soluble OPA1 from the apo bound helical assembly on a lipid membrane | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: CryoEM half map A of the locally refined...
| ファイル | emd_40204_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | CryoEM half map A of the locally refined interfaces-1,2,3 of soluble OPA1 from the apo bound helical assembly on a lipid membrane | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: CryoEM half map B of the locally refined...
| ファイル | emd_40204_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | CryoEM half map B of the locally refined interfaces-1,2,3 of soluble OPA1 from the apo bound helical assembly on a lipid membrane | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : CryoEM map of the locally refined soluble OPA1 Z-clip from the ap...
| 全体 | 名称: CryoEM map of the locally refined soluble OPA1 Z-clip from the apo helical assembly on a lipid membrane |
|---|---|
| 要素 |
|
-超分子 #1: CryoEM map of the locally refined soluble OPA1 Z-clip from the ap...
| 超分子 | 名称: CryoEM map of the locally refined soluble OPA1 Z-clip from the apo helical assembly on a lipid membrane タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: sOPA1 protein
| 分子 | 名称: sOPA1 protein / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:   Escherichia coli BL21(DE3) (大腸菌) Escherichia coli BL21(DE3) (大腸菌) |
| 配列 | 文字列: ATDRGSESDK HFRKVSDKEK IDQLQEELLH TQLKYQRILE RLEKENKELR KLVLQKDDKG IHHRKLKKSL IDMYSEVLD VLSDYDASYN TQDHLPRVVV VGDQSAGKTS VLEMIAQARI FPRGSGEMMT RSPVKVTLSE G PHHVALFK DSSREFDLTK EEDLAALRHE ...文字列: ATDRGSESDK HFRKVSDKEK IDQLQEELLH TQLKYQRILE RLEKENKELR KLVLQKDDKG IHHRKLKKSL IDMYSEVLD VLSDYDASYN TQDHLPRVVV VGDQSAGKTS VLEMIAQARI FPRGSGEMMT RSPVKVTLSE G PHHVALFK DSSREFDLTK EEDLAALRHE IELRMRKNVK EGCTVSPETI SLNVKGPGLQ RMVLVDLPGV IN TVTSGMA PDTKETIFSI SKAYMQNPNA IILCIQDGSV DAERSIVTDL VSQMDPHGRR TIFVLTKVDL AEK NVASPS RIQQIIEGKL FPMKALGYFA VVTGKGNSSE SIEAIREYEE EFFQNSKLLK TSMLKAHQVT TRNL SLAVS DCFWKMVRES VEQQADSFKA TRFNLETEWK NNYPRLRELD RNELFEKAKN EILDEVISLS QVTPK HWEE ILQQSLWERV STHVIENIYL PAAQTMNSGT FNTTVDIKLK QWTDKQLPNK AVEVAWETLQ EEFSRF MTE PKGKEHDDIF DKLKEAVKEE SIKRHKWNDF AEDSLRVIQH NALEDRSISD KQQWDAAIYF MEEALQA RL KDTENAIENM VGPDWKKRWL YWKNRTQEQC VHNETKNELE KMLKCNEEHP AYLASDEITT VRKNLESR G VEVDPSLIKD TWHQVYRRHF LKTALNHCNL CRRGFYYYQR HFVDSELECN DVVLFWRIQR MLAITANTL RQQLTNTEVR RLEKNVKEVL EDFAEDGEKK IKLLTGKRVQ LAEDLKKVRE IQEKLDAFIE ALHQEK UniProtKB: Dynamin-like 120 kDa protein, mitochondrial |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | helical array |
- 試料調製
試料調製
| 緩衝液 | pH: 7.2 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS GLACIOS |
|---|---|
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 2.4 µm / 最小 デフォーカス(公称値): 0.6 µm Bright-field microscopy / 最大 デフォーカス(公称値): 2.4 µm / 最小 デフォーカス(公称値): 0.6 µm |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 24.86 e/Å2 |
- 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT |
|---|
 ムービー
ムービー コントローラー
コントローラー



























 Z
Z Y
Y X
X