+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cation channelrhodopsin from Hyphochytrium catenoides (HcCCR) embedded in peptidisc | ||||||||||||
 マップデータ マップデータ | |||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード |  Retinal Protein (レチナール) / Retinal Protein (レチナール) /  Channelrhodopsin / Channelrhodopsin /  Cation channel (イオンチャネル) / Peptidisc / Cation channel (イオンチャネル) / Peptidisc /  Optogenetics (光遺伝学) / Optogenetics (光遺伝学) /  TRANSPORT PROTEIN (運搬体タンパク質) TRANSPORT PROTEIN (運搬体タンパク質) | ||||||||||||
| 生物種 |  Hyphochytrium catenoides (真核生物) Hyphochytrium catenoides (真核生物) | ||||||||||||
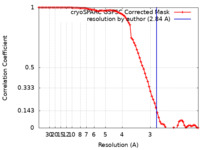
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 2.84 Å クライオ電子顕微鏡法 / 解像度: 2.84 Å | ||||||||||||
 データ登録者 データ登録者 | Morizumi T / Kim K / Li H / Spudich JL / Ernst OP | ||||||||||||
| 資金援助 |  カナダ, 3件 カナダ, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2023 ジャーナル: Nat Commun / 年: 2023タイトル: Structures of channelrhodopsin paralogs in peptidiscs explain their contrasting K and Na selectivities. 著者: Takefumi Morizumi / Kyumhyuk Kim / Hai Li / Elena G Govorunova / Oleg A Sineshchekov / Yumei Wang / Lei Zheng / Éva Bertalan / Ana-Nicoleta Bondar / Azam Askari / Leonid S Brown / John L ...著者: Takefumi Morizumi / Kyumhyuk Kim / Hai Li / Elena G Govorunova / Oleg A Sineshchekov / Yumei Wang / Lei Zheng / Éva Bertalan / Ana-Nicoleta Bondar / Azam Askari / Leonid S Brown / John L Spudich / Oliver P Ernst /     要旨: Kalium channelrhodopsin 1 from Hyphochytrium catenoides (HcKCR1) is a light-gated channel used for optogenetic silencing of mammalian neurons. It selects K over Na in the absence of the canonical ...Kalium channelrhodopsin 1 from Hyphochytrium catenoides (HcKCR1) is a light-gated channel used for optogenetic silencing of mammalian neurons. It selects K over Na in the absence of the canonical tetrameric K selectivity filter found universally in voltage- and ligand-gated channels. The genome of H. catenoides also encodes a highly homologous cation channelrhodopsin (HcCCR), a Na channel with >100-fold larger Na to K permeability ratio. Here, we use cryo-electron microscopy to determine atomic structures of these two channels embedded in peptidiscs to elucidate structural foundations of their dramatically different cation selectivity. Together with structure-guided mutagenesis, we show that K versus Na selectivity is determined at two distinct sites on the putative ion conduction pathway: in a patch of critical residues in the intracellular segment (Leu69/Phe69, Ile73/Ser73 and Asp116) and within a cluster of aromatic residues in the extracellular segment (primarily, Trp102 and Tyr222). The two filters are on the opposite sides of the photoactive site involved in channel gating. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_40063.map.gz emd_40063.map.gz | 49.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-40063-v30.xml emd-40063-v30.xml emd-40063.xml emd-40063.xml | 21 KB 21 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_40063_fsc.xml emd_40063_fsc.xml | 7.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_40063.png emd_40063.png | 54.6 KB | ||
| Filedesc metadata |  emd-40063.cif.gz emd-40063.cif.gz | 6.7 KB | ||
| その他 |  emd_40063_half_map_1.map.gz emd_40063_half_map_1.map.gz emd_40063_half_map_2.map.gz emd_40063_half_map_2.map.gz | 48.9 MB 48.9 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-40063 http://ftp.pdbj.org/pub/emdb/structures/EMD-40063 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-40063 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-40063 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_40063.map.gz / 形式: CCP4 / 大きさ: 52.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_40063.map.gz / 形式: CCP4 / 大きさ: 52.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.03 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_40063_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
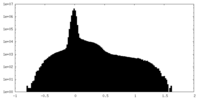
| 投影像・断面図 |
| ||||||||||||
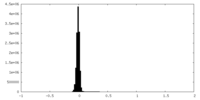
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_40063_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
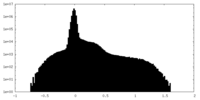
| 投影像・断面図 |
| ||||||||||||
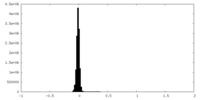
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Cation channelrhodopsin trimer reconstituted in peptidisc
| 全体 | 名称: Cation channelrhodopsin trimer reconstituted in peptidisc |
|---|---|
| 要素 |
|
-超分子 #1: Cation channelrhodopsin trimer reconstituted in peptidisc
| 超分子 | 名称: Cation channelrhodopsin trimer reconstituted in peptidisc タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Hyphochytrium catenoides (真核生物) Hyphochytrium catenoides (真核生物) |
| 分子量 | 理論値: 30.197 KDa |
-分子 #1: Cation Channelrhodopsin
| 分子 | 名称: Cation Channelrhodopsin / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Hyphochytrium catenoides (真核生物) Hyphochytrium catenoides (真核生物) |
| 分子量 | 理論値: 30.224213 KDa |
| 組換発現 | 生物種:   Komagataella pastoris (菌類) Komagataella pastoris (菌類) |
| 配列 | 文字列: MPFCGGRPED GWHHGSIHDM DYPLLGAMAA ICSVFIGGSG AWMLYRLDLG LGYSCKPHHS GYAPEANSFS ALSCLVSGTI YAAKTFDFF DGGGTPFSFN WYWYLDYVFT CPLILLDVLY TLEIPHKLRF VFAVIITLWC GVAAFVTPSA FRFGYYAVGC V WFVPFSFS ...文字列: MPFCGGRPED GWHHGSIHDM DYPLLGAMAA ICSVFIGGSG AWMLYRLDLG LGYSCKPHHS GYAPEANSFS ALSCLVSGTI YAAKTFDFF DGGGTPFSFN WYWYLDYVFT CPLILLDVLY TLEIPHKLRF VFAVIITLWC GVAAFVTPSA FRFGYYAVGC V WFVPFSFS LLRHVKQRYQ VYPPKCQKLL FWACTIFFGF WPLFPILFLF SWLGTGHIDQ QAFTIIHAFL DLFCKTVFGL IM TFFRLEL EEHTEVLGLP LNEPKGKH |
-分子 #2: RETINAL
| 分子 | 名称: RETINAL / タイプ: ligand / ID: 2 / コピー数: 1 / 式: RET |
|---|---|
| 分子量 | 理論値: 284.436 Da |
| Chemical component information |  ChemComp-RET: |
-分子 #3: SODIUM ION
| 分子 | 名称: SODIUM ION / タイプ: ligand / ID: 3 / コピー数: 3 |
|---|---|
| 分子量 | 理論値: 22.99 Da |
-分子 #4: CHOLESTEROL
| 分子 | 名称: CHOLESTEROL / タイプ: ligand / ID: 4 / コピー数: 7 / 式: CLR |
|---|---|
| 分子量 | 理論値: 386.654 Da |
| Chemical component information |  ChemComp-CLR: |
-分子 #5: 1,2-dioleoyl-sn-glycero-3-phosphoethanolamine
| 分子 | 名称: 1,2-dioleoyl-sn-glycero-3-phosphoethanolamine / タイプ: ligand / ID: 5 / コピー数: 2 / 式: PEE |
|---|---|
| 分子量 | 理論値: 744.034 Da |
| Chemical component information |  ChemComp-PEE: |
-分子 #6: water
| 分子 | 名称: water / タイプ: ligand / ID: 6 / コピー数: 15 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.35 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| |||||||||
| グリッド | モデル: Homemade / 材質: COPPER/RHODIUM / メッシュ: 400 / 支持フィルム - 材質: GOLD / 支持フィルム - トポロジー: HOLEY / 支持フィルム - Film thickness: 30 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 20 sec. / 前処理 - 雰囲気: AIR / 前処理 - 気圧: 0.038 kPa | |||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV | |||||||||
| 詳細 | Sample was kept in the dark prior to freezing. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: SPOT SCAN / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.4 µm / 最小 デフォーカス(公称値): 0.8 µm / 倍率(公称値): 75000 Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.4 µm / 最小 デフォーカス(公称値): 0.8 µm / 倍率(公称値): 75000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: OTHER / デジタル化 - サイズ - 横: 4096 pixel / デジタル化 - サイズ - 縦: 4096 pixel / 撮影したグリッド数: 1 / 実像数: 5902 / 平均電子線量: 40.0 e/Å2 / 詳細: Falcon 4i detector |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | Chain - Source name: AlphaFold / Chain - Initial model type: in silico model |
|---|---|
| 詳細 | Initial fitting done in Phenix |
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT / 温度因子: 151.1 当てはまり具合の基準: Cross correlation coefficent |
| 得られたモデル |  PDB-8gi9: |
 ムービー
ムービー コントローラー
コントローラー






 Z
Z Y
Y X
X