+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-3219 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of transcribing mammalian RNA polymerase II (EC2) | |||||||||
 マップデータ マップデータ | Bovine Pol II elongation complex EC2, unsharpened map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード |  transcription (転写 (生物学)) / elongation transcription (転写 (生物学)) / elongation | |||||||||
| 機能・相同性 | DNA-directed 5'-3' RNA polymerase activity 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |   Bos taurus (ウシ) / Bos taurus (ウシ) /   Homo sapiens (ヒト) / unidentified (未定義) Homo sapiens (ヒト) / unidentified (未定義) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.6 Å クライオ電子顕微鏡法 / 解像度: 3.6 Å | |||||||||
 データ登録者 データ登録者 | Bernecky C / Herzog F / Baumeister W / Plitzko JM / Cramer P | |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2016 ジャーナル: Nature / 年: 2016タイトル: Structure of transcribing mammalian RNA polymerase II. 著者: Carrie Bernecky / Franz Herzog / Wolfgang Baumeister / Jürgen M Plitzko / Patrick Cramer /  要旨: RNA polymerase (Pol) II produces messenger RNA during transcription of protein-coding genes in all eukaryotic cells. The Pol II structure is known at high resolution from X-ray crystallography for ...RNA polymerase (Pol) II produces messenger RNA during transcription of protein-coding genes in all eukaryotic cells. The Pol II structure is known at high resolution from X-ray crystallography for two yeast species. Structural studies of mammalian Pol II, however, remain limited to low-resolution electron microscopy analysis of human Pol II and its complexes with various proteins. Here we report the 3.4 Å resolution cryo-electron microscopy structure of mammalian Pol II in the form of a transcribing complex comprising DNA template and RNA transcript. We use bovine Pol II, which is identical to the human enzyme except for seven amino-acid residues. The obtained atomic model closely resembles its yeast counterpart, but also reveals unknown features. Binding of nucleic acids to the polymerase involves 'induced fit' of the mobile Pol II clamp and active centre region. DNA downstream of the transcription bubble contacts a conserved 'TPSA motif' in the jaw domain of the Pol II subunit RPB5, an interaction that is apparently already established during transcription initiation. Upstream DNA emanates from the active centre cleft at an angle of approximately 105° with respect to downstream DNA. This position of upstream DNA allows for binding of the general transcription elongation factor DSIF (SPT4-SPT5) that we localize over the active centre cleft in a conserved position on the clamp domain of Pol II. Our results define the structure of mammalian Pol II in its functional state, indicate that previous crystallographic analysis of yeast Pol II is relevant for understanding gene transcription in all eukaryotes, and provide a starting point for a mechanistic analysis of human transcription. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_3219.map.gz emd_3219.map.gz | 30.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-3219-v30.xml emd-3219-v30.xml emd-3219.xml emd-3219.xml | 10.6 KB 10.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_3219.tif emd_3219.tif | 157 KB | ||
| その他 |  EMD-3219_autob.map EMD-3219_autob.map | 32.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-3219 http://ftp.pdbj.org/pub/emdb/structures/EMD-3219 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3219 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3219 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_3219.map.gz / 形式: CCP4 / 大きさ: 31.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_3219.map.gz / 形式: CCP4 / 大きさ: 31.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Bovine Pol II elongation complex EC2, unsharpened map | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.35 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-添付マップデータ: EMD-3219 autob.map
| ファイル | EMD-3219_autob.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
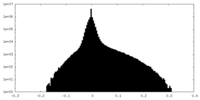
| 投影像・断面図 |
| ||||||||||||
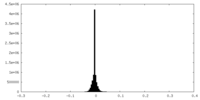
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : bovine Pol II elongation complex
| 全体 | 名称: bovine Pol II elongation complex |
|---|---|
| 要素 |
|
-超分子 #1000: bovine Pol II elongation complex
| 超分子 | 名称: bovine Pol II elongation complex / タイプ: sample / ID: 1000 詳細: Recombinant human Gdown1 was present during sample preparation but density was not observed in this map. Number unique components: 3 |
|---|---|
| 分子量 | 理論値: 590 KDa |
-分子 #1: DNA-directed RNA polymerase II
| 分子 | 名称: DNA-directed RNA polymerase II / タイプ: protein_or_peptide / ID: 1 / Name.synonym: RNA polymerase II / コピー数: 1 / 集合状態: Monomer / 組換発現: No |
|---|---|
| 由来(天然) | 生物種:   Bos taurus (ウシ) / 別称: Cow / 組織: Thymus Bos taurus (ウシ) / 別称: Cow / 組織: Thymus |
| 分子量 | 理論値: 520 KDa |
| 配列 | GO: DNA-directed 5'-3' RNA polymerase activity |
-分子 #2: DNA-directed RNA polymerase II subunit GRINL1A
| 分子 | 名称: DNA-directed RNA polymerase II subunit GRINL1A / タイプ: protein_or_peptide / ID: 2 / Name.synonym: Gdown1 / コピー数: 1 / 集合状態: Monomer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) / 別称: Human Homo sapiens (ヒト) / 別称: Human |
| 分子量 | 理論値: 40 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) / 組換株: BL21(DE3)RIL / 組換プラスミド: pOPINB Escherichia coli (大腸菌) / 組換株: BL21(DE3)RIL / 組換プラスミド: pOPINB |
-分子 #3: DNA-RNA synthetic construct
| 分子 | 名称: DNA-RNA synthetic construct / タイプ: other / ID: 3 / Name.synonym: DNA-RNA elongation scaffold / 分類: DNA/RNA / Structure: OTHER / Synthetic?: Yes |
|---|---|
| 由来(天然) | 生物種: unidentified (未定義) |
| 分子量 | 理論値: 30 KDa |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.3 mg/mL |
|---|---|
| 緩衝液 | pH: 7.25 / 詳細: 150 mM NaCl, 5 mM HEPES, 0.01 mM ZnCl2, 10 mM DTT |
| グリッド | 詳細: Quantifoil R 3.5/1 holey carbon grids |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / 装置: FEI VITROBOT MARK IV 手法: Four microliters of sample was applied to glow-discharged Quantifoil R 3.5/1 holey carbon grids, which were then blotted for 8.5s and plunge-frozen in liquid ethane. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 37037 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2 mm / 最大 デフォーカス(公称値): 3.1 µm / 最小 デフォーカス(公称値): 0.6 µm / 倍率(公称値): 37000 Bright-field microscopy / Cs: 2 mm / 最大 デフォーカス(公称値): 3.1 µm / 最小 デフォーカス(公称値): 0.6 µm / 倍率(公称値): 37000 |
| 特殊光学系 | エネルギーフィルター - 名称: GIF Quantum エネルギーフィルター - エネルギー下限: 0.0 eV エネルギーフィルター - エネルギー上限: 20.0 eV |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 日付 | 2014年12月1日 |
| 撮影 | カテゴリ: CCD フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 実像数: 1172 / 平均電子線量: 43 e/Å2 詳細: Each movie image was collected over 8 s fractionated into 40 frames (0.2 s each). |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| CTF補正 | 詳細: Each particle |
|---|---|
| 最終 再構成 | 想定した対称性 - 点群: C1 (非対称) / アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 3.6 Å / 解像度の算出法: OTHER / ソフトウェア - 名称: RELION / 使用した粒子像数: 219265 |
 ムービー
ムービー コントローラー
コントローラー
















 Z
Z Y
Y X
X