+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM imaging scaffold subunits A and B used to display KRAS G12C complex with GDP | |||||||||
 マップデータ マップデータ | scaffold subunits A and B used to display KRAS G12C complex with GDP 足場 足場 | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | CryoEM imaging scaffold /  Cancer (悪性腫瘍) / Cancer (悪性腫瘍) /  GTPase (GTPアーゼ) / GTPase (GTPアーゼ) /  STRUCTURAL PROTEIN (タンパク質) STRUCTURAL PROTEIN (タンパク質) | |||||||||
| 機能・相同性 |  Cobalamin adenosyltransferase-like / Corrinoid adenosyltransferase, PduO-type / Cobalamin adenosyltransferase-like / Corrinoid adenosyltransferase, PduO-type /  Cobalamin adenosyltransferase / Cobalamin adenosyltransferase-like superfamily / Cobalamin adenosyltransferase / Cobalamin adenosyltransferase-like superfamily /  transferase activity / transferase activity /  ATP binding / Cobalamin adenosyltransferase-like domain-containing protein ATP binding / Cobalamin adenosyltransferase-like domain-containing protein 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 | synthetic construct (人工物) /    Thermoplasma acidophilum (好酸性) Thermoplasma acidophilum (好酸性) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 2.2 Å クライオ電子顕微鏡法 / 解像度: 2.2 Å | |||||||||
 データ登録者 データ登録者 | Castells-Graells R / Sawaya MR / Yeates TO | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2023 ジャーナル: Proc Natl Acad Sci U S A / 年: 2023タイトル: Cryo-EM structure determination of small therapeutic protein targets at 3 Å-resolution using a rigid imaging scaffold. 著者: Roger Castells-Graells / Kyle Meador / Mark A Arbing / Michael R Sawaya / Morgan Gee / Duilio Cascio / Emma Gleave / Judit É Debreczeni / Jason Breed / Karoline Leopold / Ankoor Patel / ...著者: Roger Castells-Graells / Kyle Meador / Mark A Arbing / Michael R Sawaya / Morgan Gee / Duilio Cascio / Emma Gleave / Judit É Debreczeni / Jason Breed / Karoline Leopold / Ankoor Patel / Dushyant Jahagirdar / Bronwyn Lyons / Sriram Subramaniam / Chris Phillips / Todd O Yeates /    要旨: Cryoelectron microscopy (Cryo-EM) has enabled structural determination of proteins larger than about 50 kDa, including many intractable by any other method, but it has largely failed for smaller ...Cryoelectron microscopy (Cryo-EM) has enabled structural determination of proteins larger than about 50 kDa, including many intractable by any other method, but it has largely failed for smaller proteins. Here, we obtain structures of small proteins by binding them to a rigid molecular scaffold based on a designed protein cage, revealing atomic details at resolutions reaching 2.9 Å. We apply this system to the key cancer signaling protein KRAS (19 kDa in size), obtaining four structures of oncogenic mutational variants by cryo-EM. Importantly, a structure for the key G12C mutant bound to an inhibitor drug (AMG510) reveals significant conformational differences compared to prior data in the crystalline state. The findings highlight the promise of cryo-EM scaffolds for advancing the design of drug molecules against small therapeutic protein targets in cancer and other human diseases. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
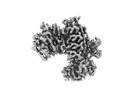
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_29700.map.gz emd_29700.map.gz | 86.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-29700-v30.xml emd-29700-v30.xml emd-29700.xml emd-29700.xml | 16.7 KB 16.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_29700_fsc.xml emd_29700_fsc.xml | 9.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_29700.png emd_29700.png | 62.3 KB | ||
| その他 |  emd_29700_half_map_1.map.gz emd_29700_half_map_1.map.gz emd_29700_half_map_2.map.gz emd_29700_half_map_2.map.gz | 84.7 MB 84.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-29700 http://ftp.pdbj.org/pub/emdb/structures/EMD-29700 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-29700 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-29700 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_29700.map.gz / 形式: CCP4 / 大きさ: 91.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_29700.map.gz / 形式: CCP4 / 大きさ: 91.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | scaffold subunits A and B used to display KRAS G12C complex with GDP | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
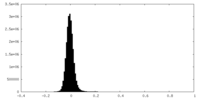
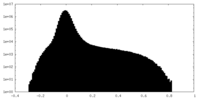
-ハーフマップ: Half Map 1
| ファイル | emd_29700_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half Map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
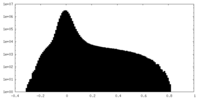
-ハーフマップ: Half Map 2
| ファイル | emd_29700_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half Map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Cryo-EM imaging scaffold displaying KRAS G12C
| 全体 | 名称: Cryo-EM imaging scaffold displaying KRAS G12C |
|---|---|
| 要素 |
|
-超分子 #1: Cryo-EM imaging scaffold displaying KRAS G12C
| 超分子 | 名称: Cryo-EM imaging scaffold displaying KRAS G12C / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
-分子 #1: Cob_adeno_trans domain-containing protein
| 分子 | 名称: Cob_adeno_trans domain-containing protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:    Thermoplasma acidophilum (好酸性) Thermoplasma acidophilum (好酸性) |
| 分子量 | 理論値: 20.173395 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: MRITTKVGDK GSTRLFGGEE VWKDDPIIEA NGTLDELTSF IGEAKHYVDE EMKGILEEIQ NDIYKIMGEI GSKGKIEGIS EERIKWLAG LIERYSEMVN KLSFVLPGGT LESAKLDVCR TIARRAERKV ATVLREFGIG TLAAIYLALL SRLLFLLARV I EIEKNKLK EVRSHHHHHH UniProtKB: Cobalamin adenosyltransferase-like domain-containing protein |
-分子 #2: Cryo-EM imaging scaffold subunit B with DARPin - RCG-33
| 分子 | 名称: Cryo-EM imaging scaffold subunit B with DARPin - RCG-33 タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 35.452441 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: MFTRRGDQGE TDLANRARVG KDSPVVEVQG TIDELNSFIG YALVLSRWDD IRNDLFRIQN DLFVLGEDVS TGGKGRTVTM DMIIYLIKR SVEMKAEIGK IELFVVPGGS VESASLHMAR AVSRRLERRI KAASELTEIN ANVLLYANML SNILFMHALI S NKRKEELD ...文字列: MFTRRGDQGE TDLANRARVG KDSPVVEVQG TIDELNSFIG YALVLSRWDD IRNDLFRIQN DLFVLGEDVS TGGKGRTVTM DMIIYLIKR SVEMKAEIGK IELFVVPGGS VESASLHMAR AVSRRLERRI KAASELTEIN ANVLLYANML SNILFMHALI S NKRKEELD KKLLEAARAG QDDEVAALLA KGADVNAHDT FGFTPLHLAA LYGHLEIVEV LLKRGADINA DDSYGRTPLH LA AMRGHLE IVELLLRWGA DVNAADEEGR TPLHLAAKRG HLEIVEVLLK NGADVNAQDK FGKTAFDISI DNGNEDLAEI LQK L |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| グリッド | モデル: Quantifoil R2/2 / 材質: COPPER / メッシュ: 300 |
| 凍結 | 凍結剤: ETHANE / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.25 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 155000 Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.25 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 155000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) 平均電子線量: 40.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: AB INITIO MODEL |
|---|---|
| 得られたモデル |  PDB-8g3k: |
 ムービー
ムービー コントローラー
コントローラー













 Z
Z Y
Y X
X