+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Inactivate state of Maribacter polysiphoniae Argonuate (short pAgo system) | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Argonuate /  TIR domain / TIR domain /  Oligomerization (オリゴマー) / Oligomerization (オリゴマー) /  NAD+ (ニコチンアミドアデニンジヌクレオチド) / NAD+ (ニコチンアミドアデニンジヌクレオチド) /  IMMUNE SYSTEM (免疫系) IMMUNE SYSTEM (免疫系) | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Maribacter polysiphoniae (バクテリア) Maribacter polysiphoniae (バクテリア) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.07 Å クライオ電子顕微鏡法 / 解像度: 3.07 Å | |||||||||
 データ登録者 データ登録者 | Shen ZF / Yang XY / Fu TM | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2023 ジャーナル: Nature / 年: 2023タイトル: Oligomerization-mediated activation of a short prokaryotic Argonaute. 著者: Zhangfei Shen / Xiao-Yuan Yang / Shiyu Xia / Wei Huang / Derek J Taylor / Kotaro Nakanishi / Tian-Min Fu /  要旨: Although eukaryotic and long prokaryotic Argonaute proteins (pAgos) cleave nucleic acids, some short pAgos lack nuclease activity and hydrolyse NAD(P) to induce bacterial cell death. Here we present ...Although eukaryotic and long prokaryotic Argonaute proteins (pAgos) cleave nucleic acids, some short pAgos lack nuclease activity and hydrolyse NAD(P) to induce bacterial cell death. Here we present a hierarchical activation pathway for SPARTA, a short pAgo consisting of an Argonaute (Ago) protein and TIR-APAZ, an associated protein. SPARTA progresses through distinct oligomeric forms, including a monomeric apo state, a monomeric RNA-DNA-bound state, two dimeric RNA-DNA-bound states and a tetrameric RNA-DNA-bound active state. These snapshots together identify oligomerization as a mechanistic principle of SPARTA activation. The RNA-DNA-binding channel of apo inactive SPARTA is occupied by an auto-inhibitory motif in TIR-APAZ. After the binding of RNA-DNA, SPARTA transitions from a monomer to a symmetric dimer and then an asymmetric dimer, in which two TIR domains interact through charge and shape complementarity. Next, two dimers assemble into a tetramer with a central TIR cluster responsible for hydrolysing NAD(P). In addition, we observe unique features of interactions between SPARTA and RNA-DNA, including competition between the DNA 3' end and the auto-inhibitory motif, interactions between the RNA G2 nucleotide and Ago, and splaying of the RNA-DNA duplex by two loops exclusive to short pAgos. Together, our findings provide a mechanistic basis for the activation of short pAgos, a large section of the Ago superfamily. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_29033.map.gz emd_29033.map.gz | 289.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-29033-v30.xml emd-29033-v30.xml emd-29033.xml emd-29033.xml | 17 KB 17 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_29033_fsc.xml emd_29033_fsc.xml | 14.3 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_29033.png emd_29033.png | 65.6 KB | ||
| その他 |  emd_29033_additional_1.map.gz emd_29033_additional_1.map.gz emd_29033_half_map_1.map.gz emd_29033_half_map_1.map.gz emd_29033_half_map_2.map.gz emd_29033_half_map_2.map.gz | 263.3 MB 285.3 MB 285.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-29033 http://ftp.pdbj.org/pub/emdb/structures/EMD-29033 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-29033 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-29033 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_29033.map.gz / 形式: CCP4 / 大きさ: 307.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_29033.map.gz / 形式: CCP4 / 大きさ: 307.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 0.5347 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: sharped map
| ファイル | emd_29033_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | sharped map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
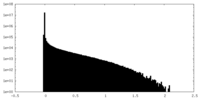
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_29033_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
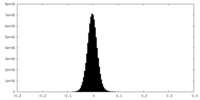
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_29033_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
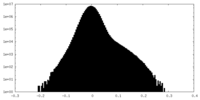
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : MapSPARTA heterodimeric complex(short pAgo)
| 全体 | 名称: MapSPARTA heterodimeric complex(short pAgo) |
|---|---|
| 要素 |
|
-超分子 #1: MapSPARTA heterodimeric complex(short pAgo)
| 超分子 | 名称: MapSPARTA heterodimeric complex(short pAgo) / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Maribacter polysiphoniae (バクテリア) Maribacter polysiphoniae (バクテリア) |
-分子 #1: TIR-APAZ
| 分子 | 名称: TIR-APAZ / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Maribacter polysiphoniae (バクテリア) Maribacter polysiphoniae (バクテリア) |
| 分子量 | 理論値: 53.270594 KDa |
| 組換発現 | 生物種:   Escherichia coli 'BL21-Gold(DE3)pLysS AG' (大腸菌) Escherichia coli 'BL21-Gold(DE3)pLysS AG' (大腸菌) |
| 配列 | 文字列: MRNKIFISHA TPDDNDFTRW LALKLIGLGY EVWCDILFLD KGVDFWSNIE KVIREDTCKF LLVSSSYSNQ REGVLKELAV AAKVKKQLK DDKFIIPLAI DEQLSYDDIN IDIVRLNAID FKMSWARGLK DILEAFEKQK VPKEVADASK SNLLYQQIFL H DKSVIEKE ...文字列: MRNKIFISHA TPDDNDFTRW LALKLIGLGY EVWCDILFLD KGVDFWSNIE KVIREDTCKF LLVSSSYSNQ REGVLKELAV AAKVKKQLK DDKFIIPLAI DEQLSYDDIN IDIVRLNAID FKMSWARGLK DILEAFEKQK VPKEVADASK SNLLYQQIFL H DKSVIEKE EIYDSNWLSI LSFPEELRFH EYNWMLPKRF DVRELTFPAV RYKNYLCTFA WAYDFTYHLP KTETYHKSKT IR IPTEEIL SGSYDSNFIR NAECKRLIVQ LLNKAFELRM KDKEVQEYEM SNKTAYWLEK GKLEKDKFEK TMLVGKQKDK NWH FAISGA SKLYPFPVLM ISSHIFFTAD GKKLIDSSSV QHSSRRRQGK NWWNNTWRTK LLAFIKYLSD DDTSFYLEMG SEEK VFVSN EPVKFKGNVS YNIPEKNTLE EEAELSGFNQ GEDIEELEEL IENLEAE UniProtKB: TIR domain-containing protein |
-分子 #2: short pAgo
| 分子 | 名称: short pAgo / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Maribacter polysiphoniae (バクテリア) Maribacter polysiphoniae (バクテリア) |
| 分子量 | 理論値: 58.09141 KDa |
| 組換発現 | 生物種:   Escherichia coli 'BL21-Gold(DE3)pLysS AG' (大腸菌) Escherichia coli 'BL21-Gold(DE3)pLysS AG' (大腸菌) |
| 配列 | 文字列: MKELIYIEEP KILFAHGQKC TDARDGLALF GPLNNLYGIK SGVIGTKQGL KIFRDYLDHI QKPIYNSNSI TRPMFPGFEA VFDCKWEST GITFKEVTNE DIGKFLYNSS THKRTYDLVS LFIDKIISAN KNEDENVDVW FVIVPDEIYK YCRPNSVLPK E MVQTKALM ...文字列: MKELIYIEEP KILFAHGQKC TDARDGLALF GPLNNLYGIK SGVIGTKQGL KIFRDYLDHI QKPIYNSNSI TRPMFPGFEA VFDCKWEST GITFKEVTNE DIGKFLYNSS THKRTYDLVS LFIDKIISAN KNEDENVDVW FVIVPDEIYK YCRPNSVLPK E MVQTKALM SKSKAKSFRY EPSLFPDINI ELKEQEKEAE TYNYDAQFHD QFKARLLKHT IPTQIFREST LAWRDFKNAF GL PIRDFSK IEGHLAWTIS TAAFYKAGGK PWKLSDVRNG VCYLGLVYKK VEKSKNPRNA CCAAQMFLDN GDGTVFKGEV GPW YNPKNG QYHLEPKEAK ALLSQSLQSY KEQIGEYPKE VFIHAKTRFN HQEWDAFLEV TPKETNLVGV TISKTKPLKL YKTE GDYTI LRGNAYVVNE RSAFLWTVGY VPKIQTALSM EVPNPLFIEI NKGEADIKQV LKDILSLTKL NYNACIFADG EPVTL RFAD KIGEILTAST DIKTPPLAFK YYI UniProtKB: Piwi domain-containing protein |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 0.5 µm Bright-field microscopy / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 0.5 µm |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 50.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー























 Z
Z Y
Y X
X