+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
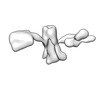
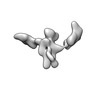
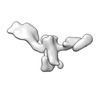
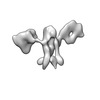
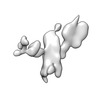
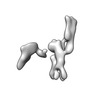
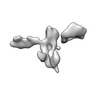
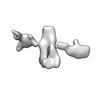
| タイトル | Single-Molecule 3D Image of 16 Helix RNA Origami Satellite by Individual Particle Electron Tomography (No. 14) | ||||||||||||||||||
 マップデータ マップデータ | 3D Image of 16 Helix RNA Origami Satellite by Individual Particle Electron Tomography (No. 14) | ||||||||||||||||||
 試料 試料 |
| ||||||||||||||||||
 キーワード キーワード |  RNA (リボ核酸) / RNA (リボ核酸) /  origami (折り紙) / Individual Particle Electron Tomography / Single-Molecule 3D Image origami (折り紙) / Individual Particle Electron Tomography / Single-Molecule 3D Image | ||||||||||||||||||
| 生物種 | unidentified (未定義) | ||||||||||||||||||
| 手法 |  電子線トモグラフィー法 / 電子線トモグラフィー法 /  クライオ電子顕微鏡法 / 解像度: 27.0 Å クライオ電子顕微鏡法 / 解像度: 27.0 Å | ||||||||||||||||||
 データ登録者 データ登録者 | Liu J / Ren G | ||||||||||||||||||
| 資金援助 |  米国, 5件 米国, 5件
| ||||||||||||||||||
 引用 引用 |  ジャーナル: Nat Nanotechnol / 年: 2023 ジャーナル: Nat Nanotechnol / 年: 2023タイトル: Structure, folding and flexibility of co-transcriptional RNA origami. 著者: Ewan K S McRae / Helena Østergaard Rasmussen / Jianfang Liu / Andreas Bøggild / Michael T A Nguyen / Nestor Sampedro Vallina / Thomas Boesen / Jan Skov Pedersen / Gang Ren / Cody Geary / Ebbe Sloth Andersen /   要旨: RNA origami is a method for designing RNA nanostructures that can self-assemble through co-transcriptional folding with applications in nanomedicine and synthetic biology. However, to advance the ...RNA origami is a method for designing RNA nanostructures that can self-assemble through co-transcriptional folding with applications in nanomedicine and synthetic biology. However, to advance the method further, an improved understanding of RNA structural properties and folding principles is required. Here we use cryogenic electron microscopy to study RNA origami sheets and bundles at sub-nanometre resolution revealing structural parameters of kissing-loop and crossover motifs, which are used to improve designs. In RNA bundle designs, we discover a kinetic folding trap that forms during folding and is only released after 10 h. Exploration of the conformational landscape of several RNA designs reveal the flexibility of helices and structural motifs. Finally, sheets and bundles are combined to construct a multidomain satellite shape, which is characterized by individual-particle cryo-electron tomography to reveal the domain flexibility. Together, the study provides a structural basis for future improvements to the design cycle of genetically encoded RNA nanodevices. | ||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_28682.map.gz emd_28682.map.gz | 59.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-28682-v30.xml emd-28682-v30.xml emd-28682.xml emd-28682.xml | 13.6 KB 13.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_28682_fsc.xml emd_28682_fsc.xml | 10.8 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_28682.png emd_28682.png | 33 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-28682 http://ftp.pdbj.org/pub/emdb/structures/EMD-28682 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-28682 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-28682 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_28682.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_28682.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | 3D Image of 16 Helix RNA Origami Satellite by Individual Particle Electron Tomography (No. 14) | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.1 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : 16 Helix RNA origami satellite
| 全体 | 名称: 16 Helix RNA origami satellite |
|---|---|
| 要素 |
|
-超分子 #1: 16 Helix RNA origami satellite
| 超分子 | 名称: 16 Helix RNA origami satellite / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all / 詳細: In vitro purified RNA. |
|---|---|
| 由来(天然) | 生物種: unidentified (未定義) |
| 分子量 | 理論値: 590 KDa |
-分子 #1: 16 Helix RNA origami satellite
| 分子 | 名称: 16 Helix RNA origami satellite / タイプ: rna / ID: 1 |
|---|---|
| 配列 | 文字列: GGAACAAGCA UAACCCGUCC AGGUUCUCAA GACCAAAGAG AACUUGGACC UAUAGUCGGC GUCUACCCGU GUCGCCACGG GUGGACGCCG GCUAUAGGCU UCCCUUUCAU CGCACCUCGC GUUCGCACGC GGGGUGCGCU GACGGUGGGG CUAGCGCCCC AUCGUCAGCC ...文字列: GGAACAAGCA UAACCCGUCC AGGUUCUCAA GACCAAAGAG AACUUGGACC UAUAGUCGGC GUCUACCCGU GUCGCCACGG GUGGACGCCG GCUAUAGGCU UCCCUUUCAU CGCACCUCGC GUUCGCACGC GGGGUGCGCU GACGGUGGGG CUAGCGCCCC AUCGUCAGCC CUAUCCCGUU ACUUUACAGA GGUCCCGCGC AAUUUGUGGA CCUUGGCAGG UCCGCAAAUU GCGUGGGACG GUGCGGGGUU UCAUGGCUGA AACCUCGCAC CGUAAUAGCG GUGAUUAACG AGCUAGCGCU CGUUGAUCAC CCACUUCGGU AACUCAAGAA CCGGAGUGCG UUUGGAUCUU CAAUCCUAGA GAAGGUCCAA ACGGCUGUUA CCUCUGUGAA GUAACGACGC GUCACAAUGG ACUAGUGAUG CGUCGCACGC UACCCCAAAA CCAGAGGGGU GGCGUGCGGG GUAGGGAUGA AGGGGAAGCG GGUUGUGUUU GUUCCCAAAU ACCUUCUUCG GGCCUCUCUU CCAUGAUACU AUUGAUUAUU UCGCUUCUAA UAUACCCCGU AGUCUCCGGA CUACGCCCGA CGUCCGCGUC GGGGAGGGGC UAACAGACUA AGCUCCUCGG CCCGUAAUAC CGAAACGGGG UAAUACCCAC CCGCAGUUCC CGGGACUGUA AUCGGUAAAC AGUCGGCGCA AAGUCUGAGC GCCCUUACUC UUCGGAGUAA GCCGGCAGUA UACGUACUGC CCGAGGGUGC UCGUAUUUGU GCGCAAAUAC CGCAUAUGGU ACGCCAUAUG GGAGUGGUGA CUUUCGAGUC AUCACUCCCA CGUUUGAAGG AUGAACAAGC GUGCGCCCUU GAAAUCGUCA CAAGGGGAGC GCCCUCGGGG AAUUGCGGCG GUCACAAGAC GAUAGUGACG GCCCAAUCAU CCAGGGCCCC AACUCUACGG AGUUGGCGCU AGUCUUACGA GACUAGGUGG GUGUUACGUA UAUUGGAAGC GAGAUAAUCA AUGGUAUCCU GGUUAAUGGA GGAGAGGGGC GUUUUAUAGU GAAAGUCCAA CACUAUGAAA CCGGGCUCCG GGAUCAACUA GGAAGAUCUC GGAGCGGCGC AUUUUCUAUU CGCCUUCUCU UACAUAUCUA AAUACUCUUU UCUCGCGCUG GUAUCCGUAC CAGCCGGAUG AUACGUCAUC CGCAGCUGAG AAUAGGUGAC UCAGCUGGCG CGGAAAUCCU GUAUCCGCCU UACUUCCGCC UACGCUCUAG GCGUGGAUAA ACAGGAAAUC CACGACCCAA CACCUAAGGG UCGAGCAAUU CCGAUUGCUC GCCCUCGAAU ACGUUCGAGG CUGGCGUGCU GGGUGGUGUC CGCACCACCG GGAUCCUGUA CGCAGGAUCG GCGAGGAGGG UUUCGACCCU UCUCGCCGAG UGAAUAACUG UUGAAUUCGC UCCCCGUCCC AAGUCUUGAG GGACGCAGCA UGCCAGCUAG GGCGUAGCGG CCUUAACAAG ACAAAGGCGA GGUAACAACA GAACCUCCCC UGUCUGCGGA CAGGGCGGUU GCUCUCCGGA GCAACGCGGA GGUAAGGAGA GAAGAGUGUU UAGAUGUGUA AGAGGAGGCG CUUGAGAAAU AGAGAAUGCC AUAGCCGUAU GUAAAUUGGU CAUACAUAUG GCUAUGGGUC UGCAGUUACG ACUGCGGACC GCCCUGAUGU AGGUACGCCU AUAUCAGCCG GGCUGCUCCG UACGCGGAGU AGCCGCCGUC GGGUGGCUAC GGCCAUCCGA CCCCGCAGUC AUGAUUCGUC AUGGCUGCCA GACGUGCGGA UUACGAUCCG UACGUCUGGA AGGAGGUGUU UG |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  電子線トモグラフィー法 電子線トモグラフィー法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1.8 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 構成要素:
| ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 90 % / チャンバー内温度: 277 K / 装置: LEICA EM GP | ||||||||||||
| 詳細 | In vitro transcribed RNA was purified by size exclusion chromatography and spin concentrated in amicon spin columns. | ||||||||||||
| 切片作成 | その他: NO SECTIONING |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 2.0 µm / 倍率(公称値): 42000 Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 2.0 µm / 倍率(公称値): 42000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 6.2 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー