+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-2750 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
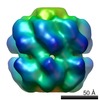
| タイトル | Structure of the CsgG-CsgE complex | |||||||||
 マップデータ マップデータ | reconstruction of the CsgG-CsgE complex | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Bacterial secretion /  membrane protein (膜タンパク質) membrane protein (膜タンパク質) | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 : / curli secretion complex / : / curli secretion complex /  curli assembly / protein secretion by the type VIII secretion system / protein transmembrane transport / single-species biofilm formation / cell outer membrane / outer membrane-bounded periplasmic space / identical protein binding / curli assembly / protein secretion by the type VIII secretion system / protein transmembrane transport / single-species biofilm formation / cell outer membrane / outer membrane-bounded periplasmic space / identical protein binding /  細胞膜 細胞膜類似検索 - 分子機能 | |||||||||
| 生物種 |   Escherichia coli K-12 (大腸菌) Escherichia coli K-12 (大腸菌) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 24.0 Å クライオ電子顕微鏡法 / 解像度: 24.0 Å | |||||||||
 データ登録者 データ登録者 | Krasteva PV / Gubellini F / Remaut H / Fronzes R | |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2014 ジャーナル: Nature / 年: 2014タイトル: Structural and mechanistic insights into the bacterial amyloid secretion channel CsgG. 著者: Parveen Goyal / Petya V Krasteva / Nani Van Gerven / Francesca Gubellini / Imke Van den Broeck / Anastassia Troupiotis-Tsaïlaki / Wim Jonckheere / Gérard Péhau-Arnaudet / Jerome S Pinkner ...著者: Parveen Goyal / Petya V Krasteva / Nani Van Gerven / Francesca Gubellini / Imke Van den Broeck / Anastassia Troupiotis-Tsaïlaki / Wim Jonckheere / Gérard Péhau-Arnaudet / Jerome S Pinkner / Matthew R Chapman / Scott J Hultgren / Stefan Howorka / Rémi Fronzes / Han Remaut /     要旨: Curli are functional amyloid fibres that constitute the major protein component of the extracellular matrix in pellicle biofilms formed by Bacteroidetes and Proteobacteria (predominantly of the α ...Curli are functional amyloid fibres that constitute the major protein component of the extracellular matrix in pellicle biofilms formed by Bacteroidetes and Proteobacteria (predominantly of the α and γ classes). They provide a fitness advantage in pathogenic strains and induce a strong pro-inflammatory response during bacteraemia. Curli formation requires a dedicated protein secretion machinery comprising the outer membrane lipoprotein CsgG and two soluble accessory proteins, CsgE and CsgF. Here we report the X-ray structure of Escherichia coli CsgG in a non-lipidated, soluble form as well as in its native membrane-extracted conformation. CsgG forms an oligomeric transport complex composed of nine anticodon-binding-domain-like units that give rise to a 36-stranded β-barrel that traverses the bilayer and is connected to a cage-like vestibule in the periplasm. The transmembrane and periplasmic domains are separated by a 0.9-nm channel constriction composed of three stacked concentric phenylalanine, asparagine and tyrosine rings that may guide the extended polypeptide substrate through the secretion pore. The specificity factor CsgE forms a nonameric adaptor that binds and closes off the periplasmic face of the secretion channel, creating a 24,000 Å(3) pre-constriction chamber. Our structural, functional and electrophysiological analyses imply that CsgG is an ungated, non-selective protein secretion channel that is expected to employ a diffusion-based, entropy-driven transport mechanism. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_2750.map.gz emd_2750.map.gz | 790.7 KB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-2750-v30.xml emd-2750-v30.xml emd-2750.xml emd-2750.xml | 11.9 KB 11.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  image2750.png image2750.png | 70.9 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-2750 http://ftp.pdbj.org/pub/emdb/structures/EMD-2750 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-2750 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-2750 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_2750.map.gz / 形式: CCP4 / 大きさ: 15.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_2750.map.gz / 形式: CCP4 / 大きさ: 15.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | reconstruction of the CsgG-CsgE complex | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.99 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : CsgG-CsgE complex
| 全体 | 名称: CsgG-CsgE complex |
|---|---|
| 要素 |
|
-超分子 #1000: CsgG-CsgE complex
| 超分子 | 名称: CsgG-CsgE complex / タイプ: sample / ID: 1000 詳細: The sample was reconstituted from purified components in vitro. 集合状態: One nonamer of CsgG bound to one nonamer of CsgE Number unique components: 2 |
|---|---|
| 分子量 | 理論値: 400 KDa |
-分子 #1: CsgG
| 分子 | 名称: CsgG / タイプ: protein_or_peptide / ID: 1 Name.synonym: Curli production assembly/transport component CsgG 詳細: Strep-tagged / コピー数: 9 / 集合状態: nonamer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:   Escherichia coli K-12 (大腸菌) / 株: MC4100 / 細胞中の位置: Outer membrane Escherichia coli K-12 (大腸菌) / 株: MC4100 / 細胞中の位置: Outer membrane |
| 分子量 | 理論値: 290 KDa |
| 組換発現 | 生物種:   Escherichia coli BL21(DE3) (大腸菌) / 組換プラスミド: pPG1 (pASK-IBA3plus derivative) Escherichia coli BL21(DE3) (大腸菌) / 組換プラスミド: pPG1 (pASK-IBA3plus derivative) |
| 配列 | UniProtKB: Curli production assembly/transport component CsgG GO: GO: 0022610, single-species biofilm formation, protein transmembrane transport, cell outer membrane InterPro: Curli production assembly/transport component CsgG |
-分子 #2: CsgE
| 分子 | 名称: CsgE / タイプ: protein_or_peptide / ID: 2 Name.synonym: Curli production assembly/transport component CsgE 詳細: Poly-histidine tagged. Used for CsgE-CsgG complex reconstitution and pull-down on Talon metal affinity resin. コピー数: 9 / 集合状態: nonamer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:   Escherichia coli K-12 (大腸菌) / 株: MC4100 / 細胞中の位置: Periplasm Escherichia coli K-12 (大腸菌) / 株: MC4100 / 細胞中の位置: Periplasm |
| 分子量 | 理論値: 110 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) / 組換株: NEBC2566 / 組換プラスミド: pNH27 (pET11d derivative) Escherichia coli (大腸菌) / 組換株: NEBC2566 / 組換プラスミド: pNH27 (pET11d derivative) |
| 配列 | UniProtKB: Curli production assembly/transport component CsgE GO: GO: 0022610, single-species biofilm formation, protein transmembrane transport, cell outer membrane InterPro: Curli assembly protein CsgE |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.05 mg/mL |
|---|---|
| 緩衝液 | pH: 8 詳細: 20 mM Tris-HCl pH 8.0, 100 mM NaCl, 100 mM imidazole, 2.5% xylitol, 0.2 mM DTT |
| グリッド | 詳細: Quantifoil R2/2 carbon coated grids (Quantifoil Micro Tools GmbH) |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 80 % / チャンバー内温度: 92 K / 装置: LEICA EM GP / 手法: Blot for 0.5 seconds before plunging |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI F20 |
|---|---|
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 70350 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 50000 Bright-field microscopy / Cs: 2 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 50000 |
| 試料ステージ | 試料ホルダーモデル: GATAN LIQUID NITROGEN |
| 温度 | 平均: 95 K |
| アライメント法 | Legacy - 非点収差: Astigmatism was checked during imaging and during CTF correction of the micrographs. Micrographs is astigmatism >10% were discarded. |
| 日付 | 2014年6月2日 |
| 撮影 | カテゴリ: CCD フィルム・検出器のモデル: FEI FALCON II (4k x 4k) 実像数: 75 / 平均電子線量: 20 e/Å2 |
| 実験機器 |  モデル: Tecnai F20 / 画像提供: FEI Company |
- 画像解析
画像解析
| CTF補正 | 詳細: Each micrograph |
|---|---|
| 最終 2次元分類 | クラス数: 125 |
| 最終 再構成 | 想定した対称性 - 点群: C9 (9回回転対称 ) / アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 24.0 Å / 解像度の算出法: OTHER / ソフトウェア - 名称: IMAGIC / 使用した粒子像数: 1221 ) / アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 24.0 Å / 解像度の算出法: OTHER / ソフトウェア - 名称: IMAGIC / 使用した粒子像数: 1221 |
| 詳細 | Particles were automatically selected from CTF-corrected micrographs using BOXER (EMAN2). |
 ムービー
ムービー コントローラー
コントローラー