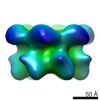
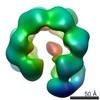
登録情報 データベース : EMDB / ID : EMD-2592タイトル Inter-ring rotations of AAA ATPase p97 Reconstruction of p97 in presence of ADP, conformation 2 試料 : p97 in presence of ADP, conformation 2タンパク質・ペプチド : Transitional endoplasmic reticulum ATPase / 機能・相同性 分子機能 ドメイン・相同性 構成要素
/ / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / 生物種 Mus musculus (ハツカネズミ)手法 / / 解像度 : 20.0 Å Yeung HO / Forster A / Bebeacua C / Niwa H / Ewens C / McKeown C / Zhang X / Freemont PS ジャーナル : Open Biol / 年 : 2014タイトル : Inter-ring rotations of AAA ATPase p97 revealed by electron cryomicroscopy.著者 : Heidi O Yeung / Andreas Förster / Cecilia Bebeacua / Hajime Niwa / Caroline Ewens / Ciarán McKeown / Xiaodong Zhang / Paul S Freemont / 要旨 : The type II AAA+ protein p97 is involved in numerous cellular activities, including endoplasmic reticulum-associated degradation, transcription activation, membrane fusion and cell-cycle control. ... The type II AAA+ protein p97 is involved in numerous cellular activities, including endoplasmic reticulum-associated degradation, transcription activation, membrane fusion and cell-cycle control. These activities are at least in part regulated by the ubiquitin system, in which p97 is thought to target ubiquitylated protein substrates within macromolecular complexes and assist in their extraction or disassembly. Although ATPase activity is essential for p97 function, little is known about how ATP binding or hydrolysis is coupled with p97 conformational changes and substrate remodelling. Here, we have used single-particle electron cryomicroscopy (cryo-EM) to study the effect of nucleotides on p97 conformation. We have identified conformational heterogeneity within the cryo-EM datasets from which we have resolved two major p97 conformations. A comparison of conformations reveals inter-ring rotations upon nucleotide binding and hydrolysis that may be linked to the remodelling of target protein complexes. 履歴 登録 2014年2月21日 - ヘッダ(付随情報) 公開 2014年2月26日 - マップ公開 2014年2月26日 - 更新 2014年3月19日 - 現状 2014年3月19日 処理サイト : PDBe / 状態 : 公開
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報 マップデータ
マップデータ 試料
試料 キーワード
キーワード AAA ATPase / p97
AAA ATPase / p97 機能・相同性情報
機能・相同性情報 Protein methylation / Translesion Synthesis by POLH / Josephin domain DUBs / N-glycan trimming in the ER and Calnexin/Calreticulin cycle / Ovarian tumor domain proteases / Hedgehog ligand biogenesis / KEAP1-NFE2L2 pathway / ABC-family proteins mediated transport ...RHOH GTPase cycle / HSF1 activation /
Protein methylation / Translesion Synthesis by POLH / Josephin domain DUBs / N-glycan trimming in the ER and Calnexin/Calreticulin cycle / Ovarian tumor domain proteases / Hedgehog ligand biogenesis / KEAP1-NFE2L2 pathway / ABC-family proteins mediated transport ...RHOH GTPase cycle / HSF1 activation /  Protein methylation / Translesion Synthesis by POLH / Josephin domain DUBs / N-glycan trimming in the ER and Calnexin/Calreticulin cycle / Ovarian tumor domain proteases / Hedgehog ligand biogenesis / KEAP1-NFE2L2 pathway / ABC-family proteins mediated transport /
Protein methylation / Translesion Synthesis by POLH / Josephin domain DUBs / N-glycan trimming in the ER and Calnexin/Calreticulin cycle / Ovarian tumor domain proteases / Hedgehog ligand biogenesis / KEAP1-NFE2L2 pathway / ABC-family proteins mediated transport /  Neddylation / flavin adenine dinucleotide catabolic process / positive regulation of oxidative phosphorylation / VCP-NSFL1C complex / endosome to lysosome transport via multivesicular body sorting pathway / protein-DNA covalent cross-linking repair / positive regulation of ubiquitin-dependent protein catabolic process / endoplasmic reticulum stress-induced pre-emptive quality control / cellular response to arsenite ion / BAT3 complex binding / Derlin-1 retrotranslocation complex / positive regulation of protein K63-linked deubiquitination / deubiquitinase activator activity / mitotic spindle disassembly / VCP-NPL4-UFD1 AAA ATPase complex /
Neddylation / flavin adenine dinucleotide catabolic process / positive regulation of oxidative phosphorylation / VCP-NSFL1C complex / endosome to lysosome transport via multivesicular body sorting pathway / protein-DNA covalent cross-linking repair / positive regulation of ubiquitin-dependent protein catabolic process / endoplasmic reticulum stress-induced pre-emptive quality control / cellular response to arsenite ion / BAT3 complex binding / Derlin-1 retrotranslocation complex / positive regulation of protein K63-linked deubiquitination / deubiquitinase activator activity / mitotic spindle disassembly / VCP-NPL4-UFD1 AAA ATPase complex /  aggresome assembly / NADH metabolic process / regulation of protein localization to chromatin /
aggresome assembly / NADH metabolic process / regulation of protein localization to chromatin /  vesicle-fusing ATPase /
vesicle-fusing ATPase /  : / stress granule disassembly / K48-linked polyubiquitin modification-dependent protein binding / positive regulation of mitochondrial membrane potential / negative regulation of protein localization to chromatin / ERAD pathway / ubiquitin-modified protein reader activity / retrograde protein transport, ER to cytosol /
: / stress granule disassembly / K48-linked polyubiquitin modification-dependent protein binding / positive regulation of mitochondrial membrane potential / negative regulation of protein localization to chromatin / ERAD pathway / ubiquitin-modified protein reader activity / retrograde protein transport, ER to cytosol /  regulation of aerobic respiration /
regulation of aerobic respiration /  ATPase complex / regulation of synapse organization / ubiquitin-specific protease binding / positive regulation of ATP biosynthetic process / ubiquitin-like protein ligase binding / autophagosome maturation / polyubiquitin modification-dependent protein binding /
ATPase complex / regulation of synapse organization / ubiquitin-specific protease binding / positive regulation of ATP biosynthetic process / ubiquitin-like protein ligase binding / autophagosome maturation / polyubiquitin modification-dependent protein binding /  DNA修復 / endoplasmic reticulum to Golgi vesicle-mediated transport / MHC class I protein binding / negative regulation of smoothened signaling pathway / interstrand cross-link repair / : / ATP metabolic process / Neutrophil degranulation /
DNA修復 / endoplasmic reticulum to Golgi vesicle-mediated transport / MHC class I protein binding / negative regulation of smoothened signaling pathway / interstrand cross-link repair / : / ATP metabolic process / Neutrophil degranulation /  proteasome complex /
proteasome complex /  lipid droplet / viral genome replication /
lipid droplet / viral genome replication /  ADP binding / proteasomal protein catabolic process / positive regulation of protein-containing complex assembly /
ADP binding / proteasomal protein catabolic process / positive regulation of protein-containing complex assembly /  オートファジー /
オートファジー /  オートファジー / cytoplasmic stress granule / positive regulation of non-canonical NF-kappaB signal transduction / positive regulation of canonical Wnt signaling pathway / positive regulation of protein catabolic process / double-strand break repair / positive regulation of proteasomal ubiquitin-dependent protein catabolic process / site of double-strand break /
オートファジー / cytoplasmic stress granule / positive regulation of non-canonical NF-kappaB signal transduction / positive regulation of canonical Wnt signaling pathway / positive regulation of protein catabolic process / double-strand break repair / positive regulation of proteasomal ubiquitin-dependent protein catabolic process / site of double-strand break /  髄鞘 / cellular response to heat / ubiquitin-dependent protein catabolic process / proteasome-mediated ubiquitin-dependent protein catabolic process /
髄鞘 / cellular response to heat / ubiquitin-dependent protein catabolic process / proteasome-mediated ubiquitin-dependent protein catabolic process /  protein phosphatase binding / protein ubiquitination / protein domain specific binding /
protein phosphatase binding / protein ubiquitination / protein domain specific binding /  DNA修復 / glutamatergic synapse /
DNA修復 / glutamatergic synapse /  lipid binding /
lipid binding /  シナプス / DNA damage response /
シナプス / DNA damage response /  ubiquitin protein ligase binding / protein-containing complex binding / endoplasmic reticulum membrane / perinuclear region of cytoplasm /
ubiquitin protein ligase binding / protein-containing complex binding / endoplasmic reticulum membrane / perinuclear region of cytoplasm /  小胞体 /
小胞体 /  ATP hydrolysis activity / protein-containing complex /
ATP hydrolysis activity / protein-containing complex /  核質 /
核質 /  ATP binding / identical protein binding /
ATP binding / identical protein binding /  細胞核 /
細胞核 /  細胞質基質 /
細胞質基質 /  細胞質
細胞質
 Mus musculus (ハツカネズミ)
Mus musculus (ハツカネズミ) 単粒子再構成法 /
単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 20.0 Å
クライオ電子顕微鏡法 / 解像度: 20.0 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Open Biol / 年: 2014
ジャーナル: Open Biol / 年: 2014
 構造の表示
構造の表示 ムービービューア
ムービービューア SurfView
SurfView Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク emd_2592.map.gz
emd_2592.map.gz EMDBマップデータ形式
EMDBマップデータ形式 emd-2592-v30.xml
emd-2592-v30.xml emd-2592.xml
emd-2592.xml EMDBヘッダ
EMDBヘッダ emd_2592.jpg
emd_2592.jpg http://ftp.pdbj.org/pub/emdb/structures/EMD-2592
http://ftp.pdbj.org/pub/emdb/structures/EMD-2592 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-2592
ftp://ftp.pdbj.org/pub/emdb/structures/EMD-2592 リンク
リンク EMDB (EBI/PDBe) /
EMDB (EBI/PDBe) /  EMDataResource
EMDataResource マップ
マップ ダウンロード / ファイル: emd_2592.map.gz / 形式: CCP4 / 大きさ: 3.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES)
ダウンロード / ファイル: emd_2592.map.gz / 形式: CCP4 / 大きさ: 3.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) 試料の構成要素
試料の構成要素
 Mus musculus (ハツカネズミ) / 別称: House mouse
Mus musculus (ハツカネズミ) / 別称: House mouse
 Escherichia coli BL21(DE3) (大腸菌) / 組換プラスミド: pET28
Escherichia coli BL21(DE3) (大腸菌) / 組換プラスミド: pET28 クライオ電子顕微鏡法
クライオ電子顕微鏡法 解析
解析 単粒子再構成法
単粒子再構成法 試料調製
試料調製 電子顕微鏡法
電子顕微鏡法 FIELD EMISSION GUN
FIELD EMISSION GUN Bright-field microscopy / Cs: 2.1 mm
Bright-field microscopy / Cs: 2.1 mm 画像解析
画像解析 ) / アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 20.0 Å / 解像度の算出法: OTHER / ソフトウェア - 名称: Imagic / 使用した粒子像数: 30300
) / アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 20.0 Å / 解像度の算出法: OTHER / ソフトウェア - 名称: Imagic / 使用した粒子像数: 30300  ムービー
ムービー コントローラー
コントローラー