+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-2201 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Characterization of the insertase for beta-barrel proteins of the outer mitochondrial membrane. 3-D reconstruction of the TOB complex | |||||||||
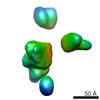
 マップデータ マップデータ | 3D reconstruction of TOB complexes isolated using the 9xHis tag on the Tob55 subunit. The TOB complex is a hetero trimer containing one copy each of Tob37, Tob38 and Tob37. Total mol. Wgt. Is 137 kDa. Contains only the short isoform of Tob55 since the his tag is on the short form. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | TOB/SAM complex /  beta-barrel proteins (Βバレル) / Tob55/Sam50 / mitochondria outer membrane beta-barrel proteins (Βバレル) / Tob55/Sam50 / mitochondria outer membrane | |||||||||
| 生物種 |   Neurospora crassa (アカパンカビ) Neurospora crassa (アカパンカビ) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 14.7 Å クライオ電子顕微鏡法 / 解像度: 14.7 Å | |||||||||
 データ登録者 データ登録者 | Klein A / Israel L / Lackey SWK / Nargang FE / Imhof A / Baumeister W / Neupert W / Thomas DR | |||||||||
 引用 引用 |  ジャーナル: J Cell Biol / 年: 2012 ジャーナル: J Cell Biol / 年: 2012タイトル: Characterization of the insertase for β-barrel proteins of the outer mitochondrial membrane. 著者: Astrid Klein / Lars Israel / Sebastian W K Lackey / Frank E Nargang / Axel Imhof / Wolfgang Baumeister / Walter Neupert / Dennis R Thomas /  要旨: The TOB-SAM complex is an essential component of the mitochondrial outer membrane that mediates the insertion of β-barrel precursor proteins into the membrane. We report here its isolation and ...The TOB-SAM complex is an essential component of the mitochondrial outer membrane that mediates the insertion of β-barrel precursor proteins into the membrane. We report here its isolation and determine its size, composition, and structural organization. The complex from Neurospora crassa was composed of Tob55-Sam50, Tob38-Sam35, and Tob37-Sam37 in a stoichiometry of 1:1:1 and had a molecular mass of 140 kD. A very minor fraction of the purified complex was associated with one Mdm10 protein. Using molecular homology modeling for Tob55 and cryoelectron microscopy reconstructions of the TOB complex, we present a model of the TOB-SAM complex that integrates biochemical and structural data. We discuss our results and the structural model in the context of a possible mechanism of the TOB insertase. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_2201.map.gz emd_2201.map.gz | 9.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-2201-v30.xml emd-2201-v30.xml emd-2201.xml emd-2201.xml | 12.3 KB 12.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  EMD-2201.jpg EMD-2201.jpg | 22 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-2201 http://ftp.pdbj.org/pub/emdb/structures/EMD-2201 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-2201 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-2201 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_2201.map.gz / 形式: CCP4 / 大きさ: 10.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_2201.map.gz / 形式: CCP4 / 大きさ: 10.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | 3D reconstruction of TOB complexes isolated using the 9xHis tag on the Tob55 subunit. The TOB complex is a hetero trimer containing one copy each of Tob37, Tob38 and Tob37. Total mol. Wgt. Is 137 kDa. Contains only the short isoform of Tob55 since the his tag is on the short form. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.78 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : TOB complex mitochondrial outer membrane complex required for ins...
| 全体 | 名称: TOB complex mitochondrial outer membrane complex required for inserting beta-barrel proteins into the outer membrane. |
|---|---|
| 要素 |
|
-超分子 #1000: TOB complex mitochondrial outer membrane complex required for ins...
| 超分子 | 名称: TOB complex mitochondrial outer membrane complex required for inserting beta-barrel proteins into the outer membrane. タイプ: sample / ID: 1000 / 詳細: The complexes were monodisperse. 集合状態: heterotrimer with one subunit each Tob37,Tob38 and Tob55 Number unique components: 3 |
|---|---|
| 分子量 | 実験値: 140 KDa / 理論値: 140 KDa 手法: Blue native gel electrophoresis and Isotope dilution mass spectroscopy analysis of bands isolated from BNGE gels. |
-分子 #1: Tob37
| 分子 | 名称: Tob37 / タイプ: protein_or_peptide / ID: 1 詳細: The TOB complex contains one copy each of T0b37, Tob38 and Tob55 コピー数: 1 / 集合状態: monomer / 組換発現: No |
|---|---|
| 由来(天然) | 生物種:   Neurospora crassa (アカパンカビ) / 株: Tob55 short HT / Organelle: mitochondria / 細胞中の位置: outer membrane Neurospora crassa (アカパンカビ) / 株: Tob55 short HT / Organelle: mitochondria / 細胞中の位置: outer membrane |
| 分子量 | 実験値: 48.6 KDa / 理論値: 48.6 KDa |
-分子 #2: Tob38
| 分子 | 名称: Tob38 / タイプ: protein_or_peptide / ID: 2 詳細: The TOB complex contains one copy each of T0b37, Tob38 and Tob55 コピー数: 1 / 集合状態: monomer / 組換発現: No |
|---|---|
| 由来(天然) | 生物種:   Neurospora crassa (アカパンカビ) / 株: Tob55 short HT / Organelle: mitochondria / 細胞中の位置: outer membrane Neurospora crassa (アカパンカビ) / 株: Tob55 short HT / Organelle: mitochondria / 細胞中の位置: outer membrane |
| 分子量 | 実験値: 37.3 KDa / 理論値: 37.3 KDa |
-分子 #3: Tob55
| 分子 | 名称: Tob55 / タイプ: protein_or_peptide / ID: 3 詳細: The TOB complex contains one copy each of T0b37, Tob38 and Tob55 コピー数: 1 / 集合状態: monomer / 組換発現: No |
|---|---|
| 由来(天然) | 生物種:   Neurospora crassa (アカパンカビ) / 株: Tob55 short HT / Organelle: mitochondria / 細胞中の位置: outer membrane Neurospora crassa (アカパンカビ) / 株: Tob55 short HT / Organelle: mitochondria / 細胞中の位置: outer membrane |
| 分子量 | 実験値: 54.7 KDa / 理論値: 54.7 KDa |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
- 試料調製
試料調製
| 濃度 | 1 mg/mL |
|---|---|
| 緩衝液 | pH: 8.5 詳細: 1mM PMSF, 0.08%(v/v) Triton X-100, 50 mM HEPES pH 8.5 |
| グリッド | 詳細: lacey carbon films on 200 mesh Molybdenum grids |
| 凍結 | 凍結剤: ETHANE / 装置: HOMEMADE PLUNGER 手法: blot for 4-5 seconds before plunging with whatman filter paper #1 |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI F20 |
|---|---|
| 電子線 | 加速電圧: 120 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 84270 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): -3.7 µm / 最小 デフォーカス(公称値): -0.5 µm / 倍率(公称値): 62000 Bright-field microscopy / 最大 デフォーカス(公称値): -3.7 µm / 最小 デフォーカス(公称値): -0.5 µm / 倍率(公称値): 62000 |
| 試料ステージ | 試料ホルダー: Gatan 656 side entry holder / 試料ホルダーモデル: GATAN LIQUID NITROGEN |
| アライメント法 | Legacy - 非点収差: Objective lens astigmatism was corrected using the live FFT at imaging magnification. |
| 日付 | 2010年5月13日 |
| 撮影 | カテゴリ: CCD / フィルム・検出器のモデル: FEI EAGLE (4k x 4k) / 実像数: 215 / 平均電子線量: 20 e/Å2 詳細: Images collected using TOM_acquisition software. 215 good micrographs were CTF corrected for phase. 80,000 particles were automatically selected. In the end 40600 were included in the final reconstruction. |
| Tilt angle min | 0 |
| Tilt angle max | 0 |
| 実験機器 |  モデル: Tecnai F20 / 画像提供: FEI Company |
- 画像解析
画像解析
| CTF補正 | 詳細: Phase and astigmatism correction applied to each micrograph |
|---|---|
| 最終 再構成 | 想定した対称性 - 点群: C1 (非対称) / アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 14.7 Å / 解像度の算出法: FSC 0.5 CUT-OFF / ソフトウェア - 名称: SPIDER, TOM_toolbox 詳細: Final maps were reconstructed from images that had stable alignment parameters over the last 4 rounds of refinement. Stable was defined by absolute accumulated changes in theta and psi of the ...詳細: Final maps were reconstructed from images that had stable alignment parameters over the last 4 rounds of refinement. Stable was defined by absolute accumulated changes in theta and psi of the projection matched of less than 10 degrees. 使用した粒子像数: 1 |
 ムービー
ムービー コントローラー
コントローラー