+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-2031 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
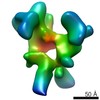
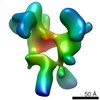
| タイトル | Unsymmetrized reconstruction of the FLNa16-21 rod2 segment | |||||||||
 マップデータ マップデータ | This is an unsymmetrized reconstruction of the filamin rod2 segment comprising domains 16-21 | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード |  cell adhesion (細胞接着) / integrin signaling / cell adhesion (細胞接着) / integrin signaling /  signal transduction (シグナル伝達) / signal transduction (シグナル伝達) /  single particle (単粒子解析法) single particle (単粒子解析法) | |||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 15.6 Å クライオ電子顕微鏡法 / 解像度: 15.6 Å | |||||||||
 データ登録者 データ登録者 | Hofmann GW / Jiang P / Campbell ID / Gilbert RJC | |||||||||
 引用 引用 |  ジャーナル: Biochem J / 年: 2012 ジャーナル: Biochem J / 年: 2012タイトル: The C-terminal rod 2 fragment of filamin A forms a compact structure that can be extended. 著者: Salla Ruskamo / Robert Gilbert / Gregor Hofmann / Pengju Jiang / Iain D Campbell / Jari Ylänne / Ulla Pentikäinen /  要旨: Filamins are large proteins that cross-link actin filaments and connect to other cellular components. The C-terminal rod 2 region of FLNa (filamin A) mediates dimerization and interacts with several ...Filamins are large proteins that cross-link actin filaments and connect to other cellular components. The C-terminal rod 2 region of FLNa (filamin A) mediates dimerization and interacts with several transmembrane receptors and intracellular signalling adaptors. SAXS (small-angle X-ray scattering) experiments were used to make a model of a six immunoglobulin-like domain fragment of the FLNa rod 2 (domains 16-21). This fragment had a surprising three-branched structural arrangement, where each branch was made of a tightly packed two-domain pair. Peptides derived from transmembrane receptors and intracellular signalling proteins induced a more open structure of the six domain fragment. Mutagenesis studies suggested that these changes are caused by peptides binding to the CD faces on domains 19 and 21 which displace the preceding domain A-strands (18 and 20 respectively), thus opening the individual domain pairs. A single particle cryo-EM map of a nine domain rod 2 fragment (domains 16-24), showed a relatively compact dimeric particle and confirmed the three-branched arrangement as well as the peptide-induced conformation changes. These findings reveal features of filamin structure that are important for its interactions and mechanical properties. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_2031.map.gz emd_2031.map.gz | 361.2 KB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-2031-v30.xml emd-2031-v30.xml emd-2031.xml emd-2031.xml | 9.8 KB 9.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  EMD-2031.png EMD-2031.png | 55.7 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-2031 http://ftp.pdbj.org/pub/emdb/structures/EMD-2031 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-2031 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-2031 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_2031.map.gz / 形式: CCP4 / 大きさ: 3.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_2031.map.gz / 形式: CCP4 / 大きさ: 3.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | This is an unsymmetrized reconstruction of the filamin rod2 segment comprising domains 16-21 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.37 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Filamin A rod-2 segment, comprising domains 16-21
| 全体 | 名称: Filamin A rod-2 segment, comprising domains 16-21 |
|---|---|
| 要素 |
|
-超分子 #1000: Filamin A rod-2 segment, comprising domains 16-21
| 超分子 | 名称: Filamin A rod-2 segment, comprising domains 16-21 / タイプ: sample / ID: 1000 / 集合状態: Dimer / Number unique components: 1 |
|---|---|
| 分子量 | 実験値: 200 KDa / 理論値: 200 KDa |
-分子 #1: FLNa16-21
| 分子 | 名称: FLNa16-21 / タイプ: protein_or_peptide / ID: 1 / Name.synonym: Filamin A rod 2 詳細: The sample was imaged as a monodisperse distribution in vitreous ice コピー数: 2 / 集合状態: Dimer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) / 別称: Human / 細胞中の位置: Cytoplasm (adjacent plasma membrane) Homo sapiens (ヒト) / 別称: Human / 細胞中の位置: Cytoplasm (adjacent plasma membrane) |
| 分子量 | 実験値: 200 KDa / 理論値: 200 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) / 組換プラスミド: pGEX Escherichia coli (大腸菌) / 組換プラスミド: pGEX |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.1 mg/mL |
|---|---|
| グリッド | 詳細: 300 mesh copper with lacey carbon |
| 凍結 | 凍結剤: ETHANE / 装置: HOMEMADE PLUNGER / 詳細: Vitrification instrument: Home made guillotine 手法: Whatman No. 1 paper blot for 1-2 seconds prior to plunging. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI F30 |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2 mm / 最大 デフォーカス(公称値): 4.0 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 59000 Bright-field microscopy / Cs: 2 mm / 最大 デフォーカス(公称値): 4.0 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 59000 |
| 試料ステージ | 試料ホルダー: Eucentric / 試料ホルダーモデル: GATAN LIQUID NITROGEN |
| 温度 | 平均: 110 K |
| アライメント法 | Legacy - 非点収差: At 112,000 times magnification |
| 日付 | 2006年9月1日 |
| 撮影 | カテゴリ: FILM / フィルム・検出器のモデル: KODAK SO-163 FILM / デジタル化 - スキャナー: ZEISS SCAI / デジタル化 - サンプリング間隔: 7 µm / 実像数: 29 / Od range: 5 / ビット/ピクセル: 8 |
| Tilt angle min | 0 |
| Tilt angle max | 0 |
| 実験機器 |  モデル: Tecnai F30 / 画像提供: FEI Company |
- 画像解析
画像解析
| CTF補正 | 詳細: per micrograph |
|---|---|
| 最終 角度割当 | 詳細: SPIDER Euler angles. |
| 最終 再構成 | 想定した対称性 - 点群: C1 (非対称) / アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 15.6 Å / 解像度の算出法: FSC 0.5 CUT-OFF / ソフトウェア - 名称: IMAGIC, SPIDER 詳細: The fineness of sampling was restricted to 15 degrees to reflect inaccuracy of angle determination at this particle size at finer spacings. The resolution of the map was therefore fixed at 30 Angstrom. 使用した粒子像数: 21133 |
| 詳細 | The raw images were treated with a three-level decomposition with a fourth-order bi-orthogonal wavelet function in MATLAB to enhance contrast. Particles were selected semi-automatically using Boxer. |
 ムービー
ムービー コントローラー
コントローラー