+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-1894 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
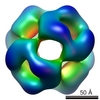
| タイトル | Cryo-EM structure of alphaB-crystallin 24mer | |||||||||
 マップデータ マップデータ | Volume map of human alphaB-crystallin 24mer | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | cryo electron microscopy / alphaB crystallin /  molecular chaperone (シャペロン) / molecular chaperone (シャペロン) /  protein aggregation (タンパク質凝集) / small heat shock protein / hybrid method protein aggregation (タンパク質凝集) / small heat shock protein / hybrid method | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報microtubule polymerization or depolymerization / negative regulation of intracellular transport / apoptotic process involved in morphogenesis / cardiac myofibril /  regulation of programmed cell death / tubulin complex assembly / structural constituent of eye lens / negative regulation of amyloid fibril formation / regulation of programmed cell death / tubulin complex assembly / structural constituent of eye lens / negative regulation of amyloid fibril formation /  M band / lens development in camera-type eye ...microtubule polymerization or depolymerization / negative regulation of intracellular transport / apoptotic process involved in morphogenesis / cardiac myofibril / M band / lens development in camera-type eye ...microtubule polymerization or depolymerization / negative regulation of intracellular transport / apoptotic process involved in morphogenesis / cardiac myofibril /  regulation of programmed cell death / tubulin complex assembly / structural constituent of eye lens / negative regulation of amyloid fibril formation / regulation of programmed cell death / tubulin complex assembly / structural constituent of eye lens / negative regulation of amyloid fibril formation /  M band / lens development in camera-type eye / muscle organ development / actin filament bundle / HSF1-dependent transactivation / negative regulation of reactive oxygen species metabolic process / negative regulation of protein-containing complex assembly / stress-activated MAPK cascade / M band / lens development in camera-type eye / muscle organ development / actin filament bundle / HSF1-dependent transactivation / negative regulation of reactive oxygen species metabolic process / negative regulation of protein-containing complex assembly / stress-activated MAPK cascade /  synaptic membrane / synaptic membrane /  muscle contraction / cellular response to gamma radiation / response to hydrogen peroxide / negative regulation of cell growth / Z disc / unfolded protein binding / muscle contraction / cellular response to gamma radiation / response to hydrogen peroxide / negative regulation of cell growth / Z disc / unfolded protein binding /  フォールディング / response to estradiol / フォールディング / response to estradiol /  amyloid-beta binding / response to heat / amyloid-beta binding / response to heat /  perikaryon / protein refolding / perikaryon / protein refolding /  microtubule binding / microtubule binding /  樹状突起スパイン / 樹状突起スパイン /  リソソーム / response to hypoxia / protein stabilization / リソソーム / response to hypoxia / protein stabilization /  神経繊維 / negative regulation of gene expression / negative regulation of DNA-templated transcription / protein-containing complex binding / negative regulation of apoptotic process / structural molecule activity / 神経繊維 / negative regulation of gene expression / negative regulation of DNA-templated transcription / protein-containing complex binding / negative regulation of apoptotic process / structural molecule activity /  細胞膜 / protein homodimerization activity / protein-containing complex / 細胞膜 / protein homodimerization activity / protein-containing complex /  ミトコンドリア / extracellular exosome / ミトコンドリア / extracellular exosome /  核質 / identical protein binding / 核質 / identical protein binding /  metal ion binding / metal ion binding /  細胞核 / 細胞核 /  細胞質基質 / 細胞質基質 /  細胞質 細胞質類似検索 - 分子機能 | |||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / クライオ電子顕微鏡法 /  ネガティブ染色法 / 解像度: 9.4 Å ネガティブ染色法 / 解像度: 9.4 Å | |||||||||
 データ登録者 データ登録者 | Braun N / Zacharias M / Peschek J / Kastenmueller A / Hanzlik M / Haslbeck M / Buchner J / Weinkauf S | |||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2011 ジャーナル: Proc Natl Acad Sci U S A / 年: 2011タイトル: Multiple molecular architectures of the eye lens chaperone αB-crystallin elucidated by a triple hybrid approach. 著者: Nathalie Braun / Martin Zacharias / Jirka Peschek / Andreas Kastenmüller / Juan Zou / Marianne Hanzlik / Martin Haslbeck / Juri Rappsilber / Johannes Buchner / Sevil Weinkauf /  要旨: The molecular chaperone αB-crystallin, the major player in maintaining the transparency of the eye lens, prevents stress-damaged and aging lens proteins from aggregation. In nonlenticular cells, it ...The molecular chaperone αB-crystallin, the major player in maintaining the transparency of the eye lens, prevents stress-damaged and aging lens proteins from aggregation. In nonlenticular cells, it is involved in various neurological diseases, diabetes, and cancer. Given its structural plasticity and dynamics, structure analysis of αB-crystallin presented hitherto a formidable challenge. Here we present a pseudoatomic model of a 24-meric αB-crystallin assembly obtained by a triple hybrid approach combining data from cryoelectron microscopy, NMR spectroscopy, and structural modeling. The model, confirmed by cross-linking and mass spectrometry, shows that the subunits interact within the oligomer in different, defined conformations. We further present the molecular architectures of additional well-defined αB-crystallin assemblies with larger or smaller numbers of subunits, provide the mechanism how "heterogeneity" is achieved by a small set of defined structural variations, and analyze the factors modulating the oligomer equilibrium of αB-crystallin and thus its chaperone activity. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_1894.map.gz emd_1894.map.gz | 428.8 KB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-1894-v30.xml emd-1894-v30.xml emd-1894.xml emd-1894.xml | 9.9 KB 9.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  1894_1894.jpg 1894_1894.jpg | 106.7 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-1894 http://ftp.pdbj.org/pub/emdb/structures/EMD-1894 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1894 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1894 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_1894.map.gz / 形式: CCP4 / 大きさ: 7.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_1894.map.gz / 形式: CCP4 / 大きさ: 7.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Volume map of human alphaB-crystallin 24mer | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.8 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Human alphaB crystallin
| 全体 | 名称: Human alphaB crystallin |
|---|---|
| 要素 |
|
-超分子 #1000: Human alphaB crystallin
| 超分子 | 名称: Human alphaB crystallin / タイプ: sample / ID: 1000 / 集合状態: Tetrahedral 24mer / Number unique components: 1 |
|---|---|
| 分子量 | 実験値: 485 KDa / 理論値: 485 KDa |
-分子 #1: AlphaB crystallin
| 分子 | 名称: AlphaB crystallin / タイプ: protein_or_peptide / ID: 1 / Name.synonym: Human alphaB crystallin / コピー数: 24 / 集合状態: 24mer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) / 別称: Human Homo sapiens (ヒト) / 別称: Human |
| 分子量 | 理論値: 485 KDa |
| 組換発現 | 生物種: Echerichia coli / 組換プラスミド: pET28b |
| 配列 | InterPro: INTERPRO: IPR012273 |
-実験情報
-構造解析
| 手法 |  ネガティブ染色法, ネガティブ染色法,  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.2 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 / 詳細: PBS buffer |
| 染色 | タイプ: NEGATIVE / 詳細: Vitrification |
| グリッド | 詳細: Quantifoil multi A (400 mesh copper grid) |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 50 % / 装置: OTHER / 手法: Blot for 1 second before plunging |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | JEOL 2010HT |
|---|---|
| 電子線 | 加速電圧: 120 kV / 電子線源: LAB6 |
| 電子光学系 | 倍率(補正後): 47000 / 照射モード: SPOT SCAN / 撮影モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 1.5 µm / 最小 デフォーカス(公称値): 0.6 µm / 倍率(公称値): 50000 Bright-field microscopy / 最大 デフォーカス(公称値): 1.5 µm / 最小 デフォーカス(公称値): 0.6 µm / 倍率(公称値): 50000 |
| 試料ステージ | 試料ホルダー: Eucentric / 試料ホルダーモデル: GATAN LIQUID NITROGEN |
| 温度 | 平均: 100 K |
| 撮影 | デジタル化 - スキャナー: OTHER / デジタル化 - サンプリング間隔: 8.47 µm / 実像数: 33 / 平均電子線量: 10 e/Å2 |
- 画像解析
画像解析
| CTF補正 | 詳細: Each micrograph, phase flipping |
|---|---|
| 最終 2次元分類 | クラス数: 878 |
| 最終 再構成 | 想定した対称性 - 点群: T (正4面体型対称 ) / アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 9.4 Å / 解像度の算出法: FSC 0.5 CUT-OFF / ソフトウェア - 名称: Imagic / 使用した粒子像数: 17560 ) / アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 9.4 Å / 解像度の算出法: FSC 0.5 CUT-OFF / ソフトウェア - 名称: Imagic / 使用した粒子像数: 17560 |
-原子モデル構築 1
| 初期モデル | PDB ID: |
|---|---|
| 詳細 | PDB ID 2KLR was used for residues 69-150. Structure-elements for the remaining C- and N-termini were obtained by the found EM-Structure and structure prediction tools (fitted to the EM-Volume) |
| 精密化 | 空間: REAL |
| 得られたモデル |  PDB-2ygd: |
 ムービー
ムービー コントローラー
コントローラー