+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cysteine tRNA ligase homodimer | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード |  cysteine (システイン) / cysteine (システイン) /  trna (転移RNA) / trna (転移RNA) /  ligase (リガーゼ) / synthetase / ligase (リガーゼ) / synthetase /  homodimer / homodimer /  RNA (リボ核酸) RNA (リボ核酸) | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 システインtRNAリガーゼ / システインtRNAリガーゼ /  cysteine-tRNA ligase activity / cysteinyl-tRNA aminoacylation / zinc ion binding / cysteine-tRNA ligase activity / cysteinyl-tRNA aminoacylation / zinc ion binding /  ATP binding / ATP binding /  細胞質 細胞質類似検索 - 分子機能 | |||||||||
| 生物種 |    Pyrococcus furiosus DSM 3638 (ピュロコックス・フリオスス) Pyrococcus furiosus DSM 3638 (ピュロコックス・フリオスス) | |||||||||
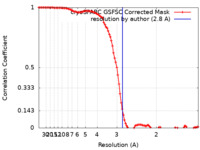
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 2.8 Å クライオ電子顕微鏡法 / 解像度: 2.8 Å | |||||||||
 データ登録者 データ登録者 | Pacesa M / Correia BE / Levy ED | |||||||||
| 資金援助 | European Union, 1件
| |||||||||
 引用 引用 |  ジャーナル: Cell / 年: 2024 ジャーナル: Cell / 年: 2024タイトル: An atlas of protein homo-oligomerization across domains of life. 著者: Hugo Schweke / Martin Pacesa / Tal Levin / Casper A Goverde / Prasun Kumar / Yoan Duhoo / Lars J Dornfeld / Benjamin Dubreuil / Sandrine Georgeon / Sergey Ovchinnikov / Derek N Woolfson / ...著者: Hugo Schweke / Martin Pacesa / Tal Levin / Casper A Goverde / Prasun Kumar / Yoan Duhoo / Lars J Dornfeld / Benjamin Dubreuil / Sandrine Georgeon / Sergey Ovchinnikov / Derek N Woolfson / Bruno E Correia / Sucharita Dey / Emmanuel D Levy /      要旨: Protein structures are essential to understanding cellular processes in molecular detail. While advances in artificial intelligence revealed the tertiary structure of proteins at scale, their ...Protein structures are essential to understanding cellular processes in molecular detail. While advances in artificial intelligence revealed the tertiary structure of proteins at scale, their quaternary structure remains mostly unknown. We devise a scalable strategy based on AlphaFold2 to predict homo-oligomeric assemblies across four proteomes spanning the tree of life. Our results suggest that approximately 45% of an archaeal proteome and a bacterial proteome and 20% of two eukaryotic proteomes form homomers. Our predictions accurately capture protein homo-oligomerization, recapitulate megadalton complexes, and unveil hundreds of homo-oligomer types, including three confirmed experimentally by structure determination. Integrating these datasets with omics information suggests that a majority of known protein complexes are symmetric. Finally, these datasets provide a structural context for interpreting disease mutations and reveal coiled-coil regions as major enablers of quaternary structure evolution in human. Our strategy is applicable to any organism and provides a comprehensive view of homo-oligomerization in proteomes. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_18415.map.gz emd_18415.map.gz | 51.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-18415-v30.xml emd-18415-v30.xml emd-18415.xml emd-18415.xml | 17 KB 17 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_18415_fsc.xml emd_18415_fsc.xml | 9.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_18415.png emd_18415.png | 65.9 KB | ||
| Filedesc metadata |  emd-18415.cif.gz emd-18415.cif.gz | 6 KB | ||
| その他 |  emd_18415_half_map_1.map.gz emd_18415_half_map_1.map.gz emd_18415_half_map_2.map.gz emd_18415_half_map_2.map.gz | 95.5 MB 95.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-18415 http://ftp.pdbj.org/pub/emdb/structures/EMD-18415 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-18415 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-18415 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_18415.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_18415.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 0.726 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #1
| ファイル | emd_18415_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
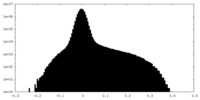
| 投影像・断面図 |
| ||||||||||||
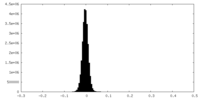
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_18415_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
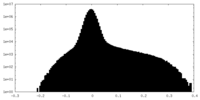
| 投影像・断面図 |
| ||||||||||||
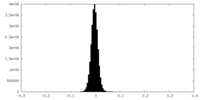
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Homodimeric form of Cysteine tRNA ligase
| 全体 | 名称: Homodimeric form of Cysteine tRNA ligase |
|---|---|
| 要素 |
|
-超分子 #1: Homodimeric form of Cysteine tRNA ligase
| 超分子 | 名称: Homodimeric form of Cysteine tRNA ligase / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:    Pyrococcus furiosus DSM 3638 (ピュロコックス・フリオスス) Pyrococcus furiosus DSM 3638 (ピュロコックス・フリオスス) |
| 分子量 | 理論値: 114 KDa |
-分子 #1: Cysteine--tRNA ligase
| 分子 | 名称: Cysteine--tRNA ligase / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:    Pyrococcus furiosus DSM 3638 (ピュロコックス・フリオスス) Pyrococcus furiosus DSM 3638 (ピュロコックス・フリオスス) |
| 分子量 | 理論値: 57.558605 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: MKHHHHHHGG SSGGLKVYNT LTKQKEEFKP LREGEVKMYV CGPTVYDYPH LGHARTYIAF DVIRRYLEHK GYTVLMVMNF TDIDDKIIK RARETGEDPK ELAERFIKIF LEDMEALKVK PADIYPRVTD HIDDIIEFIG KLKEKGYAYE GSDGIYFEVK K FPEYGKLS ...文字列: MKHHHHHHGG SSGGLKVYNT LTKQKEEFKP LREGEVKMYV CGPTVYDYPH LGHARTYIAF DVIRRYLEHK GYTVLMVMNF TDIDDKIIK RARETGEDPK ELAERFIKIF LEDMEALKVK PADIYPRVTD HIDDIIEFIG KLKEKGYAYE GSDGIYFEVK K FPEYGKLS GVKIEDLQKG ARVEPGEGKK NPEDFALWKK AKPGEPKWDS PWGEGRPGWH IECSVMSSKY LGESFDIHGG GN DLIFPHH ENEIAQSEAC FGHEWVKYWL HTGFVMVKGE KMSKSLGNFV TIRELLKRYE PEVIRFFVLQ KHYRSPLEYT EEG LQHAKN NLQRLYNTLE NIRVALRNAE ISYTWGELEF KTYEIIREGK RKFYEAMDDD FNTAEALKAV FEVANAINKY LTEA NKPKE SILRKALEFF KIVSEVFGVF EDYFREETKE REESEKLIEL LVEVRKQLRK EKRYELADMI REELKKLGIQ LEDRG SETT WKRIIT UniProtKB: Cysteine--tRNA ligase |
-分子 #2: ZINC ION
| 分子 | 名称: ZINC ION / タイプ: ligand / ID: 2 / コピー数: 2 / 式: ZN |
|---|---|
| 分子量 | 理論値: 65.409 Da |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 60 sec. |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / チャンバー内温度: 283.15 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 0.8 µm Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 0.8 µm |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 温度 | 最低: 186.0 K / 最高: 192.0 K |
| 撮影 | フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) デジタル化 - サイズ - 横: 4096 pixel / デジタル化 - サイズ - 縦: 4096 pixel / 平均電子線量: 60.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | Chain - Source name: AlphaFold / Chain - Initial model type: in silico model |
|---|---|
| 精密化 | 空間: REAL / プロトコル: AB INITIO MODEL |
| 得られたモデル |  PDB-8qhp: |
 ムービー
ムービー コントローラー
コントローラー








 Z
Z Y
Y X
X