[日本語] English
 万見
万見- EMDB-18247: Outer kinetochore Dam1 protomer monomer Ndc80-Nuf2 coiled-coil complex -
+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | 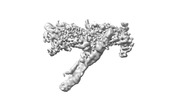 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Outer kinetochore Dam1 protomer monomer Ndc80-Nuf2 coiled-coil complex | |||||||||
 マップデータ マップデータ | Local resolution filtered map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード |  Kinetochore (動原体) / Kinetochore (動原体) /  microtubule (微小管) / error correction / microtubule (微小管) / error correction /  chromosome segregation / chromosome segregation /  CELL CYCLE (細胞周期) CELL CYCLE (細胞周期) | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報mitotic spindle polar microtubule /  Ndc80 complex / DASH complex / protein transport along microtubule to mitotic spindle pole body / mitotic sister chromatid biorientation / kinetochore organization / positive regulation of attachment of spindle microtubules to kinetochore / mitotic spindle pole body / meiotic chromosome segregation / mitotic spindle midzone ...mitotic spindle polar microtubule / Ndc80 complex / DASH complex / protein transport along microtubule to mitotic spindle pole body / mitotic sister chromatid biorientation / kinetochore organization / positive regulation of attachment of spindle microtubules to kinetochore / mitotic spindle pole body / meiotic chromosome segregation / mitotic spindle midzone ...mitotic spindle polar microtubule /  Ndc80 complex / DASH complex / protein transport along microtubule to mitotic spindle pole body / mitotic sister chromatid biorientation / kinetochore organization / positive regulation of attachment of spindle microtubules to kinetochore / mitotic spindle pole body / meiotic chromosome segregation / mitotic spindle midzone / attachment of spindle microtubules to kinetochore / attachment of mitotic spindle microtubules to kinetochore / condensed chromosome, centromeric region / Ndc80 complex / DASH complex / protein transport along microtubule to mitotic spindle pole body / mitotic sister chromatid biorientation / kinetochore organization / positive regulation of attachment of spindle microtubules to kinetochore / mitotic spindle pole body / meiotic chromosome segregation / mitotic spindle midzone / attachment of spindle microtubules to kinetochore / attachment of mitotic spindle microtubules to kinetochore / condensed chromosome, centromeric region /  spindle pole body / protein localization to kinetochore / positive regulation of microtubule polymerization / mitotic spindle organization / spindle pole body / protein localization to kinetochore / positive regulation of microtubule polymerization / mitotic spindle organization /  chromosome segregation / spindle microtubule / chromosome segregation / spindle microtubule /  動原体 / 動原体 /  紡錘体 / 紡錘体 /  紡錘体 / 紡錘体 /  微小管 / 微小管 /  細胞分裂 / protein-containing complex binding / identical protein binding / 細胞分裂 / protein-containing complex binding / identical protein binding /  細胞核 / 細胞核 /  細胞質 細胞質類似検索 - 分子機能 | |||||||||
| 生物種 |   Saccharomyces cerevisiae (パン酵母) Saccharomyces cerevisiae (パン酵母) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.97 Å クライオ電子顕微鏡法 / 解像度: 3.97 Å | |||||||||
 データ登録者 データ登録者 | Muir KW / Barford D | |||||||||
| 資金援助 |  英国, 2件 英国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Science / 年: 2023 ジャーナル: Science / 年: 2023タイトル: Structural mechanism of outer kinetochore Dam1-Ndc80 complex assembly on microtubules. 著者: Kyle W Muir / Christopher Batters / Tom Dendooven / Jing Yang / Ziguo Zhang / Alister Burt / David Barford /  要旨: Kinetochores couple chromosomes to the mitotic spindle to segregate the genome during cell division. An error correction mechanism drives the turnover of kinetochore-microtubule attachments until ...Kinetochores couple chromosomes to the mitotic spindle to segregate the genome during cell division. An error correction mechanism drives the turnover of kinetochore-microtubule attachments until biorientation is achieved. The structural basis for how kinetochore-mediated chromosome segregation is accomplished and regulated remains an outstanding question. In this work, we describe the cryo-electron microscopy structure of the budding yeast outer kinetochore Ndc80 and Dam1 ring complexes assembled onto microtubules. Complex assembly occurs through multiple interfaces, and a staple within Dam1 aids ring assembly. Perturbation of key interfaces suppresses yeast viability. Force-rupture assays indicated that this is a consequence of impaired kinetochore-microtubule attachment. The presence of error correction phosphorylation sites at Ndc80-Dam1 ring complex interfaces and the Dam1 staple explains how kinetochore-microtubule attachments are destabilized and reset. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_18247.map.gz emd_18247.map.gz | 5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-18247-v30.xml emd-18247-v30.xml emd-18247.xml emd-18247.xml | 37.5 KB 37.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_18247.png emd_18247.png | 31.3 KB | ||
| Filedesc metadata |  emd-18247.cif.gz emd-18247.cif.gz | 8.2 KB | ||
| その他 |  emd_18247_additional_1.map.gz emd_18247_additional_1.map.gz emd_18247_additional_2.map.gz emd_18247_additional_2.map.gz emd_18247_additional_3.map.gz emd_18247_additional_3.map.gz emd_18247_additional_4.map.gz emd_18247_additional_4.map.gz emd_18247_half_map_1.map.gz emd_18247_half_map_1.map.gz emd_18247_half_map_2.map.gz emd_18247_half_map_2.map.gz | 50.6 MB 96.3 MB 95.7 MB 95.7 MB 95.7 MB 95.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-18247 http://ftp.pdbj.org/pub/emdb/structures/EMD-18247 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-18247 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-18247 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_18247.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_18247.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Local resolution filtered map | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.06 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: Homogeneous refinement map
| ファイル | emd_18247_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Homogeneous refinement map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
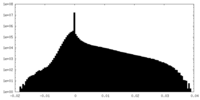
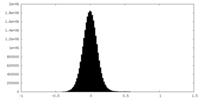
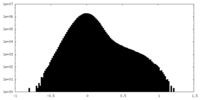
| 密度ヒストグラム |
-追加マップ: 3D Flex EM map
| ファイル | emd_18247_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | 3D Flex EM map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
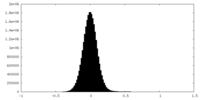
| 密度ヒストグラム |
-追加マップ: 3D Flex EM half map B
| ファイル | emd_18247_additional_3.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | 3D Flex EM half map B | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
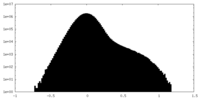
| 密度ヒストグラム |
-追加マップ: 3D Flex EM half map A
| ファイル | emd_18247_additional_4.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | 3D Flex EM half map A | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Homogeneous refinement half map B
| ファイル | emd_18247_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Homogeneous refinement half map B | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
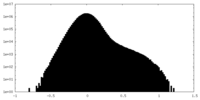
| 密度ヒストグラム |
-ハーフマップ: Homogeneous refinement half map A
| ファイル | emd_18247_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Homogeneous refinement half map A | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
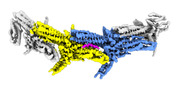
- 試料の構成要素
試料の構成要素
+全体 : Outer kinetochore Dam1 protomer monomer with staple and Ndc80-Nuf...
+超分子 #1: Outer kinetochore Dam1 protomer monomer with staple and Ndc80-Nuf...
+分子 #1: Kinetochore protein NDC80
+分子 #2: Kinetochore protein NUF2
+分子 #3: DASH complex subunit DAM1
+分子 #4: DASH complex subunit DUO1
+分子 #5: DASH complex subunit DAD2
+分子 #6: DASH complex subunit DAD1
+分子 #7: DASH complex subunit DAD4
+分子 #8: DASH complex subunit DAD3
+分子 #9: DASH complex subunit SPC34
+分子 #10: DASH complex subunit ASK1
+分子 #11: DASH complex subunit HSK3
+分子 #12: DASH complex subunit SPC19
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 6.8 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.2 µm Bright-field microscopy / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.2 µm |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 50.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: EMDB MAP EMDB ID: |
|---|---|
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD ソフトウェア: (名称: cryoSPARC (ver. 3.3.2), RELION (ver. 4.0-dev)) |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD / ソフトウェア - 名称: cryoSPARC (ver. 4.2) |
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 3.97 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 77272 |
-原子モデル構築 1
| 初期モデル | Chain - Source name: AlphaFold / Chain - Initial model type: in silico model |
|---|---|
| 詳細 | Initial rigid body fitting was performed in chimera, with manual correction in coot and real-space refinement in PHENIX |
| 精密化 | 空間: REAL / プロトコル: OTHER / 温度因子: 540.98 |
| 得られたモデル |  PDB-8q85: |
 ムービー
ムービー コントローラー
コントローラー






 Z
Z Y
Y X
X