+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-1397 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Multiple states of a nucleotide-bound group 2 chaperonin. | |||||||||
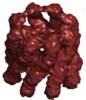
 マップデータ マップデータ | Reconstruction of the bullet conformation of the M. maripaludis group 2 chaperonin | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  ATP binding ATP binding 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |   Methanococcus maripaludis (古細菌) Methanococcus maripaludis (古細菌) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 9.5 Å クライオ電子顕微鏡法 / 解像度: 9.5 Å | |||||||||
 データ登録者 データ登録者 | Clare DK / Stagg S / Quispe J / Farr GW / Horwich AL / Saibil HR | |||||||||
 引用 引用 |  ジャーナル: Structure / 年: 2008 ジャーナル: Structure / 年: 2008タイトル: Multiple states of a nucleotide-bound group 2 chaperonin. 著者: Daniel K Clare / Scott Stagg / Joel Quispe / George W Farr / Arthur L Horwich / Helen R Saibil /  要旨: Chaperonin action is controlled by cycles of nucleotide binding and hydrolysis. Here, we examine the effects of nucleotide binding on an archaeal group 2 chaperonin. In contrast to the ordered apo ...Chaperonin action is controlled by cycles of nucleotide binding and hydrolysis. Here, we examine the effects of nucleotide binding on an archaeal group 2 chaperonin. In contrast to the ordered apo state of the group 1 chaperonin GroEL, the unliganded form of the homo-16-mer Methanococcus maripaludis group 2 chaperonin is very open and flexible, with intersubunit contacts only in the central double belt of equatorial domains. The intermediate and apical domains are free of contacts and deviate significantly from the overall 8-fold symmetry. Nucleotide binding results in three distinct, ordered 8-fold symmetric conformations--open, partially closed, and fully closed. The partially closed ring encloses a 40% larger volume than does the GroEL-GroES folding chamber, enabling it to encapsulate proteins up to 80 kDa, in contrast to the fully closed form, whose cavities are 20% smaller than those of the GroEL-GroES chamber. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_1397.map.gz emd_1397.map.gz | 173.7 KB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-1397-v30.xml emd-1397-v30.xml emd-1397.xml emd-1397.xml | 10.6 KB 10.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  1397.gif 1397.gif | 146.5 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-1397 http://ftp.pdbj.org/pub/emdb/structures/EMD-1397 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1397 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1397 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_1397.map.gz / 形式: CCP4 / 大きさ: 3.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_1397.map.gz / 形式: CCP4 / 大きさ: 3.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Reconstruction of the bullet conformation of the M. maripaludis group 2 chaperonin | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 3.26 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : cpn60
| 全体 | 名称: cpn60 |
|---|---|
| 要素 |
|
-超分子 #1000: cpn60
| 超分子 | 名称: cpn60 / タイプ: sample / ID: 1000 / 集合状態: hexadecamer / Number unique components: 2 |
|---|---|
| 分子量 | 実験値: 900 KDa / 理論値: 900 KDa |
-分子 #1: cpn60
| 分子 | 名称: cpn60 / タイプ: protein_or_peptide / ID: 1 / Name.synonym: cpn60 / 詳細: chaperonin / コピー数: 16 / 集合状態: hexadecamer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:   Methanococcus maripaludis (古細菌) / 株: LL / 別称: M. maripaludis / 細胞中の位置: cytoplasm Methanococcus maripaludis (古細菌) / 株: LL / 別称: M. maripaludis / 細胞中の位置: cytoplasm |
| 分子量 | 実験値: 900 KDa / 理論値: 900 KDa |
| 組換発現 | 生物種:  Escherichia coli BL21, DE3 (大腸菌) / 組換プラスミド: pET30b Escherichia coli BL21, DE3 (大腸菌) / 組換プラスミド: pET30b |
| 配列 | GO:  ATP binding ATP binding |
-分子 #2: ADPAlF3
| 分子 | 名称: ADPAlF3 / タイプ: ligand / ID: 2 / Name.synonym: ADPAlF3 / 詳細: ATP analogue / 組換発現: No |
|---|---|
| 分子量 | 実験値: 500 Da / 理論値: 500 Da |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 3 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 詳細: 50mM Tris-HCL, 150mM KCl, 10mM MgCl, 1mM DTT 1mM ADP, 5mMKF, 0.5mM Al |
| グリッド | 詳細: 400 mesh c2/2 |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 100 K / 装置: OTHER 詳細: Vitrification instrument: vitrobot. vitobot chamber was maintained at 37C 手法: 2 second blot |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI F20 |
|---|---|
| 電子線 | 加速電圧: 120 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 92000 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2 mm / 最大 デフォーカス(公称値): 3.4 µm / 最小 デフォーカス(公称値): 0.9 µm / 倍率(公称値): 50000 Bright-field microscopy / Cs: 2 mm / 最大 デフォーカス(公称値): 3.4 µm / 最小 デフォーカス(公称値): 0.9 µm / 倍率(公称値): 50000 |
| 試料ステージ | 試料ホルダー: Side entry single tilt holder / 試料ホルダーモデル: GATAN LIQUID NITROGEN |
| 温度 | 平均: 100 K |
| アライメント法 | Legacy - 非点収差: objective astigmatism corrected by leginon |
| 日付 | 2006年5月12日 |
| 撮影 | カテゴリ: CCD フィルム・検出器のモデル: GENERIC GATAN (4k x 4k) 実像数: 531 / 平均電子線量: 20 e/Å2 / 詳細: collected using the Leginon system at Scripps |
| 実験機器 |  モデル: Tecnai F20 / 画像提供: FEI Company |
- 画像解析
画像解析
| CTF補正 | 詳細: Each particle |
|---|---|
| 最終 角度割当 | 詳細: theta 80-100, phi 0-45 |
| 最終 再構成 | 想定した対称性 - 点群: C8 (8回回転対称 ) / アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 9.5 Å / 解像度の算出法: FSC 0.5 CUT-OFF / ソフトウェア - 名称: Spider / 使用した粒子像数: 3751 ) / アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 9.5 Å / 解像度の算出法: FSC 0.5 CUT-OFF / ソフトウェア - 名称: Spider / 使用した粒子像数: 3751 |
| 詳細 | We used FindEM to pick the particle |
-原子モデル構築 1
| ソフトウェア | 名称: URO |
|---|---|
| 詳細 | Protocol: rigid body. the domains were separately fitted using pymol and then their postions were refined using URO |
| 精密化 | 空間: RECIPROCAL / プロトコル: RIGID BODY FIT / 当てはまり具合の基準: correlation coefficient |
 ムービー
ムービー コントローラー
コントローラー










 Z (Sec.)
Z (Sec.) X (Row.)
X (Row.) Y (Col.)
Y (Col.)