+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Twist-corrected RNA origami 5-helix Tile A | |||||||||
 マップデータ マップデータ | Sharpened map with b factor of -252. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード |  RNA (リボ核酸) / RNA (リボ核酸) /  Origami (折り紙) / Origami (折り紙) /  nanostructure (ナノ構造体) nanostructure (ナノ構造体) | |||||||||
| 生物種 | synthetic construct (人工物) | |||||||||
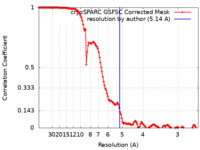
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 5.14 Å クライオ電子顕微鏡法 / 解像度: 5.14 Å | |||||||||
 データ登録者 データ登録者 | McRae EKS / Bogglid A / Boesen T / Andersen ES | |||||||||
| 資金援助 |  デンマーク, 1件 デンマーク, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Nanotechnol / 年: 2023 ジャーナル: Nat Nanotechnol / 年: 2023タイトル: Structure, folding and flexibility of co-transcriptional RNA origami. 著者: Ewan K S McRae / Helena Østergaard Rasmussen / Jianfang Liu / Andreas Bøggild / Michael T A Nguyen / Nestor Sampedro Vallina / Thomas Boesen / Jan Skov Pedersen / Gang Ren / Cody Geary / Ebbe Sloth Andersen /   要旨: RNA origami is a method for designing RNA nanostructures that can self-assemble through co-transcriptional folding with applications in nanomedicine and synthetic biology. However, to advance the ...RNA origami is a method for designing RNA nanostructures that can self-assemble through co-transcriptional folding with applications in nanomedicine and synthetic biology. However, to advance the method further, an improved understanding of RNA structural properties and folding principles is required. Here we use cryogenic electron microscopy to study RNA origami sheets and bundles at sub-nanometre resolution revealing structural parameters of kissing-loop and crossover motifs, which are used to improve designs. In RNA bundle designs, we discover a kinetic folding trap that forms during folding and is only released after 10 h. Exploration of the conformational landscape of several RNA designs reveal the flexibility of helices and structural motifs. Finally, sheets and bundles are combined to construct a multidomain satellite shape, which is characterized by individual-particle cryo-electron tomography to reveal the domain flexibility. Together, the study provides a structural basis for future improvements to the design cycle of genetically encoded RNA nanodevices. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_13926.map.gz emd_13926.map.gz | 220.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-13926-v30.xml emd-13926-v30.xml emd-13926.xml emd-13926.xml | 19.1 KB 19.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_13926_fsc.xml emd_13926_fsc.xml | 17.8 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_13926.png emd_13926.png | 40.8 KB | ||
| マスクデータ |  emd_13926_msk_1.map emd_13926_msk_1.map | 233.3 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-13926.cif.gz emd-13926.cif.gz | 5.8 KB | ||
| その他 |  emd_13926_half_map_1.map.gz emd_13926_half_map_1.map.gz emd_13926_half_map_2.map.gz emd_13926_half_map_2.map.gz | 216.8 MB 216.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-13926 http://ftp.pdbj.org/pub/emdb/structures/EMD-13926 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-13926 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-13926 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_13926.map.gz / 形式: CCP4 / 大きさ: 233.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_13926.map.gz / 形式: CCP4 / 大きさ: 233.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Sharpened map with b factor of -252. | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.294 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_13926_msk_1.map emd_13926_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map B
| ファイル | emd_13926_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map B | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map A
| ファイル | emd_13926_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map A | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Twist-corrected 5-helix tile A.
| 全体 | 名称: Twist-corrected 5-helix tile A. |
|---|---|
| 要素 |
|
-超分子 #1: Twist-corrected 5-helix tile A.
| 超分子 | 名称: Twist-corrected 5-helix tile A. / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all / 詳細: In vitro transcribed RNA purified by SEC. |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 175 KDa |
-分子 #1: Chains: Q
| 分子 | 名称: Chains: Q / タイプ: rna / ID: 1 / 詳細: In vitro transcribed RNA / コピー数: 1 |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 177.720031 KDa |
| 配列 | 文字列: GGGCACUUAC CCUUUAGUGC GAAGGUUUCG ACCUUCGAUC CAUUUGUUCG CAAAUGGGAU GAUCUUCGGA UCAUCCGCGU AGUCUGUUC AGUCGUUUCG ACGACUGGCC CCACUUCGGU GGGGCCACGG UACUUAGAAG UGAACACUAA GUGUCGUGAA C ACCAUUUG ...文字列: GGGCACUUAC CCUUUAGUGC GAAGGUUUCG ACCUUCGAUC CAUUUGUUCG CAAAUGGGAU GAUCUUCGGA UCAUCCGCGU AGUCUGUUC AGUCGUUUCG ACGACUGGCC CCACUUCGGU GGGGCCACGG UACUUAGAAG UGAACACUAA GUGUCGUGAA C ACCAUUUG GUUAACUGCU CAAACUAAAU GGUGAUGAGG GAAGGAAUGA CCCUCAUCGG ACUACGCGAU CCGAGUGAUG GG AAUGGCU GACCCAUCGC UCGGCACUGG AGGGUGAGUG CCCCUCAUUC GCAUAAGGGC CGACCCAGAC AACAGCCAAG UUU GGGUCG GAGAUGCGAA CAUUCCACGC AUCUGAACGG UUGAGAACUU ACAAGGGCAA GAGCAGAGUC CUUGUAAGGG CUUU ACACG UCAAGUUCAC AGACGUGUAA GGCCCGUCGC CCUUCGGGGC GACGUUCACG GCAUUUCGAU GCCGUGCAGC CUGUU CGCA GGCUGCUUGA CCGUUCCCCU GCCCUUUCGA GGGCAGACUA CUCUUCGGAG UAGUCUUAUG UGAAUGAG |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 3.0 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 構成要素:
詳細: Freshly prepared and filtered through 0.22 micron filter prior to use. | ||||||||||||
| グリッド | モデル: UltrAuFoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 45 sec. / 前処理 - 雰囲気: AIR / 詳細: 15mA current on a glocube. | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 99 % / チャンバー内温度: 294 K / 装置: LEICA EM GP 詳細: 3 microlitre droplet, 4 second delay before blotting, 6 second blot, 0 second delay before plunging.. | ||||||||||||
| 詳細 | Sample was purified by size exclusion chromatography and concentrated in an Amicon spin concentrator. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 倍率(公称値): 130000 Bright-field microscopy / Cs: 2.7 mm / 倍率(公称値): 130000 |
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 撮影したグリッド数: 1 / 実像数: 6122 / 平均露光時間: 1.5 sec. / 平均電子線量: 60.0 e/Å2 / 詳細: Images were collected as 56 frame movies. |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT / 当てはまり具合の基準: Correlation Coefficient |
|---|---|
| 得られたモデル |  PDB-7qdu: |
 ムービー
ムービー コントローラー
コントローラー













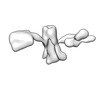
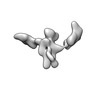
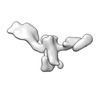

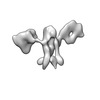
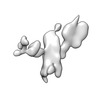
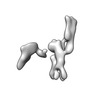






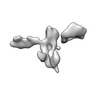
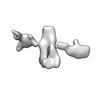




 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)