+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-1254 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
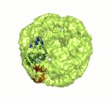
| タイトル | Hexameric ring structure of human MCM10 DNA replication factor. | |||||||||
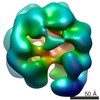
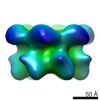
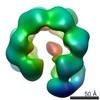
 マップデータ マップデータ | Surface views of human Mcm10 top side bottom stereo | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  ネガティブ染色法 / 解像度: 16.0 Å ネガティブ染色法 / 解像度: 16.0 Å | |||||||||
 データ登録者 データ登録者 | Okorokov AL / Orlova EV | |||||||||
 引用 引用 |  ジャーナル: EMBO Rep / 年: 2007 ジャーナル: EMBO Rep / 年: 2007タイトル: Hexameric ring structure of human MCM10 DNA replication factor. 著者: Andrei L Okorokov / Alastair Waugh / Julie Hodgkinson / Andal Murthy / Hye Kyung Hong / Elisabetta Leo / Michael B Sherman / Kai Stoeber / Elena V Orlova / Gareth H Williams /  要旨: The DNA replication factor minichromosome maintenance 10 (MCM10) is a conserved, abundant nuclear protein crucial for origin firing. During the transition from pre-replicative complexes to pre- ...The DNA replication factor minichromosome maintenance 10 (MCM10) is a conserved, abundant nuclear protein crucial for origin firing. During the transition from pre-replicative complexes to pre-initiation complexes, MCM10 recruitment to replication origins is required to provide a physical link between the MCM2-7 complex DNA helicase and DNA polymerases. Here, we report the molecular structure of human MCM10 as determined by electron microscopy and single-particle analysis. The MCM10 molecule is a ring-shaped hexamer with large central and smaller lateral channels and a system of inner chambers. This structure, together with biochemical data, suggests that this important protein uses its architecture to provide a docking module for assembly of the molecular machinery required for eukaryotic DNA replication. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_1254.map.gz emd_1254.map.gz | 1.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-1254-v30.xml emd-1254-v30.xml emd-1254.xml emd-1254.xml | 10.4 KB 10.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  1254.gif 1254.gif | 64.6 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-1254 http://ftp.pdbj.org/pub/emdb/structures/EMD-1254 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1254 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1254 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_1254.map.gz / 形式: CCP4 / 大きさ: 6.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_1254.map.gz / 形式: CCP4 / 大きさ: 6.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Surface views of human Mcm10 top side bottom stereo | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.59 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : recombinant human Mcm10
| 全体 | 名称: recombinant human Mcm10 |
|---|---|
| 要素 |
|
-超分子 #1000: recombinant human Mcm10
| 超分子 | 名称: recombinant human Mcm10 / タイプ: sample / ID: 1000 / 詳細: monodisperse / 集合状態: homohexamer / Number unique components: 1 |
|---|---|
| 分子量 | 実験値: 650 KDa / 理論値: 590 KDa / 手法: size-exclusion FPLC |
-分子 #1: Mcm10
| 分子 | 名称: Mcm10 / タイプ: protein_or_peptide / ID: 1 / Name.synonym: DNA replication factor / 詳細: UniProtKB/TrEMBL entry Q3MIR3 / コピー数: 6 / 集合状態: hexamer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) / 別称: Human / 細胞中の位置: nuclear Homo sapiens (ヒト) / 別称: Human / 細胞中の位置: nuclear |
| 分子量 | 実験値: 590 KDa / 理論値: 650 KDa |
| 組換発現 | 生物種: Escherichia coli, Rosetta / 組換プラスミド: pProEX-HT-B |
-実験情報
-構造解析
| 手法 |  ネガティブ染色法 ネガティブ染色法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.01 mg/mL |
|---|---|
| 緩衝液 | pH: 6.8 詳細: 25 mM Tris-HCl pH 9.0, 150 mM NaCl, 10 mM MgCl2, 50 mM KCl |
| 染色 | タイプ: NEGATIVE 詳細: Grids were stained with 2% w/v methylamine tungstate, (Nano-W, Nanoprobes Inc.) for 1 min. |
| グリッド | 詳細: 400 mesh, freshly glow-discharged in air |
| 凍結 | 凍結剤: NONE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI 12 |
|---|---|
| 電子線 | 加速電圧: 100 kV / 電子線源: TUNGSTEN HAIRPIN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.2 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.5 µm / 倍率(公称値): 44000 Bright-field microscopy / Cs: 2.2 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.5 µm / 倍率(公称値): 44000 |
| 試料ステージ | 試料ホルダー: eucentric / 試料ホルダーモデル: OTHER |
| 詳細 | images were taken on FEI Technai T10 microscope in low dose mode. |
| 日付 | 2005年1月1日 |
| 撮影 | カテゴリ: FILM / フィルム・検出器のモデル: KODAK SO-163 FILM / デジタル化 - スキャナー: ZEISS SCAI / デジタル化 - サンプリング間隔: 7 µm / 実像数: 15 / 平均電子線量: 15 e/Å2 / カメラ長: 500 / Od range: 1.4 / ビット/ピクセル: 8 |
- 画像解析
画像解析
| CTF補正 | 詳細: each particle |
|---|---|
| 最終 2次元分類 | クラス数: 197 |
| 最終 再構成 | 想定した対称性 - 点群: C6 (6回回転対称 ) / アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 16.0 Å / 解像度の算出法: FSC 0.5 CUT-OFF / ソフトウェア - 名称: Imagic / 詳細: Final map was calculated from 197 best classes / 使用した粒子像数: 5000 ) / アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 16.0 Å / 解像度の算出法: FSC 0.5 CUT-OFF / ソフトウェア - 名称: Imagic / 詳細: Final map was calculated from 197 best classes / 使用した粒子像数: 5000 |
| 詳細 | The particles were selected interactively at the computer terminal. close |
 ムービー
ムービー コントローラー
コントローラー