+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-1082 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
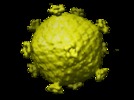
| タイトル | The PM2 virion has a novel organization with an internal membrane and pentameric receptor binding spikes. | |||||||||
 マップデータ マップデータ | This is the PM2 virion reconstruction | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |   Pseudoalteromonas phage PM2 (ファージ) Pseudoalteromonas phage PM2 (ファージ) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 8.4 Å クライオ電子顕微鏡法 / 解像度: 8.4 Å | |||||||||
 データ登録者 データ登録者 | Huiskonen JT / Kivela HM / Bamford DH / Butcher SJ | |||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2004 ジャーナル: Nat Struct Mol Biol / 年: 2004タイトル: The PM2 virion has a novel organization with an internal membrane and pentameric receptor binding spikes. 著者: Juha T Huiskonen / Hanna M Kivelä / Dennis H Bamford / Sarah J Butcher /  要旨: Biological membranes are notoriously resistant to structural analysis. Excellent candidates to tackle this problem in situ are membrane-containing viruses where the membrane is constrained by an ...Biological membranes are notoriously resistant to structural analysis. Excellent candidates to tackle this problem in situ are membrane-containing viruses where the membrane is constrained by an icosahedral capsid. Cryo-EM and image reconstruction of bacteriophage PM2 revealed a membrane bilayer following the internal surface of the capsid. The viral genome closely interacts with the inner leaflet. The capsid, at a resolution of 8.4 A, reveals 200 trimeric capsomers with a pseudo T = 21 dextro organization. Pentameric receptor-binding spikes protrude from the surface. It is evident from the structure that the PM2 membrane has at least two important roles in the life cycle. First, it acts as a scaffold to nucleate capsid assembly. Second, after host recognition, it fuses with the host outer membrane to promote genome entry. The structure also sheds light on how the viral supercoiled circular double-stranded DNA genome might be packaged and released. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_1082.map.gz emd_1082.map.gz | 169.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-1082-v30.xml emd-1082-v30.xml emd-1082.xml emd-1082.xml | 10.7 KB 10.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  1082.gif 1082.gif | 20.2 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-1082 http://ftp.pdbj.org/pub/emdb/structures/EMD-1082 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1082 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1082 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_1082.map.gz / 形式: CCP4 / 大きさ: 392.4 MB / タイプ: IMAGE STORED AS SIGNED INTEGER (2 BYTES) ダウンロード / ファイル: emd_1082.map.gz / 形式: CCP4 / 大きさ: 392.4 MB / タイプ: IMAGE STORED AS SIGNED INTEGER (2 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | This is the PM2 virion reconstruction | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.4 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : PM2 virion
| 全体 | 名称: PM2 virion |
|---|---|
| 要素 |
|
-超分子 #1000: PM2 virion
| 超分子 | 名称: PM2 virion / タイプ: sample / ID: 1000 詳細: Purified virion (1 mg/ml) was purified, pelleted and resuspended just prior to application to the grid. Number unique components: 1 |
|---|---|
| 分子量 | 理論値: 33.8 MDa 手法: Reported mass of the whole virion in the literature is 47 MDa, 72 percent of which is protein. |
-超分子 #1: Pseudoalteromonas phage PM2
| 超分子 | 名称: Pseudoalteromonas phage PM2 / タイプ: virus / ID: 1 / Name.synonym: PM2 / 詳細: includes mass of DNA, lipid and protein / NCBI-ID: 10661 / 生物種: Pseudoalteromonas phage PM2 / ウイルスタイプ: VIRION / ウイルス・単離状態: STRAIN / ウイルス・エンベロープ: Yes / ウイルス・中空状態: No / Syn species name: PM2 |
|---|---|
| 宿主 | 生物種:  Pseudoalteromonas sp. ER72M2 (バクテリア) / 別称: BACTERIA(EUBACTERIA) Pseudoalteromonas sp. ER72M2 (バクテリア) / 別称: BACTERIA(EUBACTERIA) |
| 分子量 | 実験値: 47 MDa |
| ウイルス殻 | Shell ID: 1 / 名称: P2 / 直径: 597 Å / T番号(三角分割数): 21 |
| ウイルス殻 | Shell ID: 2 / 名称: virion (spike-spike distance) / 直径: 765 Å / T番号(三角分割数): 21 |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 4 mg/mL |
|---|---|
| 緩衝液 | pH: 7.2 / 詳細: CaCl2, 5mM Tris-HCl, 10-20 mM NaCl, 25-125 mM |
| グリッド | 詳細: 400 mesh copper grid, R2/2 quantifoil holey |
| 凍結 | 凍結剤: ETHANE / チャンバー内温度: 90 K / 装置: HOMEMADE PLUNGER / 詳細: Vitrification instrument: EMBL design 手法: A small vial of ethane is placed inside a larger liquid nitrogen reservoir. The grid holding 3 microliters of the sample is held in place at the bottom of a plunger by the means of fine ...手法: A small vial of ethane is placed inside a larger liquid nitrogen reservoir. The grid holding 3 microliters of the sample is held in place at the bottom of a plunger by the means of fine tweezers. When the liquid ethane is ready, a piece of filter paper is then pressed against the sample to blot off excess buffer, sufficient to leave a thin layer on the grid. The filter paper is removed, and the plunger is allowed to drop into the liquid ethane. Once the grid enters the liquid ethane, the sample is rapidly frozen, and the grid is transferred under liquid nitrogen to a storage box immersed in liquid nitrogen for later use in the microscope. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI F20 |
|---|---|
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2 mm / 最大 デフォーカス(公称値): 2.9 µm / 最小 デフォーカス(公称値): 0.6 µm / 倍率(公称値): 50000 Bright-field microscopy / Cs: 2 mm / 最大 デフォーカス(公称値): 2.9 µm / 最小 デフォーカス(公称値): 0.6 µm / 倍率(公称値): 50000 |
| 試料ステージ | 試料ホルダー: Side entry liquid nitrogen-cooled cryo specimen holder 試料ホルダーモデル: GATAN LIQUID NITROGEN |
| 温度 | 最低: 90 K / 最高: 94 K / 平均: 93 K |
| アライメント法 | Legacy - 非点収差: objective lens astigmatism was corrected at |
| 詳細 | A box anticontaminator was fitted with a minimum achievable temperature of 88 K |
| 撮影 | カテゴリ: FILM / フィルム・検出器のモデル: KODAK SO-163 FILM / デジタル化 - スキャナー: ZEISS SCAI / デジタル化 - サンプリング間隔: 7 µm / 実像数: 111 / 平均電子線量: 10 e/Å2 / ビット/ピクセル: 12 |
| 実験機器 |  モデル: Tecnai F20 / 画像提供: FEI Company |
- 画像解析
画像解析
| CTF補正 | 詳細: each particle, wiener factor 0.1 |
|---|---|
| 最終 再構成 | 想定した対称性 - 点群: I (正20面体型対称 ) / アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 8.4 Å / 解像度の算出法: FSC 0.5 CUT-OFF / ソフトウェア - 名称: EMBL, P3DR / 使用した粒子像数: 13887 ) / アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 8.4 Å / 解像度の算出法: FSC 0.5 CUT-OFF / ソフトウェア - 名称: EMBL, P3DR / 使用した粒子像数: 13887 |
| 詳細 | The particles were first automatically selected using ETHAN, and then boxed out manually in EMAN. |
 ムービー
ムービー コントローラー
コントローラー