+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-10814 | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | In-situ structure of HEF from filamentous influenza C viral particles by cryo-ET | ||||||||||||
 マップデータ マップデータ | |||||||||||||
 試料 試料 |
| ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報sialate 9-O-acetylesterase activity / sialate 4-O-acetylesterase activity /  シアル酸-O-アセチルエステラーゼ / シアル酸-O-アセチルエステラーゼ /  viral budding from plasma membrane / endocytosis involved in viral entry into host cell / host cell surface receptor binding / fusion of virus membrane with host plasma membrane / fusion of virus membrane with host endosome membrane / viral budding from plasma membrane / endocytosis involved in viral entry into host cell / host cell surface receptor binding / fusion of virus membrane with host plasma membrane / fusion of virus membrane with host endosome membrane /  エンベロープ (ウイルス) / virion attachment to host cell ...sialate 9-O-acetylesterase activity / sialate 4-O-acetylesterase activity / エンベロープ (ウイルス) / virion attachment to host cell ...sialate 9-O-acetylesterase activity / sialate 4-O-acetylesterase activity /  シアル酸-O-アセチルエステラーゼ / シアル酸-O-アセチルエステラーゼ /  viral budding from plasma membrane / endocytosis involved in viral entry into host cell / host cell surface receptor binding / fusion of virus membrane with host plasma membrane / fusion of virus membrane with host endosome membrane / viral budding from plasma membrane / endocytosis involved in viral entry into host cell / host cell surface receptor binding / fusion of virus membrane with host plasma membrane / fusion of virus membrane with host endosome membrane /  エンベロープ (ウイルス) / virion attachment to host cell / host cell plasma membrane / virion membrane / エンベロープ (ウイルス) / virion attachment to host cell / host cell plasma membrane / virion membrane /  生体膜 生体膜類似検索 - 分子機能 | ||||||||||||
| 生物種 |   Canis lupus (オオカミ) / Canis lupus (オオカミ) /   Influenza C virus (インフルエンザウイルス) Influenza C virus (インフルエンザウイルス) | ||||||||||||
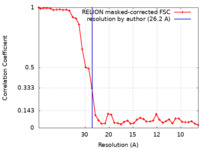
| 手法 | サブトモグラム平均法 /  クライオ電子顕微鏡法 / 解像度: 26.2 Å クライオ電子顕微鏡法 / 解像度: 26.2 Å | ||||||||||||
 データ登録者 データ登録者 | Halldorsson S / Rosenthal PB | ||||||||||||
| 資金援助 |  英国, 3件 英国, 3件
| ||||||||||||
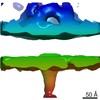
 引用 引用 |  ジャーナル: Nat Commun / 年: 2021 ジャーナル: Nat Commun / 年: 2021タイトル: In situ structure and organization of the influenza C virus surface glycoprotein. 著者: Steinar Halldorsson / Kasim Sader / Jack Turner / Lesley J Calder / Peter B Rosenthal /   要旨: The lipid-enveloped influenza C virus contains a single surface glycoprotein, the haemagglutinin-esterase-fusion (HEF) protein, that mediates receptor binding, receptor destruction, and membrane ...The lipid-enveloped influenza C virus contains a single surface glycoprotein, the haemagglutinin-esterase-fusion (HEF) protein, that mediates receptor binding, receptor destruction, and membrane fusion at the low pH of the endosome. Here we apply electron cryotomography and subtomogram averaging to describe the structural basis for hexagonal lattice formation by HEF on the viral surface. The conformation of the glycoprotein in situ is distinct from the structure of the isolated trimeric ectodomain, showing that a splaying of the membrane distal domains is required to mediate contacts that form the lattice. The splaying of these domains is also coupled to changes in the structure of the stem region which is involved in membrane fusion, thereby linking HEF's membrane fusion conformation with its assembly on the virus surface. The glycoprotein lattice can form independent of other virion components but we show a major role for the matrix layer in particle formation. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_10814.map.gz emd_10814.map.gz | 3.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-10814-v30.xml emd-10814-v30.xml emd-10814.xml emd-10814.xml | 20.4 KB 20.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_10814_fsc.xml emd_10814_fsc.xml | 3.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_10814.png emd_10814.png | 105.9 KB | ||
| マスクデータ |  emd_10814_msk_1.map emd_10814_msk_1.map | 3.8 MB |  マスクマップ マスクマップ | |
| その他 |  emd_10814_additional_1.map.gz emd_10814_additional_1.map.gz emd_10814_half_map_1.map.gz emd_10814_half_map_1.map.gz emd_10814_half_map_2.map.gz emd_10814_half_map_2.map.gz | 2.4 MB 2.4 MB 2.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-10814 http://ftp.pdbj.org/pub/emdb/structures/EMD-10814 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10814 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10814 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_10814.map.gz / 形式: CCP4 / 大きさ: 3.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_10814.map.gz / 形式: CCP4 / 大きさ: 3.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 4.46 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_10814_msk_1.map emd_10814_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Unfiltered map
| ファイル | emd_10814_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unfiltered map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map 1
| ファイル | emd_10814_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map 2
| ファイル | emd_10814_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Influenza C virus
| 全体 | 名称:   Influenza C virus (インフルエンザウイルス) Influenza C virus (インフルエンザウイルス) |
|---|---|
| 要素 |
|
-超分子 #1: Influenza C virus
| 超分子 | 名称: Influenza C virus / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: all / 詳細: Purified from infected cells. / NCBI-ID: 11552 / 生物種: Influenza C virus / Sci species strain: C/Johannesburg/1/1966 / ウイルスタイプ: VIRION / ウイルス・単離状態: STRAIN / ウイルス・エンベロープ: Yes / ウイルス・中空状態: No |
|---|---|
| 宿主 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Hemagglutinin esterase fusion
| 分子 | 名称: Hemagglutinin esterase fusion / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO / EC番号:  シアル酸-O-アセチルエステラーゼ シアル酸-O-アセチルエステラーゼ |
|---|---|
| 由来(天然) | 生物種:   Canis lupus (オオカミ) Canis lupus (オオカミ) |
| 配列 | 文字列: MFFSLLLVLG LTEAEKIKIC LQKQVNSSFS LHNGFGGNLY ATEEKRMFEL VKPKAGASVL NQSTWIGFG DSRTDKSNSA FPRSADVSAK TADKFRFLSG GSLMLSMFGP PGKVDYLYQG C GKHKVFYE GVNWSPHAAI NCYRKNWTDI KLNFQKNIYE LASQSHCMSL ...文字列: MFFSLLLVLG LTEAEKIKIC LQKQVNSSFS LHNGFGGNLY ATEEKRMFEL VKPKAGASVL NQSTWIGFG DSRTDKSNSA FPRSADVSAK TADKFRFLSG GSLMLSMFGP PGKVDYLYQG C GKHKVFYE GVNWSPHAAI NCYRKNWTDI KLNFQKNIYE LASQSHCMSL VNALDKTIPL QV TAGTAGN CNNSFLKNPA LYTQEVKPSE NKCGKENLAF FTLPTQFGTY ECKLHLVASC YFI YDSKEV YNKRGCDNYF QVIYDSFGKV VGGLDNRVSP YTGNSGDTPT MQCDMLQLKP GRYS VRSSP RFLLMPERSY CFDMKEKGPV TAVQSIWGKG RESDYAVDQA CLSTPGCMLI QKQKP YIGE ADDHHGDQEM RELLSGLDYE ARCISQSGWV NETSPFTEKY LLPPKFGRCP LAAKEE SIP KIPDGLLIPT SGTDTTVTKP KSRIFGIDDL IIGVLFVAIV ETGIGGYLLG SRKESGG GV TKESAEKGFE KIGNDIQILK SSINIAIEKL NDRISHDEQA IRDLTLEIEN ARSEALLG E LGIIRALLVG NISIGLQESL WELASEITNR AGDLAVEVSP GCWIIDNNIC DQSCQNFIF KFNETAPVPT IPPLDTKIDL QSDPFYWGSS LGLAITATIS LAALVISGIA ICRTK |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | サブトモグラム平均法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.4 構成要素:
| ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK III | ||||||||||||
| 詳細 | Heterogeneous mix of viral particles, viral like particles and cellular vesicles |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm Bright-field microscopy / Cs: 2.7 mm最大 デフォーカス(公称値): 0.0045000000000000005 µm 最小 デフォーカス(公称値): 0.0025 µm / 倍率(公称値): 37000 |
| 特殊光学系 | エネルギーフィルター - スリット幅: 20 eV |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: COUNTING / デジタル化 - サイズ - 横: 4096 pixel / デジタル化 - サイズ - 縦: 4096 pixel / 撮影したグリッド数: 1 / 平均電子線量: 1.36 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | PDB ID: |
|---|---|
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT |
 ムービー
ムービー コントローラー
コントローラー




















 Z
Z Y
Y X
X