+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 8of4 | ||||||
|---|---|---|---|---|---|---|---|
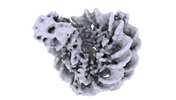
| タイトル | Nucleosome Bound human SIRT6 (Composite) | ||||||
 要素 要素 |
| ||||||
 キーワード キーワード |  GENE REGULATION (遺伝子発現の調節) / GENE REGULATION (遺伝子発現の調節) /  Transferase (転移酵素) / Transferase (転移酵素) /  Deacetylase / Histone H3 deacetylation Deacetylase / Histone H3 deacetylation | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報NAD-dependent histone H3K56 deacetylase activity / NAD-dependent histone H3K18 deacetylase activity / ketone biosynthetic process / NAD-dependent histone H3K9 deacetylase activity / protein delipidation / regulation of lipid catabolic process / NAD+- protein-lysine ADP-ribosyltransferase activity /  chromosome, subtelomeric region / NAD-dependent protein demyristoylase activity / NAD-dependent protein depalmitoylase activity ...NAD-dependent histone H3K56 deacetylase activity / NAD-dependent histone H3K18 deacetylase activity / ketone biosynthetic process / NAD-dependent histone H3K9 deacetylase activity / protein delipidation / regulation of lipid catabolic process / NAD+- protein-lysine ADP-ribosyltransferase activity / chromosome, subtelomeric region / NAD-dependent protein demyristoylase activity / NAD-dependent protein depalmitoylase activity ...NAD-dependent histone H3K56 deacetylase activity / NAD-dependent histone H3K18 deacetylase activity / ketone biosynthetic process / NAD-dependent histone H3K9 deacetylase activity / protein delipidation / regulation of lipid catabolic process / NAD+- protein-lysine ADP-ribosyltransferase activity /  chromosome, subtelomeric region / NAD-dependent protein demyristoylase activity / NAD-dependent protein depalmitoylase activity / NAD+-protein-arginine ADP-ribosyltransferase activity / positive regulation of protein localization to chromatin / DNA damage sensor activity / positive regulation of stem cell differentiation / positive regulation of blood vessel branching / NAD-dependent protein lysine deacetylase activity / retrotransposon silencing / protein localization to site of double-strand break / protein acetyllysine N-acetyltransferase / cardiac muscle cell differentiation / positive regulation of chondrocyte proliferation / pericentric heterochromatin formation / positive regulation of telomere maintenance / NAD-dependent histone deacetylase activity / protein deacetylation / negative regulation of glucose import / TORC2 complex binding / chromosome, subtelomeric region / NAD-dependent protein demyristoylase activity / NAD-dependent protein depalmitoylase activity / NAD+-protein-arginine ADP-ribosyltransferase activity / positive regulation of protein localization to chromatin / DNA damage sensor activity / positive regulation of stem cell differentiation / positive regulation of blood vessel branching / NAD-dependent protein lysine deacetylase activity / retrotransposon silencing / protein localization to site of double-strand break / protein acetyllysine N-acetyltransferase / cardiac muscle cell differentiation / positive regulation of chondrocyte proliferation / pericentric heterochromatin formation / positive regulation of telomere maintenance / NAD-dependent histone deacetylase activity / protein deacetylation / negative regulation of glucose import / TORC2 complex binding /  lncRNA binding / negative regulation of glycolytic process / negative regulation of protein localization to chromatin / positive regulation of double-strand break repair / DNA repair-dependent chromatin remodeling / positive regulation of vascular endothelial cell proliferation / regulation of double-strand break repair via homologous recombination / negative regulation of protein import into nucleus / negative regulation of gene expression, epigenetic / positive regulation of stem cell population maintenance / positive regulation of stem cell proliferation / lncRNA binding / negative regulation of glycolytic process / negative regulation of protein localization to chromatin / positive regulation of double-strand break repair / DNA repair-dependent chromatin remodeling / positive regulation of vascular endothelial cell proliferation / regulation of double-strand break repair via homologous recombination / negative regulation of protein import into nucleus / negative regulation of gene expression, epigenetic / positive regulation of stem cell population maintenance / positive regulation of stem cell proliferation /  regulation of protein secretion / negative regulation of transcription elongation by RNA polymerase II / site of DNA damage / NAD+-protein ADP-ribosyltransferase activity / negative regulation of cellular senescence / regulation of lipid metabolic process / regulation of protein secretion / negative regulation of transcription elongation by RNA polymerase II / site of DNA damage / NAD+-protein ADP-ribosyltransferase activity / negative regulation of cellular senescence / regulation of lipid metabolic process /  転移酵素; グリコシル基を移すもの; 五炭糖残基を移すもの / 転移酵素; グリコシル基を移すもの; 五炭糖残基を移すもの /  NAD+ ADP-ribosyltransferase activity / NAD+ ADP-ribosyltransferase activity /  NAD+ binding / subtelomeric heterochromatin formation / positive regulation of fat cell differentiation / regulation of protein localization to plasma membrane / pericentric heterochromatin / NAD+ binding / subtelomeric heterochromatin formation / positive regulation of fat cell differentiation / regulation of protein localization to plasma membrane / pericentric heterochromatin /  nucleosome binding / negative regulation of gluconeogenesis / response to UV / nucleosome binding / negative regulation of gluconeogenesis / response to UV /  転移酵素; アシル基を移すもの; アミノアシル基以外のアシル基を移すもの / 転移酵素; アシル基を移すもの; アミノアシル基以外のアシル基を移すもの /  nucleotidyltransferase activity / positive regulation of protein export from nucleus / determination of adult lifespan / circadian regulation of gene expression / protein destabilization / nucleotidyltransferase activity / positive regulation of protein export from nucleus / determination of adult lifespan / circadian regulation of gene expression / protein destabilization /  塩基除去修復 / 塩基除去修復 /  regulation of circadian rhythm / positive regulation of insulin secretion / chromatin DNA binding / Pre-NOTCH Transcription and Translation / structural constituent of chromatin / transcription corepressor activity / double-strand break repair / positive regulation of fibroblast proliferation / regulation of circadian rhythm / positive regulation of insulin secretion / chromatin DNA binding / Pre-NOTCH Transcription and Translation / structural constituent of chromatin / transcription corepressor activity / double-strand break repair / positive regulation of fibroblast proliferation /  ヌクレオソーム / ヌクレオソーム /  nucleosome assembly / positive regulation of proteasomal ubiquitin-dependent protein catabolic process / nucleosome assembly / positive regulation of proteasomal ubiquitin-dependent protein catabolic process /  glucose homeostasis / site of double-strand break / positive regulation of cold-induced thermogenesis / Processing of DNA double-strand break ends / damaged DNA binding / glucose homeostasis / site of double-strand break / positive regulation of cold-induced thermogenesis / Processing of DNA double-strand break ends / damaged DNA binding /  クロマチンリモデリング / protein heterodimerization activity / negative regulation of cell population proliferation / intracellular membrane-bounded organelle / クロマチンリモデリング / protein heterodimerization activity / negative regulation of cell population proliferation / intracellular membrane-bounded organelle /  chromatin binding / chromatin binding /  クロマチン / negative regulation of transcription by RNA polymerase II / クロマチン / negative regulation of transcription by RNA polymerase II /  小胞体 / protein homodimerization activity / 小胞体 / protein homodimerization activity /  DNA binding / zinc ion binding / DNA binding / zinc ion binding /  核質 / 核質 /  細胞核 細胞核類似検索 - 分子機能 | ||||||
| 生物種 |  Xenopus laevis (アフリカツメガエル) Xenopus laevis (アフリカツメガエル)  Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||
| 手法 |  電子顕微鏡法 / 電子顕微鏡法 /  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 2.94 Å クライオ電子顕微鏡法 / 解像度: 2.94 Å | ||||||
 データ登録者 データ登録者 | Smirnova, E. / Bignon, E. / Schultz, P. / Papai, G. / Ben-Shem, A. | ||||||
| 資金援助 |  フランス, 1件 フランス, 1件
| ||||||
 引用 引用 |  ジャーナル: Elife / 年: 2024 ジャーナル: Elife / 年: 2024タイトル: Binding to nucleosome poises human SIRT6 for histone H3 deacetylation. 著者: Ekaterina Smirnova / Emmanuelle Bignon / Patrick Schultz / Gabor Papai / Adam Ben Shem /  要旨: Sirtuin 6 (SIRT6) is an NAD-dependent histone H3 deacetylase that is prominently found associated with chromatin, attenuates transcriptionally active promoters and regulates DNA repair, metabolic ...Sirtuin 6 (SIRT6) is an NAD-dependent histone H3 deacetylase that is prominently found associated with chromatin, attenuates transcriptionally active promoters and regulates DNA repair, metabolic homeostasis and lifespan. Unlike other sirtuins, it has low affinity to free histone tails but demonstrates strong binding to nucleosomes. It is poorly understood how SIRT6 docking on nucleosomes stimulates its histone deacetylation activity. Here, we present the structure of human SIRT6 bound to a nucleosome determined by cryogenic electron microscopy. The zinc finger domain of SIRT6 associates tightly with the acidic patch of the nucleosome through multiple arginine anchors. The Rossmann fold domain binds to the terminus of the looser DNA half of the nucleosome, detaching two turns of the DNA from the histone octamer and placing the NAD binding pocket close to the DNA exit site. This domain shows flexibility with respect to the fixed zinc finger and moves with, but also relative to, the unwrapped DNA terminus. We apply molecular dynamics simulations of the histone tails in the nucleosome to show that in this mode of interaction, the active site of SIRT6 is perfectly poised to catalyze deacetylation of the H3 histone tail and that the partial unwrapping of the DNA allows even lysines close to the H3 core to reach the enzyme. #1:  ジャーナル: Elife / 年: 2023 ジャーナル: Elife / 年: 2023タイトル: Binding to nucleosome poises SIRT6 for histone H3 de-acetylation 著者: Smirnova, E. / Bignon, E. / Schultz, P. / Papai, G. / Ben-Shem, A. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  8of4.cif.gz 8of4.cif.gz | 358 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb8of4.ent.gz pdb8of4.ent.gz | 265.8 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  8of4.json.gz 8of4.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/of/8of4 https://data.pdbj.org/pub/pdb/validation_reports/of/8of4 ftp://data.pdbj.org/pub/pdb/validation_reports/of/8of4 ftp://data.pdbj.org/pub/pdb/validation_reports/of/8of4 | HTTPS FTP |
|---|
-関連構造データ
| 関連構造データ |  16845MC C: 同じ文献を引用 ( M: このデータのモデリングに利用したマップデータ |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
-タンパク質 , 5種, 9分子 AEBFCGDHL
| #1: タンパク質 | 分子量: 15421.101 Da / 分子数: 2 / 由来タイプ: 組換発現 由来: (組換発現)  Xenopus laevis (アフリカツメガエル) Xenopus laevis (アフリカツメガエル)発現宿主:   Escherichia coli (大腸菌) / 参照: UniProt: P84233 Escherichia coli (大腸菌) / 参照: UniProt: P84233#2: タンパク質 |  ヒストンH4 ヒストンH4分子量: 11394.426 Da / 分子数: 2 / 由来タイプ: 組換発現 由来: (組換発現)  Xenopus laevis (アフリカツメガエル) Xenopus laevis (アフリカツメガエル)発現宿主:   Escherichia coli (大腸菌) / 参照: UniProt: P62799 Escherichia coli (大腸菌) / 参照: UniProt: P62799#3: タンパク質 | 分子量: 14093.436 Da / 分子数: 2 / 由来タイプ: 組換発現 由来: (組換発現)  Xenopus laevis (アフリカツメガエル) Xenopus laevis (アフリカツメガエル)発現宿主:   Escherichia coli (大腸菌) / 参照: UniProt: P06897 Escherichia coli (大腸菌) / 参照: UniProt: P06897#4: タンパク質 |  ヒストンH2B ヒストンH2B分子量: 13979.291 Da / 分子数: 2 / 由来タイプ: 組換発現 由来: (組換発現)  Xenopus laevis (アフリカツメガエル) Xenopus laevis (アフリカツメガエル)遺伝子: LOC108704303 / 発現宿主:   Escherichia coli (大腸菌) / 参照: UniProt: A0A8J0U496 Escherichia coli (大腸菌) / 参照: UniProt: A0A8J0U496#7: タンパク質 | | 分子量: 39180.914 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)   Homo sapiens (ヒト) / 遺伝子: SIRT6, SIR2L6 / 発現宿主: Homo sapiens (ヒト) / 遺伝子: SIRT6, SIR2L6 / 発現宿主:   Escherichia coli (大腸菌) Escherichia coli (大腸菌)参照: UniProt: Q8N6T7, 転移酵素; アシル基を移すもの; アミノアシル基以外のアシル基を移すもの, protein acetyllysine N-acetyltransferase, 転移酵素; ...参照: UniProt: Q8N6T7,  転移酵素; アシル基を移すもの; アミノアシル基以外のアシル基を移すもの, protein acetyllysine N-acetyltransferase, 転移酵素; アシル基を移すもの; アミノアシル基以外のアシル基を移すもの, protein acetyllysine N-acetyltransferase,  転移酵素; グリコシル基を移すもの; 五炭糖残基を移すもの 転移酵素; グリコシル基を移すもの; 五炭糖残基を移すもの |
|---|
-DNA鎖 , 2種, 2分子 IJ
| #5: DNA鎖 | 分子量: 44520.383 Da / 分子数: 1 / 由来タイプ: 組換発現 由来: (組換発現)  Xenopus laevis (アフリカツメガエル) Xenopus laevis (アフリカツメガエル)発現宿主:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
|---|---|
| #6: DNA鎖 | 分子量: 44991.660 Da / 分子数: 1 / 由来タイプ: 組換発現 由来: (組換発現)  Xenopus laevis (アフリカツメガエル) Xenopus laevis (アフリカツメガエル)発現宿主:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
-非ポリマー , 1種, 1分子 
| #8: 化合物 | ChemComp-ZN / |
|---|
-詳細
| 研究の焦点であるリガンドがあるか | Y |
|---|
-実験情報
-実験
| 実験 | 手法:  電子顕微鏡法 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法:  単粒子再構成法 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 |
| ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 分子量 |
| ||||||||||||||||||
| 由来(天然) |
| ||||||||||||||||||
| 由来(組換発現) |
| ||||||||||||||||||
| 緩衝液 | pH: 7.5 | ||||||||||||||||||
| 試料 | 包埋: NO / シャドウイング: NO / 染色 : NO / 凍結 : NO / 凍結 : YES : YES | ||||||||||||||||||
| 試料支持 | グリッドの材料: COPPER/RHODIUM / グリッドのサイズ: 300 divisions/in. / グリッドのタイプ: Quantifoil R3.5/1 | ||||||||||||||||||
急速凍結 | 凍結剤: ETHANE |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| EM imaging | 加速電圧: 300 kV / C2レンズ絞り径: 50 µm / 凍結剤: NITROGEN / 電子線源
| ||||||||||||
| 撮影 |
| ||||||||||||
| 電子光学装置 |
|
- 解析
解析
| EMソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
CTF補正 | タイプ: PHASE FLIPPING AND AMPLITUDE CORRECTION | ||||||||||||||||||||||||||||||||||||||||||||||||
| 粒子像の選択 | 選択した粒子像数: 2033169 | ||||||||||||||||||||||||||||||||||||||||||||||||
3次元再構成 | 解像度: 2.94 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 439796 / アルゴリズム: BACK PROJECTION / 対称性のタイプ: POINT | ||||||||||||||||||||||||||||||||||||||||||||||||
| 原子モデル構築 |
|
 ムービー
ムービー コントローラー
コントローラー






 PDBj
PDBj











































