+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 8dh6 | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of Saccharomyces cerevisiae cytochrome c oxidase (Complex IV) extracted in lipid nanodiscs | ||||||
 要素 要素 | (Cytochrome c oxidase subunit ... シトクロムcオキシダーゼ) x 9 シトクロムcオキシダーゼ) x 9 | ||||||
 キーワード キーワード |  MEMBRANE PROTEIN (膜タンパク質) / MEMBRANE PROTEIN (膜タンパク質) /  cytochrome c oxidase (シトクロムcオキシダーゼ) / cytochrome c oxidase (シトクロムcオキシダーゼ) /  Complex IV (シトクロムcオキシダーゼ) Complex IV (シトクロムcオキシダーゼ) | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報mitochondrial cytochrome c oxidase assembly / mitochondrial respiratory chain complex IV /  シトクロムcオキシダーゼ / シトクロムcオキシダーゼ /  細胞呼吸 / mitochondrial electron transport, cytochrome c to oxygen / 細胞呼吸 / mitochondrial electron transport, cytochrome c to oxygen /  cytochrome-c oxidase activity / electron transport coupled proton transport / ATP synthesis coupled electron transport / cytochrome-c oxidase activity / electron transport coupled proton transport / ATP synthesis coupled electron transport /  細胞呼吸 / proton transmembrane transport ...mitochondrial cytochrome c oxidase assembly / mitochondrial respiratory chain complex IV / 細胞呼吸 / proton transmembrane transport ...mitochondrial cytochrome c oxidase assembly / mitochondrial respiratory chain complex IV /  シトクロムcオキシダーゼ / シトクロムcオキシダーゼ /  細胞呼吸 / mitochondrial electron transport, cytochrome c to oxygen / 細胞呼吸 / mitochondrial electron transport, cytochrome c to oxygen /  cytochrome-c oxidase activity / electron transport coupled proton transport / ATP synthesis coupled electron transport / cytochrome-c oxidase activity / electron transport coupled proton transport / ATP synthesis coupled electron transport /  細胞呼吸 / proton transmembrane transport / 細胞呼吸 / proton transmembrane transport /  ミトコンドリア / ミトコンドリア /  ミトコンドリア / ミトコンドリア /  ミトコンドリア内膜 / ミトコンドリア内膜 /  oxidoreductase activity / copper ion binding / oxidoreductase activity / copper ion binding /  heme binding / heme binding /  ミトコンドリア / zinc ion binding / ミトコンドリア / zinc ion binding /  metal ion binding metal ion binding類似検索 - 分子機能 | ||||||
| 生物種 |   Saccharomyces cerevisiae (パン酵母) Saccharomyces cerevisiae (パン酵母) | ||||||
| 手法 |  電子顕微鏡法 / 電子顕微鏡法 /  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 2.94 Å クライオ電子顕微鏡法 / 解像度: 2.94 Å | ||||||
 データ登録者 データ登録者 | Godoy, A.S. / Song, Y. / Cheruvara, H. / Quigley, A. / Oliva, G. | ||||||
| 資金援助 |  ブラジル, 1件 ブラジル, 1件
| ||||||
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Publishedタイトル: Cryo-EM structure of Saccharomyces cerevisiae cytochrome c oxidase (Complex IV) extracted in lipid nanodiscs 著者: Godoy, A.S. / Song, Y. / Cheruvara, H. / Quigley, A. / Oliva, G. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  8dh6.cif.gz 8dh6.cif.gz | 296 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb8dh6.ent.gz pdb8dh6.ent.gz | 236.4 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  8dh6.json.gz 8dh6.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/dh/8dh6 https://data.pdbj.org/pub/pdb/validation_reports/dh/8dh6 ftp://data.pdbj.org/pub/pdb/validation_reports/dh/8dh6 ftp://data.pdbj.org/pub/pdb/validation_reports/dh/8dh6 | HTTPS FTP |
|---|
-関連構造データ
| 関連構造データ | 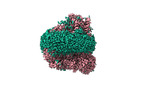 27430MC  8dh7C M: このデータのモデリングに利用したマップデータ C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
-Cytochrome c oxidase subunit ... , 9種, 9分子 abcdefghi
| #1: タンパク質 |  シトクロムcオキシダーゼ / Cytochrome c oxidase polypeptide I シトクロムcオキシダーゼ / Cytochrome c oxidase polypeptide I分子量: 58832.586 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)   Saccharomyces cerevisiae (パン酵母) / 参照: UniProt: P00401, Saccharomyces cerevisiae (パン酵母) / 参照: UniProt: P00401,  シトクロムcオキシダーゼ シトクロムcオキシダーゼ |
|---|---|
| #2: タンパク質 |  / Cytochrome c oxidase polypeptide II / Cytochrome c oxidase polypeptide II分子量: 26779.816 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)   Saccharomyces cerevisiae (パン酵母) / 参照: UniProt: P00410, Saccharomyces cerevisiae (パン酵母) / 参照: UniProt: P00410,  シトクロムcオキシダーゼ シトクロムcオキシダーゼ |
| #3: タンパク質 |  シトクロムcオキシダーゼ / Cytochrome c oxidase polypeptide III シトクロムcオキシダーゼ / Cytochrome c oxidase polypeptide III分子量: 30383.582 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)   Saccharomyces cerevisiae (パン酵母) / 参照: UniProt: P00420, Saccharomyces cerevisiae (パン酵母) / 参照: UniProt: P00420,  シトクロムcオキシダーゼ シトクロムcオキシダーゼ |
| #4: タンパク質 |  シトクロムcオキシダーゼ / Cytochrome c oxidase polypeptide IV シトクロムcオキシダーゼ / Cytochrome c oxidase polypeptide IV分子量: 14188.949 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)   Saccharomyces cerevisiae (パン酵母) / 参照: UniProt: P04037 Saccharomyces cerevisiae (パン酵母) / 参照: UniProt: P04037 |
| #5: タンパク質 |  シトクロムcオキシダーゼ / Cytochrome c oxidase polypeptide Va シトクロムcオキシダーゼ / Cytochrome c oxidase polypeptide Va分子量: 14891.784 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)   Saccharomyces cerevisiae (パン酵母) / 参照: UniProt: P00424 Saccharomyces cerevisiae (パン酵母) / 参照: UniProt: P00424 |
| #6: タンパク質 |  シトクロムcオキシダーゼ / Cytochrome c oxidase polypeptide VI シトクロムcオキシダーゼ / Cytochrome c oxidase polypeptide VI分子量: 12641.998 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)   Saccharomyces cerevisiae (パン酵母) / 参照: UniProt: P00427 Saccharomyces cerevisiae (パン酵母) / 参照: UniProt: P00427 |
| #7: タンパク質 |  シトクロムcオキシダーゼ / Cytochrome c oxidase polypeptide VII シトクロムcオキシダーゼ / Cytochrome c oxidase polypeptide VII分子量: 6811.154 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)   Saccharomyces cerevisiae (パン酵母) / 参照: UniProt: P10174 Saccharomyces cerevisiae (パン酵母) / 参照: UniProt: P10174 |
| #8: タンパク質 |  シトクロムcオキシダーゼ / Cytochrome c oxidase polypeptide VIII シトクロムcオキシダーゼ / Cytochrome c oxidase polypeptide VIII分子量: 5737.735 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)   Saccharomyces cerevisiae (パン酵母) / 参照: UniProt: P04039 Saccharomyces cerevisiae (パン酵母) / 参照: UniProt: P04039 |
| #9: タンパク質 |  シトクロムcオキシダーゼ / Cytochrome c oxidase polypeptide VIIA シトクロムcオキシダーゼ / Cytochrome c oxidase polypeptide VIIA分子量: 6471.684 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)   Saccharomyces cerevisiae (パン酵母) / 参照: UniProt: P07255 Saccharomyces cerevisiae (パン酵母) / 参照: UniProt: P07255 |
-非ポリマー , 6種, 17分子 










| #10: 化合物 | ChemComp-CA / | ||||||
|---|---|---|---|---|---|---|---|
| #11: 化合物 | ChemComp-MG / | ||||||
| #12: 化合物 |  銅 銅#13: 化合物 |  Heme A Heme A#14: 化合物 | ChemComp-PEF /  ホスファチジルエタノールアミン ホスファチジルエタノールアミン#15: 化合物 | ChemComp-ZN / | |
-詳細
| 研究の焦点であるリガンドがあるか | N |
|---|
-実験情報
-実験
| 実験 | 手法:  電子顕微鏡法 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法:  単粒子再構成法 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 | 名称: Complex IV シトクロムcオキシダーゼ / タイプ: COMPLEX / Entity ID: #1-#9 / 由来: NATURAL シトクロムcオキシダーゼ / タイプ: COMPLEX / Entity ID: #1-#9 / 由来: NATURAL |
|---|---|
| 由来(天然) | 生物種:   Saccharomyces cerevisiae (パン酵母) Saccharomyces cerevisiae (パン酵母) |
| 緩衝液 | pH: 7 |
| 試料 | 包埋: NO / シャドウイング: NO / 染色 : NO / 凍結 : NO / 凍結 : YES : YES |
急速凍結 | 凍結剤: ETHANE |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: FEI TITAN KRIOS |
| 電子銃 | 電子線源 : :  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM |
| 電子レンズ | モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 3200 nm / 最小 デフォーカス(公称値): 1000 nm Bright-field microscopy / 最大 デフォーカス(公称値): 3200 nm / 最小 デフォーカス(公称値): 1000 nm |
| 撮影 | 電子線照射量: 1.011775103 e/Å2 フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) |
- 解析
解析
| ソフトウェア | 名称: PHENIX / バージョン: 1.19.2_4158: / 分類: 精密化 | ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
CTF補正 | タイプ: NONE | ||||||||||||||||||||||||
3次元再構成 | 解像度: 2.94 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 247631 / 対称性のタイプ: POINT | ||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー




 PDBj
PDBj







