+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 8d3i | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Crystal structure of human Apoptosis-Inducing Factor (AIF) W196A mutant complexed with quinolin-4-amine | ||||||||||||
 要素 要素 | (Apoptosis-inducing factor 1, ... ) x 2 ) x 2 | ||||||||||||
 キーワード キーワード |  OXIDOREDUCTASE (酸化還元酵素) / mitochondrial import / OXIDOREDUCTASE (酸化還元酵素) / mitochondrial import /  oxidative phosphorylation (酸化的リン酸化) / oxidative phosphorylation (酸化的リン酸化) /  SAXS (X線小角散乱) SAXS (X線小角散乱) | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 酸化還元酵素; その他が電子受容体 / 酸化還元酵素; その他が電子受容体 /  regulation of apoptotic DNA fragmentation / mitochondrial respiratory chain complex assembly / NAD(P)H oxidase H2O2-forming activity / poly-ADP-D-ribose binding / cellular response to aldosterone / positive regulation of necroptotic process / protein import into mitochondrial intermembrane space / chromosome condensation / oxidoreductase activity, acting on NAD(P)H ... regulation of apoptotic DNA fragmentation / mitochondrial respiratory chain complex assembly / NAD(P)H oxidase H2O2-forming activity / poly-ADP-D-ribose binding / cellular response to aldosterone / positive regulation of necroptotic process / protein import into mitochondrial intermembrane space / chromosome condensation / oxidoreductase activity, acting on NAD(P)H ... 酸化還元酵素; その他が電子受容体 / 酸化還元酵素; その他が電子受容体 /  regulation of apoptotic DNA fragmentation / mitochondrial respiratory chain complex assembly / NAD(P)H oxidase H2O2-forming activity / poly-ADP-D-ribose binding / cellular response to aldosterone / positive regulation of necroptotic process / protein import into mitochondrial intermembrane space / chromosome condensation / oxidoreductase activity, acting on NAD(P)H / response to L-glutamate / regulation of apoptotic DNA fragmentation / mitochondrial respiratory chain complex assembly / NAD(P)H oxidase H2O2-forming activity / poly-ADP-D-ribose binding / cellular response to aldosterone / positive regulation of necroptotic process / protein import into mitochondrial intermembrane space / chromosome condensation / oxidoreductase activity, acting on NAD(P)H / response to L-glutamate /  NADH dehydrogenase activity / mitochondrial respiratory chain complex I assembly / intrinsic apoptotic signaling pathway in response to endoplasmic reticulum stress / cellular response to nitric oxide / FAD binding / cellular response to estradiol stimulus / response to ischemia / neuron differentiation / NADH dehydrogenase activity / mitochondrial respiratory chain complex I assembly / intrinsic apoptotic signaling pathway in response to endoplasmic reticulum stress / cellular response to nitric oxide / FAD binding / cellular response to estradiol stimulus / response to ischemia / neuron differentiation /  ミトコンドリア / response to toxic substance / cellular response to hydrogen peroxide / activation of cysteine-type endopeptidase activity involved in apoptotic process / positive regulation of neuron apoptotic process / cellular response to hypoxia / neuron apoptotic process / ミトコンドリア / response to toxic substance / cellular response to hydrogen peroxide / activation of cysteine-type endopeptidase activity involved in apoptotic process / positive regulation of neuron apoptotic process / cellular response to hypoxia / neuron apoptotic process /  ミトコンドリア内膜 / ミトコンドリア内膜 /  protein dimerization activity / positive regulation of apoptotic process / apoptotic process / perinuclear region of cytoplasm / protein dimerization activity / positive regulation of apoptotic process / apoptotic process / perinuclear region of cytoplasm /  ミトコンドリア / ミトコンドリア /  DNA binding / DNA binding /  細胞核 / 細胞核 /  細胞質基質 / 細胞質基質 /  細胞質 細胞質類似検索 - 分子機能 | ||||||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2.65 Å 分子置換 / 解像度: 2.65 Å | ||||||||||||
 データ登録者 データ登録者 | Brosey, C.A. / Tainer, J.A. | ||||||||||||
| 資金援助 |  米国, 3件 米国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Nat.Chem.Biol. / 年: 2024 ジャーナル: Nat.Chem.Biol. / 年: 2024タイトル: Chemical screening by time-resolved X-ray scattering to discover allosteric probes. 著者: Brosey, C.A. / Link, T.M. / Shen, R. / Moiani, D. / Burnett, K. / Hura, G.L. / Jones, D.E. / Tainer, J.A. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  8d3i.cif.gz 8d3i.cif.gz | 579 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb8d3i.ent.gz pdb8d3i.ent.gz | 396.3 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  8d3i.json.gz 8d3i.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/d3/8d3i https://data.pdbj.org/pub/pdb/validation_reports/d3/8d3i ftp://data.pdbj.org/pub/pdb/validation_reports/d3/8d3i ftp://data.pdbj.org/pub/pdb/validation_reports/d3/8d3i | HTTPS FTP |
|---|
-関連構造データ
| 関連構造データ |  8d3eC  8d3gC  8d3hC  8d3jC  8d3kC  8d3nC  8d3oC  5kvhS S: 精密化の開始モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 単位格子 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 非結晶学的対称性 (NCS) | NCSドメイン:
NCSドメイン領域:
NCS oper: (Code: givenMatrix: (-0.539998331022, -0.84113386077, -0.0299270907284), (-0.841452857521, 0.540320470331, -0.00329816783788), (0.018944420385, 0.0234012308828, -0.999546642898)ベクター: ...NCS oper: (Code: given Matrix: (-0.539998331022, -0.84113386077, -0.0299270907284), ベクター  : : |
- 要素
要素
-Apoptosis-inducing factor 1, ... , 2種, 2分子 AB
| #1: タンパク質 |  / Programmed cell death protein 8 / Programmed cell death protein 8分子量: 59355.434 Da / 分子数: 1 / 変異: W196A / 由来タイプ: 組換発現 / 由来: (組換発現)   Homo sapiens (ヒト) / 遺伝子: AIFM1, AIF, PDCD8 / 発現宿主: Homo sapiens (ヒト) / 遺伝子: AIFM1, AIF, PDCD8 / 発現宿主:   Escherichia coli BL21(DE3) (大腸菌) / Variant (発現宿主): Rosetta2 Escherichia coli BL21(DE3) (大腸菌) / Variant (発現宿主): Rosetta2参照: UniProt: O95831,  酸化還元酵素; その他が電子受容体 酸化還元酵素; その他が電子受容体 |
|---|---|
| #2: タンパク質 |  / Programmed cell death protein 8 / Programmed cell death protein 8分子量: 59371.434 Da / 分子数: 1 / 変異: W196A / 由来タイプ: 組換発現 / 由来: (組換発現)   Homo sapiens (ヒト) / 遺伝子: AIFM1, AIF, PDCD8 / 発現宿主: Homo sapiens (ヒト) / 遺伝子: AIFM1, AIF, PDCD8 / 発現宿主:   Escherichia coli BL21(DE3) (大腸菌) / Variant (発現宿主): Rosetta2 Escherichia coli BL21(DE3) (大腸菌) / Variant (発現宿主): Rosetta2参照: UniProt: O95831,  酸化還元酵素; その他が電子受容体 酸化還元酵素; その他が電子受容体 |
-非ポリマー , 5種, 60分子 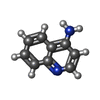








| #3: 化合物 |  4-アミノキノリン 4-アミノキノリン#4: 化合物 | ChemComp-EDO / |  エチレングリコール エチレングリコール#5: 化合物 |  フラビンアデニンジヌクレオチド フラビンアデニンジヌクレオチド#6: 化合物 |  硫酸塩 硫酸塩#7: 水 | ChemComp-HOH / |  水 水 |
|---|
-詳細
| 研究の焦点であるリガンドがあるか | Y |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.67 Å3/Da / 溶媒含有率: 54 % |
|---|---|
結晶化 | 温度: 295 K / 手法: 蒸気拡散法, ハンギングドロップ法 / pH: 7.5 / 詳細: 0.1 M Tris, pH 8.5, 0.32 M Na2SO4, 18% PEG3350 |
-データ収集
| 回折 | 平均測定温度: 100 K / Serial crystal experiment: N |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  NSLS-II NSLS-II  / ビームライン: 17-ID-2 / 波長: 0.9793 Å / ビームライン: 17-ID-2 / 波長: 0.9793 Å |
| 検出器 | タイプ: DECTRIS EIGER X 16M / 検出器: PIXEL / 日付: 2020年11月2日 |
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長 : 0.9793 Å / 相対比: 1 : 0.9793 Å / 相対比: 1 |
| 反射 | 解像度: 2.65→29.49 Å / Num. obs: 37931 / % possible obs: 99.9 % / 冗長度: 11 % / Biso Wilson estimate: 49.64 Å2 / CC1/2: 0.998 / Rmerge(I) obs: 0.129 / Rpim(I) all: 0.04 / Rrim(I) all: 0.135 / Net I/σ(I): 15.4 |
| 反射 シェル | 解像度: 2.65→2.77 Å / Rmerge(I) obs: 0.925 / Mean I/σ(I) obs: 2.6 / Num. unique obs: 4578 / CC1/2: 0.865 / Rpim(I) all: 0.287 / Rrim(I) all: 0.969 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法 : :  分子置換 分子置換開始モデル: 5KVH 解像度: 2.65→29.49 Å / SU ML: 0.328 / 交差検証法: FREE R-VALUE / σ(F): 1.34 / 位相誤差: 25.7679 立体化学のターゲット値: GeoStd + Monomer Library + CDL v1.2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | 減衰半径: 0.9 Å / VDWプローブ半径: 1.11 Å / 溶媒モデル: FLAT BULK SOLVENT MODEL | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 55.34 Å2 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 2.65→29.49 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine LS restraints NCS | タイプ: Torsion NCS / Rms dev position: 0.495592137127 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLS | 手法: refined / Refine-ID: X-RAY DIFFRACTION
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLSグループ | Refine-ID: X-RAY DIFFRACTION
|
 ムービー
ムービー コントローラー
コントローラー



 PDBj
PDBj






