| 登録構造単位 | A: Motile sperm domain-containing protein 2
B: Motile sperm domain-containing protein 2
C: Motile sperm domain-containing protein 2
D: Motile sperm domain-containing protein 2
E: Motile sperm domain-containing protein 2
F: Motile sperm domain-containing protein 2
G: Oxysterol-binding protein-related protein 1
H: Oxysterol-binding protein-related protein 1
I: Oxysterol-binding protein-related protein 1
J: Oxysterol-binding protein-related protein 1
K: Oxysterol-binding protein-related protein 1
ヘテロ分子
| 分子量 (理論値) | 分子数 |
|---|
| 合計 (水以外) | 159,233 | 30 |
|---|
| ポリマ- | 157,229 | 11 |
|---|
| 非ポリマー | 2,005 | 19 |
|---|
| 水 | 4,864 | 270 |
|---|
|
|---|
| 1 | A: Motile sperm domain-containing protein 2
G: Oxysterol-binding protein-related protein 1
ヘテロ分子
| 分子量 (理論値) | 分子数 |
|---|
| 合計 (水以外) | 27,114 | 8 |
|---|
| ポリマ- | 26,592 | 2 |
|---|
| 非ポリマー | 522 | 6 |
|---|
| 水 | 18 | 1 |
|---|
| タイプ | 名称 | 対称操作 | 数 |
|---|
| identity operation | 1_555 | x,y,z | 1 |
| Buried area | 1310 Å2 |
|---|
| ΔGint | -14 kcal/mol |
|---|
| Surface area | 8240 Å2 |
|---|
| 手法 | PISA |
|---|
|
|---|
| 2 | B: Motile sperm domain-containing protein 2
H: Oxysterol-binding protein-related protein 1
ヘテロ分子
| 分子量 (理論値) | 分子数 |
|---|
| 合計 (水以外) | 26,798 | 4 |
|---|
| ポリマ- | 26,592 | 2 |
|---|
| 非ポリマー | 206 | 2 |
|---|
| 水 | 18 | 1 |
|---|
| タイプ | 名称 | 対称操作 | 数 |
|---|
| identity operation | 1_555 | x,y,z | 1 |
| Buried area | 1070 Å2 |
|---|
| ΔGint | -4 kcal/mol |
|---|
| Surface area | 8310 Å2 |
|---|
| 手法 | PISA |
|---|
|
|---|
| 3 | C: Motile sperm domain-containing protein 2
I: Oxysterol-binding protein-related protein 1
ヘテロ分子
| 分子量 (理論値) | 分子数 |
|---|
| 合計 (水以外) | 27,000 | 6 |
|---|
| ポリマ- | 26,592 | 2 |
|---|
| 非ポリマー | 408 | 4 |
|---|
| 水 | 36 | 2 |
|---|
| タイプ | 名称 | 対称操作 | 数 |
|---|
| identity operation | 1_555 | x,y,z | 1 |
| Buried area | 1290 Å2 |
|---|
| ΔGint | -15 kcal/mol |
|---|
| Surface area | 8290 Å2 |
|---|
| 手法 | PISA |
|---|
|
|---|
| 4 | D: Motile sperm domain-containing protein 2
J: Oxysterol-binding protein-related protein 1
ヘテロ分子
| 分子量 (理論値) | 分子数 |
|---|
| 合計 (水以外) | 26,706 | 3 |
|---|
| ポリマ- | 26,592 | 2 |
|---|
| 非ポリマー | 114 | 1 |
|---|
| 水 | 18 | 1 |
|---|
| タイプ | 名称 | 対称操作 | 数 |
|---|
| identity operation | 1_555 | x,y,z | 1 |
| Buried area | 1160 Å2 |
|---|
| ΔGint | -7 kcal/mol |
|---|
| Surface area | 8010 Å2 |
|---|
| 手法 | PISA |
|---|
|
|---|
| 5 | E: Motile sperm domain-containing protein 2
K: Oxysterol-binding protein-related protein 1
ヘテロ分子
| 分子量 (理論値) | 分子数 |
|---|
| 合計 (水以外) | 26,898 | 5 |
|---|
| ポリマ- | 26,592 | 2 |
|---|
| 非ポリマー | 306 | 3 |
|---|
| 水 | 36 | 2 |
|---|
| タイプ | 名称 | 対称操作 | 数 |
|---|
| identity operation | 1_555 | x,y,z | 1 |
| Buried area | 1170 Å2 |
|---|
| ΔGint | -23 kcal/mol |
|---|
| Surface area | 7960 Å2 |
|---|
| 手法 | PISA |
|---|
|
|---|
| 6 | F: Motile sperm domain-containing protein 2
ヘテロ分子
| 分子量 (理論値) | 分子数 |
|---|
| 合計 (水以外) | 24,717 | 4 |
|---|
| ポリマ- | 24,269 | 1 |
|---|
| 非ポリマー | 448 | 3 |
|---|
| 水 | 18 | 1 |
|---|
| タイプ | 名称 | 対称操作 | 数 |
|---|
| identity operation | 1_555 | x,y,z | 1 |
| Buried area | 910 Å2 |
|---|
| ΔGint | -16 kcal/mol |
|---|
| Surface area | 8100 Å2 |
|---|
| 手法 | PISA |
|---|
|
|---|
| 単位格子 | | Length a, b, c (Å) | 126.874, 126.874, 184.449 |
|---|
| Angle α, β, γ (deg.) | 90.000, 90.000, 120.000 |
|---|
| Int Tables number | 178 |
|---|
| Space group name H-M | P6122 |
|---|
|
|---|
| Components on special symmetry positions | | ID | モデル | 要素 |
|---|
| 1 | 1 | F-629- HOH | | 2 | 1 | F-646- HOH |
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード PROTEIN BINDING (タンパク質) /
PROTEIN BINDING (タンパク質) /  Membrane contact sites /
Membrane contact sites /  FFAT motif / MSP domain /
FFAT motif / MSP domain /  Endoplasmic reticulum (小胞体)
Endoplasmic reticulum (小胞体) 機能・相同性情報
機能・相同性情報 FFAT motif binding / lipid droplet formation / organelle membrane contact site /
FFAT motif binding / lipid droplet formation / organelle membrane contact site /  : / perinuclear endoplasmic reticulum / bile acid biosynthetic process / RHOD GTPase cycle / positive regulation of neutrophil chemotaxis / positive regulation of monocyte chemotaxis ...endoplasmic reticulum-endosome membrane contact site /
: / perinuclear endoplasmic reticulum / bile acid biosynthetic process / RHOD GTPase cycle / positive regulation of neutrophil chemotaxis / positive regulation of monocyte chemotaxis ...endoplasmic reticulum-endosome membrane contact site /  FFAT motif binding / lipid droplet formation / organelle membrane contact site /
FFAT motif binding / lipid droplet formation / organelle membrane contact site /  : / perinuclear endoplasmic reticulum / bile acid biosynthetic process / RHOD GTPase cycle / positive regulation of neutrophil chemotaxis / positive regulation of monocyte chemotaxis /
: / perinuclear endoplasmic reticulum / bile acid biosynthetic process / RHOD GTPase cycle / positive regulation of neutrophil chemotaxis / positive regulation of monocyte chemotaxis /  cholesterol binding / Synthesis of bile acids and bile salts /
cholesterol binding / Synthesis of bile acids and bile salts /  細胞内膜系 / specific granule membrane / vesicle-mediated transport / MHC class II antigen presentation / cholesterol metabolic process /
細胞内膜系 / specific granule membrane / vesicle-mediated transport / MHC class II antigen presentation / cholesterol metabolic process /  phospholipid binding / protein homooligomerization /
phospholipid binding / protein homooligomerization /  走化性 / late endosome /
走化性 / late endosome /  エンドソーム / intracellular membrane-bounded organelle / Neutrophil degranulation / endoplasmic reticulum membrane /
エンドソーム / intracellular membrane-bounded organelle / Neutrophil degranulation / endoplasmic reticulum membrane /  小胞体 / extracellular exosome /
小胞体 / extracellular exosome /  生体膜 /
生体膜 /  細胞膜 /
細胞膜 /  細胞質基質
細胞質基質
 Homo sapiens (ヒト)
Homo sapiens (ヒト) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 2.25 Å
分子置換 / 解像度: 2.25 Å  データ登録者
データ登録者 フランス, 4件
フランス, 4件  引用
引用 ジャーナル: Embo J. / 年: 2020
ジャーナル: Embo J. / 年: 2020 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 6tqs.cif.gz
6tqs.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb6tqs.ent.gz
pdb6tqs.ent.gz PDB形式
PDB形式 6tqs.json.gz
6tqs.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/tq/6tqs
https://data.pdbj.org/pub/pdb/validation_reports/tq/6tqs ftp://data.pdbj.org/pub/pdb/validation_reports/tq/6tqs
ftp://data.pdbj.org/pub/pdb/validation_reports/tq/6tqs リンク
リンク 集合体
集合体





 要素
要素
 Homo sapiens (ヒト) / 遺伝子: MOSPD2 / 発現宿主:
Homo sapiens (ヒト) / 遺伝子: MOSPD2 / 発現宿主: 
 Escherichia coli BL21(DE3) (大腸菌) / 参照: UniProt: Q8NHP6
Escherichia coli BL21(DE3) (大腸菌) / 参照: UniProt: Q8NHP6
 Homo sapiens (ヒト) / 参照: UniProt: Q9BXW6
Homo sapiens (ヒト) / 参照: UniProt: Q9BXW6
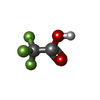











 グリセリン
グリセリン トリフルオロ酢酸
トリフルオロ酢酸 硫酸塩
硫酸塩 塩化物
塩化物 ジエチレングリコール
ジエチレングリコール ポリエチレングリコール
ポリエチレングリコール 水
水 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製
 シンクロトロン / サイト:
シンクロトロン / サイト:  SOLEIL
SOLEIL  / ビームライン: PROXIMA 2 / 波長: 0.98 Å
/ ビームライン: PROXIMA 2 / 波長: 0.98 Å : 0.98 Å / 相対比: 1
: 0.98 Å / 相対比: 1 
 分子置換
分子置換 解析
解析 :
:  分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー















 PDBj
PDBj
















