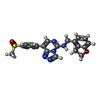
登録情報 データベース : PDB / ID : 6sfbタイトル EED in complex with a triazolopyrimidine Polycomb protein EED キーワード / 機能・相同性 生物種 Homo sapiens (ヒト)手法 / / / 解像度 : 1.52 Å データ登録者 Read, J.A. ジャーナル : Acs Chem.Biol. / 年 : 2019タイトル : Rapid Identification of Novel Allosteric PRC2 Inhibitors.著者 : Read, J.A. / Tart, J. / Rawlins, P.B. / Gregson, C. / Jones, K. / Gao, N. / Zhu, X. / Tomlinson, R. / Code, E. / Cheung, T. / Chen, H. / Kawatkar, S.P. / Bloecher, A. / Bagal, S. / O'Donovan, D.H. / Robinson, J. 履歴 登録 2019年8月1日 登録サイト / 処理サイト 改定 1.0 2019年9月25日 Provider / タイプ 改定 1.1 2019年10月30日 Group / Database references / カテゴリ / citation_authorItem _citation.journal_volume / _citation.page_first ... _citation.journal_volume / _citation.page_first / _citation.page_last / _citation.title / _citation_author.name 改定 2.0 2021年7月14日 Group Atomic model / Author supporting evidence ... Atomic model / Author supporting evidence / Data collection / Database references / Derived calculations / Non-polymer description / Refinement description / Structure summary カテゴリ atom_site / chem_comp ... atom_site / chem_comp / citation_author / entity / pdbx_entity_instance_feature / pdbx_entity_nonpoly / pdbx_nonpoly_scheme / pdbx_refine_tls_group / struct_site Item _atom_site.B_iso_or_equiv / _atom_site.Cartn_x ... _atom_site.B_iso_or_equiv / _atom_site.Cartn_x / _atom_site.Cartn_y / _atom_site.Cartn_z / _atom_site.auth_atom_id / _atom_site.auth_comp_id / _atom_site.label_atom_id / _atom_site.label_comp_id / _atom_site.type_symbol / _chem_comp.formula / _chem_comp.formula_weight / _chem_comp.id / _chem_comp.name / _citation_author.identifier_ORCID / _entity.formula_weight / _entity.pdbx_description / _pdbx_entity_instance_feature.auth_comp_id / _pdbx_entity_instance_feature.comp_id / _pdbx_entity_nonpoly.comp_id / _pdbx_entity_nonpoly.name / _pdbx_nonpoly_scheme.mon_id / _pdbx_nonpoly_scheme.pdb_mon_id / _pdbx_refine_tls_group.selection_details / _struct_site.details / _struct_site.pdbx_auth_comp_id 改定 2.1 2024年5月15日 Group / Database references / カテゴリ / chem_comp_bond / database_2Item / _database_2.pdbx_database_accession
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報 要素
要素
 キーワード
キーワード PROTEIN BINDING (タンパク質) / Methyltransferase EED PRC2 epigenetic H3K27 WD40 Inhibitor
PROTEIN BINDING (タンパク質) / Methyltransferase EED PRC2 epigenetic H3K27 WD40 Inhibitor 機能・相同性情報
機能・相同性情報 histone methyltransferase activity / Transcriptional Regulation by E2F6 /
histone methyltransferase activity / Transcriptional Regulation by E2F6 /  enzyme activator activity / transcription corepressor binding / PRC2 methylates histones and DNA / Regulation of PTEN gene transcription / Defective pyroptosis / PKMTs methylate histone lysines ...ESC/E(Z) complex / spinal cord development /
enzyme activator activity / transcription corepressor binding / PRC2 methylates histones and DNA / Regulation of PTEN gene transcription / Defective pyroptosis / PKMTs methylate histone lysines ...ESC/E(Z) complex / spinal cord development /  histone methyltransferase activity / Transcriptional Regulation by E2F6 /
histone methyltransferase activity / Transcriptional Regulation by E2F6 /  enzyme activator activity / transcription corepressor binding / PRC2 methylates histones and DNA / Regulation of PTEN gene transcription / Defective pyroptosis / PKMTs methylate histone lysines / Activation of anterior HOX genes in hindbrain development during early embryogenesis / HCMV Early Events /
enzyme activator activity / transcription corepressor binding / PRC2 methylates histones and DNA / Regulation of PTEN gene transcription / Defective pyroptosis / PKMTs methylate histone lysines / Activation of anterior HOX genes in hindbrain development during early embryogenesis / HCMV Early Events /  染色体 / Oxidative Stress Induced Senescence / negative regulation of DNA-templated transcription /
染色体 / Oxidative Stress Induced Senescence / negative regulation of DNA-templated transcription /  chromatin binding / negative regulation of transcription by RNA polymerase II /
chromatin binding / negative regulation of transcription by RNA polymerase II /  核質 / identical protein binding /
核質 / identical protein binding /  細胞核 /
細胞核 /  細胞質基質
細胞質基質
 Homo sapiens (ヒト)
Homo sapiens (ヒト) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 1.52 Å
分子置換 / 解像度: 1.52 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Acs Chem.Biol. / 年: 2019
ジャーナル: Acs Chem.Biol. / 年: 2019 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 6sfb.cif.gz
6sfb.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb6sfb.ent.gz
pdb6sfb.ent.gz PDB形式
PDB形式 6sfb.json.gz
6sfb.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/sf/6sfb
https://data.pdbj.org/pub/pdb/validation_reports/sf/6sfb ftp://data.pdbj.org/pub/pdb/validation_reports/sf/6sfb
ftp://data.pdbj.org/pub/pdb/validation_reports/sf/6sfb リンク
リンク 集合体
集合体


 要素
要素 / hEED / Embryonic ectoderm development protein / WD protein associating with integrin cytoplasmic ...hEED / Embryonic ectoderm development protein / WD protein associating with integrin cytoplasmic tails 1 / WAIT-1
/ hEED / Embryonic ectoderm development protein / WD protein associating with integrin cytoplasmic ...hEED / Embryonic ectoderm development protein / WD protein associating with integrin cytoplasmic tails 1 / WAIT-1
 Homo sapiens (ヒト) / 遺伝子: EED / 発現宿主:
Homo sapiens (ヒト) / 遺伝子: EED / 発現宿主: 
 Escherichia coli (大腸菌) / 参照: UniProt: O75530
Escherichia coli (大腸菌) / 参照: UniProt: O75530 グリセリン
グリセリン 水
水 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製
 シンクロトロン / サイト:
シンクロトロン / サイト:  Diamond
Diamond  / ビームライン: I04-1 / 波長: 0.987 Å
/ ビームライン: I04-1 / 波長: 0.987 Å : 0.987 Å / 相対比: 1
: 0.987 Å / 相対比: 1  解析
解析 :
:  分子置換 / 解像度: 1.52→22.15 Å / Cor.coef. Fo:Fc: 0.961 / Cor.coef. Fo:Fc free: 0.953 / SU R Cruickshank DPI: 0.076 / 交差検証法: THROUGHOUT / σ(F): 0 / SU R Blow DPI: 0.081 / SU Rfree Blow DPI: 0.077 / SU Rfree Cruickshank DPI: 0.074
分子置換 / 解像度: 1.52→22.15 Å / Cor.coef. Fo:Fc: 0.961 / Cor.coef. Fo:Fc free: 0.953 / SU R Cruickshank DPI: 0.076 / 交差検証法: THROUGHOUT / σ(F): 0 / SU R Blow DPI: 0.081 / SU Rfree Blow DPI: 0.077 / SU Rfree Cruickshank DPI: 0.074  ムービー
ムービー コントローラー
コントローラー













 PDBj
PDBj