登録情報 データベース : PDB / ID : 6ru9タイトル THE 3D STRUCTURE OF [NIFESE] HYDROGENASE G491A VARIANT FROM DESULFOVIBRIO VULGARIS HILDENBOROUGH AT 1.36 ANGSTROM RESOLUTION (Periplasmic [NiFeSe] hydrogenase, ...) x 2 キーワード / / / 機能・相同性 分子機能 ドメイン・相同性 構成要素
/ / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / 生物種 Desulfovibrio vulgaris str. Hildenborough (バクテリア)手法 / / / 解像度 : 1.355 Å データ登録者 Matias, P.M. / Zacarias, S. / Pereita, I. 資金援助 組織 認可番号 国 Fundacao para a Ciencia e a Tecnologia PTDC/BBB-BEP/2885/2014 Fundacao para a Ciencia e a Tecnologia SFRH/BD/100314/2014
ジャーナル : Acs Catalysis / 年 : 2019タイトル : A Hydrophilic Channel Is Involved in Oxidative Inactivation of a [NiFeSe] Hydrogenase著者 : Zacarias, S. / Temporao, A. / del Barrio, M. / Fourmond, V. / Leger, C. / Matias, P.M. / Pereira, I.A.C. 履歴 登録 2019年5月27日 登録サイト / 処理サイト 改定 1.0 2019年9月4日 Provider / タイプ 改定 1.1 2024年1月24日 Group Data collection / Database references ... Data collection / Database references / Derived calculations / Refinement description カテゴリ chem_comp_atom / chem_comp_bond ... chem_comp_atom / chem_comp_bond / database_2 / pdbx_initial_refinement_model / pdbx_struct_conn_angle / struct_conn Item _database_2.pdbx_DOI / _database_2.pdbx_database_accession ... _database_2.pdbx_DOI / _database_2.pdbx_database_accession / _pdbx_struct_conn_angle.ptnr1_auth_seq_id / _pdbx_struct_conn_angle.ptnr3_auth_seq_id / _pdbx_struct_conn_angle.value / _struct_conn.pdbx_dist_value / _struct_conn.ptnr2_auth_seq_id
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード OXIDOREDUCTASE (酸化還元酵素) / NIFESE-SITE / H2 CLEAVAGE/PRODUCTION / O2 TOLERANCE
OXIDOREDUCTASE (酸化還元酵素) / NIFESE-SITE / H2 CLEAVAGE/PRODUCTION / O2 TOLERANCE 機能・相同性情報
機能・相同性情報 シトクロムc3ヒドロゲナーゼ /
シトクロムc3ヒドロゲナーゼ /  フェレドキシンヒドロゲナーゼ /
フェレドキシンヒドロゲナーゼ /  cytochrome-c3 hydrogenase activity /
cytochrome-c3 hydrogenase activity /  ferredoxin hydrogenase complex /
ferredoxin hydrogenase complex /  ferredoxin hydrogenase activity / 3 iron, 4 sulfur cluster binding / nickel cation binding / 4 iron, 4 sulfur cluster binding /
ferredoxin hydrogenase activity / 3 iron, 4 sulfur cluster binding / nickel cation binding / 4 iron, 4 sulfur cluster binding /  ペリプラズム /
ペリプラズム /  metal ion binding
metal ion binding
 Desulfovibrio vulgaris str. Hildenborough (バクテリア)
Desulfovibrio vulgaris str. Hildenborough (バクテリア) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 1.355 Å
分子置換 / 解像度: 1.355 Å  データ登録者
データ登録者 ポルトガル, 2件
ポルトガル, 2件  引用
引用 ジャーナル: Acs Catalysis / 年: 2019
ジャーナル: Acs Catalysis / 年: 2019 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 6ru9.cif.gz
6ru9.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb6ru9.ent.gz
pdb6ru9.ent.gz PDB形式
PDB形式 6ru9.json.gz
6ru9.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/ru/6ru9
https://data.pdbj.org/pub/pdb/validation_reports/ru/6ru9 ftp://data.pdbj.org/pub/pdb/validation_reports/ru/6ru9
ftp://data.pdbj.org/pub/pdb/validation_reports/ru/6ru9 リンク
リンク 集合体
集合体
 要素
要素
 Desulfovibrio vulgaris str. Hildenborough (バクテリア)
Desulfovibrio vulgaris str. Hildenborough (バクテリア)
 Desulfovibrio vulgaris str. Hildenborough (バクテリア)
Desulfovibrio vulgaris str. Hildenborough (バクテリア) フェレドキシンヒドロゲナーゼ
フェレドキシンヒドロゲナーゼ
 Desulfovibrio vulgaris str. Hildenborough (バクテリア)
Desulfovibrio vulgaris str. Hildenborough (バクテリア)
 Desulfovibrio vulgaris str. Hildenborough (バクテリア)
Desulfovibrio vulgaris str. Hildenborough (バクテリア) フェレドキシンヒドロゲナーゼ
フェレドキシンヒドロゲナーゼ
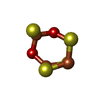













 鉄・硫黄クラスター
鉄・硫黄クラスター ニッケル
ニッケル 硫化水素
硫化水素 グリセリン
グリセリン 水
水 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製
 シンクロトロン / サイト:
シンクロトロン / サイト:  Diamond
Diamond  / ビームライン: I02 / 波長: 0.9795 Å
/ ビームライン: I02 / 波長: 0.9795 Å : 0.9795 Å / 相対比: 1
: 0.9795 Å / 相対比: 1 
 分子置換
分子置換 解析
解析 :
:  分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー

























 PDBj
PDBj












