+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 6ga8 | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | BACTERIORHODOPSIN, 330 FS STATE, REAL-SPACE REFINED AGAINST 15% EXTRAPOLATED STRUCTURE FACTORS | ||||||
 要素 要素 | Bacteriorhodopsin バクテリオロドプシン バクテリオロドプシン | ||||||
 キーワード キーワード |  MEMBRANE PROTEIN (膜タンパク質) / MEMBRANE PROTEIN (膜タンパク質) /  proton pump (プロトンポンプ) / time-resolved crystallography / proton pump (プロトンポンプ) / time-resolved crystallography /  free-electron laser (自由電子レーザー) free-electron laser (自由電子レーザー) | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 photoreceptor activity / photoreceptor activity /  phototransduction / proton transmembrane transport / monoatomic ion channel activity / phototransduction / proton transmembrane transport / monoatomic ion channel activity /  細胞膜 細胞膜類似検索 - 分子機能 | ||||||
| 生物種 |   Halobacterium salinarum (好塩性) Halobacterium salinarum (好塩性) | ||||||
| 手法 |  X線回折 / X線回折 /  自由電子レーザー / 解像度: 1.8 Å 自由電子レーザー / 解像度: 1.8 Å | ||||||
 データ登録者 データ登録者 | Nass Kovacs, G. / Colletier, J.-P. / Gruenbein, M.L. / Stensitzki, T. / Batyuk, A. / Carbajo, S. / Doak, R.B. / Ehrenberg, D. / Foucar, L. / Gasper, R. ...Nass Kovacs, G. / Colletier, J.-P. / Gruenbein, M.L. / Stensitzki, T. / Batyuk, A. / Carbajo, S. / Doak, R.B. / Ehrenberg, D. / Foucar, L. / Gasper, R. / Gorel, A. / Hilpert, M. / Kloos, M. / Koglin, J. / Reinstein, J. / Roome, C.M. / Schlesinger, R. / Seaberg, M. / Shoeman, R.L. / Stricker, M. / Boutet, S. / Haacke, S. / Heberle, J. / Domratcheva, T. / Schlichting, I. | ||||||
| 資金援助 | 1件
| ||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2019 ジャーナル: Nat Commun / 年: 2019タイトル: Three-dimensional view of ultrafast dynamics in photoexcited bacteriorhodopsin. 著者: Nass Kovacs, G. / Colletier, J.P. / Grunbein, M.L. / Yang, Y. / Stensitzki, T. / Batyuk, A. / Carbajo, S. / Doak, R.B. / Ehrenberg, D. / Foucar, L. / Gasper, R. / Gorel, A. / Hilpert, M. / ...著者: Nass Kovacs, G. / Colletier, J.P. / Grunbein, M.L. / Yang, Y. / Stensitzki, T. / Batyuk, A. / Carbajo, S. / Doak, R.B. / Ehrenberg, D. / Foucar, L. / Gasper, R. / Gorel, A. / Hilpert, M. / Kloos, M. / Koglin, J.E. / Reinstein, J. / Roome, C.M. / Schlesinger, R. / Seaberg, M. / Shoeman, R.L. / Stricker, M. / Boutet, S. / Haacke, S. / Heberle, J. / Heyne, K. / Domratcheva, T. / Barends, T.R.M. / Schlichting, I. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  6ga8.cif.gz 6ga8.cif.gz | 93.8 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb6ga8.ent.gz pdb6ga8.ent.gz | 79.2 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  6ga8.json.gz 6ga8.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/ga/6ga8 https://data.pdbj.org/pub/pdb/validation_reports/ga/6ga8 ftp://data.pdbj.org/pub/pdb/validation_reports/ga/6ga8 ftp://data.pdbj.org/pub/pdb/validation_reports/ga/6ga8 | HTTPS FTP |
|---|
-関連構造データ
| 関連構造データ |  6ga1C  6ga2C  6ga3C  6ga4C  6ga5C  6ga6C  6ga7C  6ga9C  6gaaC  6gabC  6gacC  6gadC  6gaeC  6gafC  6gagC  6gahC  6gaiC  6rmkC C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| |||||||||
| 単位格子 |
| |||||||||
| Components on special symmetry positions |
|
- 要素
要素
-タンパク質 , 1種, 1分子 A
| #1: タンパク質 |  バクテリオロドプシン / BR / Bacterioopsin / BO バクテリオロドプシン / BR / Bacterioopsin / BO分子量: 26929.500 Da / 分子数: 1 / 由来タイプ: 組換発現 由来: (組換発現)   Halobacterium salinarum (strain ATCC 700922 / JCM 11081 / NRC-1) (好塩性) Halobacterium salinarum (strain ATCC 700922 / JCM 11081 / NRC-1) (好塩性)株: ATCC 700922 / JCM 11081 / NRC-1 / 遺伝子: bop, VNG_1467G / 発現宿主:   Halobacterium salinarum (好塩性) / 参照: UniProt: P02945 Halobacterium salinarum (好塩性) / 参照: UniProt: P02945 |
|---|
-非ポリマー , 11種, 53分子 
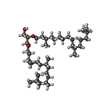



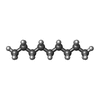












| #2: 化合物 | ChemComp-RET /  レチナール レチナール | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
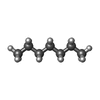
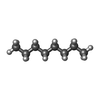
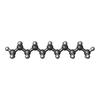
| #3: 化合物 | | #4: 化合物 | ChemComp-TRD / |  トリデカン トリデカン#5: 化合物 | ChemComp-D10 / |  デカン デカン#6: 化合物 | ChemComp-HP6 / |  Heptane Heptane#7: 化合物 |  オクタン オクタン#8: 化合物 | ChemComp-MYS / |  ペンタデカン ペンタデカン#9: 化合物 |  ウンデカン ウンデカン#10: 化合物 | ChemComp-DD9 / |  ノナン ノナン#11: 化合物 | ChemComp-C14 / |  テトラデカン テトラデカン#12: 水 | ChemComp-HOH / |  水 水 |
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.28 Å3/Da / 溶媒含有率: 46.15 % |
|---|---|
結晶化 | 温度: 293 K / 手法: 脂質キュービック相法 / pH: 5.6 / 詳細: 32% (w/v) PEG 2000, 0.1 M K2HPO4 /NaH2PO4 |
-データ収集
| 回折 | 平均測定温度: 293 K / Serial crystal experiment: Y |
|---|---|
| 放射光源 | 由来:  自由電子レーザー / サイト: 自由電子レーザー / サイト:  SLAC LCLS SLAC LCLS  / ビームライン: CXI / 波長: 1.26 Å / ビームライン: CXI / 波長: 1.26 Å |
| 検出器 | タイプ: CS-PAD CXI-2 / 検出器: PIXEL / 日付: 2017年7月25日 |
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長 : 1.26 Å / 相対比: 1 : 1.26 Å / 相対比: 1 |
| 反射 | 解像度: 1.8→21 Å / Num. obs: 22397 / % possible obs: 100 % / 冗長度: 139 % / R split: 0.119 / Net I/σ(I): 6.1 |
| 反射 シェル | 解像度: 1.8→1.9 Å / R split: 0.768 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 解像度: 1.8→20 Å / 交差検証法: NONE 詳細: A MOLECULE IS THE DARK STATE B MOLECULE WAS REAL-SPACE REFINED AGAINST MAP CALCULATED FROM EXTRAPOLATED STRUCTURE FACTORS (AT 15% OCCUPANCY) MODEL/MAP FIT (CCmask)=0.8365
| ||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 1.8→20 Å
|
 ムービー
ムービー コントローラー
コントローラー













 PDBj
PDBj

















