+ データを開く
データを開く
- 基本情報
基本情報
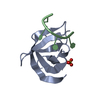
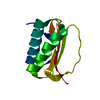
| 登録情報 | データベース: PDB / ID: 5ytt | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Crystal structure of YB1 cold-shock domain in complex with UCAUGU | |||||||||
 要素 要素 |
| |||||||||
 キーワード キーワード | RNA BINDING PROTEIN/RNA / YB1 /  cold-shock domain / CAUG / cold-shock domain / CAUG /  RNA BINDING PROTEIN (RNA結合タンパク質) / RNA BINDING PROTEIN-RNA complex RNA BINDING PROTEIN (RNA結合タンパク質) / RNA BINDING PROTEIN-RNA complex | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報tRNA transport / CRD-mediated mRNA stability complex / C5-methylcytidine-containing RNA reader activity / miRNA transport / negative regulation of striated muscle cell differentiation / RNA transport / Noncanonical activation of NOTCH3 / negative regulation of nuclear-transcribed mRNA catabolic process, deadenylation-dependent decay / CRD-mediated mRNA stabilization / protein localization to cytoplasmic stress granule ...tRNA transport / CRD-mediated mRNA stability complex / C5-methylcytidine-containing RNA reader activity / miRNA transport / negative regulation of striated muscle cell differentiation / RNA transport / Noncanonical activation of NOTCH3 / negative regulation of nuclear-transcribed mRNA catabolic process, deadenylation-dependent decay / CRD-mediated mRNA stabilization / protein localization to cytoplasmic stress granule /  messenger ribonucleoprotein complex / histone pre-mRNA 3'end processing complex / embryonic morphogenesis / U12-type spliceosomal complex / positive regulation of cytoplasmic translation / mRNA stabilization / cellular response to interleukin-7 / miRNA binding / mRNA Splicing - Minor Pathway / negative regulation of cellular senescence / Processing of Capped Intron-Containing Pre-mRNA / positive regulation of cell division / epidermis development / mRNA Splicing - Major Pathway / messenger ribonucleoprotein complex / histone pre-mRNA 3'end processing complex / embryonic morphogenesis / U12-type spliceosomal complex / positive regulation of cytoplasmic translation / mRNA stabilization / cellular response to interleukin-7 / miRNA binding / mRNA Splicing - Minor Pathway / negative regulation of cellular senescence / Processing of Capped Intron-Containing Pre-mRNA / positive regulation of cell division / epidermis development / mRNA Splicing - Major Pathway /  RNA splicing / RNA splicing /  P-body / SMAD2/SMAD3:SMAD4 heterotrimer regulates transcription / P-body / SMAD2/SMAD3:SMAD4 heterotrimer regulates transcription /  転写後修飾 / cytoplasmic stress granule / Interferon gamma signaling / sequence-specific double-stranded DNA binding / 転写後修飾 / cytoplasmic stress granule / Interferon gamma signaling / sequence-specific double-stranded DNA binding /  single-stranded DNA binding / single-stranded DNA binding /  GTPase binding / GTPase binding /  double-stranded DNA binding / double-stranded DNA binding /  遺伝子発現の調節 / in utero embryonic development / 遺伝子発現の調節 / in utero embryonic development /  nucleic acid binding / negative regulation of translation / nucleic acid binding / negative regulation of translation /  ribonucleoprotein complex / intracellular membrane-bounded organelle / ribonucleoprotein complex / intracellular membrane-bounded organelle /  mRNA binding / mRNA binding /  シナプス / シナプス /  chromatin binding / regulation of DNA-templated transcription / negative regulation of transcription by RNA polymerase II / chromatin binding / regulation of DNA-templated transcription / negative regulation of transcription by RNA polymerase II /  小胞体 / positive regulation of transcription by RNA polymerase II / 小胞体 / positive regulation of transcription by RNA polymerase II /  DNA binding / DNA binding /  RNA binding / extracellular exosome / extracellular region / RNA binding / extracellular exosome / extracellular region /  核質 / 核質 /  細胞核 / 細胞核 /  細胞膜 / 細胞膜 /  細胞質基質 / 細胞質基質 /  細胞質 細胞質類似検索 - 分子機能 | |||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト)synthetic construct (人工物) | |||||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 1.6 Å 分子置換 / 解像度: 1.6 Å | |||||||||
 データ登録者 データ登録者 | Yang, X. / Huang, Y. | |||||||||
| 資金援助 |  中国, 2件 中国, 2件
| |||||||||
 引用 引用 |  ジャーナル: J.Biol.Chem. / 年: 2019 ジャーナル: J.Biol.Chem. / 年: 2019タイトル: Crystal structure of a Y-box binding protein 1 (YB-1)-RNA complex reveals key features and residues interacting with RNA. 著者: Yang, X.J. / Zhu, H. / Mu, S.R. / Wei, W.J. / Yuan, X. / Wang, M. / Liu, Y. / Hui, J. / Huang, Y. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  5ytt.cif.gz 5ytt.cif.gz | 57.4 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb5ytt.ent.gz pdb5ytt.ent.gz | 38.3 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  5ytt.json.gz 5ytt.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/yt/5ytt https://data.pdbj.org/pub/pdb/validation_reports/yt/5ytt ftp://data.pdbj.org/pub/pdb/validation_reports/yt/5ytt ftp://data.pdbj.org/pub/pdb/validation_reports/yt/5ytt | HTTPS FTP |
|---|
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| 単位格子 |
|
- 要素
要素
| #1: タンパク質 | 分子量: 8997.026 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)   Homo sapiens (ヒト) / 遺伝子: YBX1, NSEP1, YB1 / 発現宿主: Homo sapiens (ヒト) / 遺伝子: YBX1, NSEP1, YB1 / 発現宿主:   Escherichia coli (大腸菌) / 参照: UniProt: P67809 Escherichia coli (大腸菌) / 参照: UniProt: P67809 | ||
|---|---|---|---|
| #2: RNA鎖 | 分子量: 2159.299 Da / 分子数: 1 / 由来タイプ: 合成 / 由来: (合成) synthetic construct (人工物) | ||
| #3: 化合物 |  硫酸塩 硫酸塩#4: 水 | ChemComp-HOH / |  水 水 |
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.1 Å3/Da / 溶媒含有率: 41.56 % |
|---|---|
結晶化 | 温度: 290 K / 手法: 蒸気拡散法, ハンギングドロップ法 / pH: 4.5 詳細: 30 % (v/v) 2-methyl-2,4-pentanediol, 0.1 M sodium acetate, pH 4.5, 25 % (w/v) PEG 1500 |
-データ収集
| 回折 | 平均測定温度: 100 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  SSRF SSRF  / ビームライン: BL17U1 / 波長: 0.97852 Å / ビームライン: BL17U1 / 波長: 0.97852 Å |
| 検出器 | タイプ: ADSC QUANTUM 315 / 検出器: CCD / 日付: 2014年12月12日 |
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長 : 0.97852 Å / 相対比: 1 : 0.97852 Å / 相対比: 1 |
| 反射 | 解像度: 1.6→50 Å / Num. obs: 11696 / % possible obs: 96.4 % / 冗長度: 9.2 % / Net I/σ(I): 26.6 |
| 反射 シェル | 解像度: 1.6→1.66 Å |
- 解析
解析
| ソフトウェア |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法 : :  分子置換 分子置換開始モデル: 3PF5 解像度: 1.6→30 Å / SU ML: 0.17 / 交差検証法: FREE R-VALUE / σ(F): 1.36 / 位相誤差: 22.64
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | 減衰半径: 0.9 Å / VDWプローブ半径: 1.11 Å | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 1.6→30 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLS | 手法: refined / Origin x: -6.2485 Å / Origin y: 23.1717 Å / Origin z: 36.1764 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLSグループ | Selection details: all |
 ムービー
ムービー コントローラー
コントローラー















 PDBj
PDBj







