| 登録情報 | データベース: PDB / ID: 5ms4
|
|---|
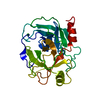
| タイトル | Kallikrein-related peptidase 8 leupeptin inhibitor complex |
|---|
 要素 要素 | - Kallikrein-8
 カリクレイン カリクレイン - LEUPEPTIN
 ロイペプチン ロイペプチン
|
|---|
 キーワード キーワード | Hydrolase/Inhibitor / trypsin-like serine protease / aldehyde inhibitor /  Ca2+ complex / synaptic remodeling / Hydrolase-inhibitor complex Ca2+ complex / synaptic remodeling / Hydrolase-inhibitor complex |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
 kallikrein 8 / Formation of the cornified envelope / regulation of synapse organization / keratinocyte proliferation / kallikrein 8 / Formation of the cornified envelope / regulation of synapse organization / keratinocyte proliferation /  serine protease inhibitor complex / neuron projection morphogenesis / serine protease inhibitor complex / neuron projection morphogenesis /  記憶 / response to wounding / serine-type endopeptidase activity / 記憶 / response to wounding / serine-type endopeptidase activity /  タンパク質分解 ... タンパク質分解 ... kallikrein 8 / Formation of the cornified envelope / regulation of synapse organization / keratinocyte proliferation / kallikrein 8 / Formation of the cornified envelope / regulation of synapse organization / keratinocyte proliferation /  serine protease inhibitor complex / neuron projection morphogenesis / serine protease inhibitor complex / neuron projection morphogenesis /  記憶 / response to wounding / serine-type endopeptidase activity / 記憶 / response to wounding / serine-type endopeptidase activity /  タンパク質分解 / タンパク質分解 /  extracellular space / extracellular region / extracellular space / extracellular region /  細胞質類似検索 - 分子機能 細胞質類似検索 - 分子機能 Serine proteases, trypsin family, histidine active site / Serine proteases, trypsin family, histidine active site /  Serine proteases, trypsin family, serine active site / Peptidase S1A, chymotrypsin family / Serine proteases, trypsin family, serine active site / Peptidase S1A, chymotrypsin family /  Serine proteases, trypsin family, histidine active site. / Serine proteases, trypsin family, histidine active site. /  Serine proteases, trypsin domain profile. / Serine proteases, trypsin domain profile. /  Serine proteases, trypsin family, serine active site. / Trypsin-like serine protease / Serine proteases, trypsin family, serine active site. / Trypsin-like serine protease /  Serine proteases, trypsin domain / Serine proteases, trypsin domain /  トリプシン / Trypsin-like serine proteases ... トリプシン / Trypsin-like serine proteases ... Serine proteases, trypsin family, histidine active site / Serine proteases, trypsin family, histidine active site /  Serine proteases, trypsin family, serine active site / Peptidase S1A, chymotrypsin family / Serine proteases, trypsin family, serine active site / Peptidase S1A, chymotrypsin family /  Serine proteases, trypsin family, histidine active site. / Serine proteases, trypsin family, histidine active site. /  Serine proteases, trypsin domain profile. / Serine proteases, trypsin domain profile. /  Serine proteases, trypsin family, serine active site. / Trypsin-like serine protease / Serine proteases, trypsin family, serine active site. / Trypsin-like serine protease /  Serine proteases, trypsin domain / Serine proteases, trypsin domain /  トリプシン / Trypsin-like serine proteases / トリプシン / Trypsin-like serine proteases /  Thrombin, subunit H / Peptidase S1, PA clan, chymotrypsin-like fold / Peptidase S1, PA clan / Thrombin, subunit H / Peptidase S1, PA clan, chymotrypsin-like fold / Peptidase S1, PA clan /  Βバレル / Mainly Beta類似検索 - ドメイン・相同性 Βバレル / Mainly Beta類似検索 - ドメイン・相同性 |
|---|
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト)
synthetic construct (人工物) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2.1 Å 分子置換 / 解像度: 2.1 Å |
|---|
 データ登録者 データ登録者 | Debela, M. / Magdolen, V. / Skala, W. / Bode, W. / Brandstetter, H. / Goettig, P. |
|---|
| 資金援助 |  オーストリア, 1件 オーストリア, 1件 | 組織 | 認可番号 | 国 |
|---|
| Austrian Science Fund | P25003-B21 |  オーストリア オーストリア |
|
|---|
 引用 引用 |  ジャーナル: Sci Rep / 年: 2018 ジャーナル: Sci Rep / 年: 2018
タイトル: Structural determinants of specificity and regulation of activity in the allosteric loop network of human KLK8/neuropsin.
著者: Debela, M. / Magdolen, V. / Skala, W. / Elsasser, B. / Schneider, E.L. / Craik, C.S. / Biniossek, M.L. / Schilling, O. / Bode, W. / Brandstetter, H. / Goettig, P. |
|---|
| 履歴 | | 登録 | 2016年12月30日 | 登録サイト: PDBE / 処理サイト: PDBE |
|---|
| 改定 1.0 | 2018年1月17日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2018年8月1日 | Group: Data collection / Database references / カテゴリ: citation / citation_author
Item: _citation.country / _citation.journal_abbrev ..._citation.country / _citation.journal_abbrev / _citation.journal_id_CSD / _citation.journal_id_ISSN / _citation.journal_volume / _citation.page_first / _citation.page_last / _citation.pdbx_database_id_DOI / _citation.pdbx_database_id_PubMed / _citation.title / _citation.year / _citation_author.name |
|---|
| 改定 2.0 | 2019年9月18日 | Group: Advisory / Atomic model ...Advisory / Atomic model / Data collection / Database references / Derived calculations / Non-polymer description / Polymer sequence / Source and taxonomy / Structure summary
カテゴリ: atom_site / chem_comp ...atom_site / chem_comp / entity / entity_poly / entity_poly_seq / pdbx_distant_solvent_atoms / pdbx_entity_nonpoly / pdbx_entity_src_syn / pdbx_molecule_features / pdbx_nonpoly_scheme / pdbx_poly_seq_scheme / pdbx_struct_assembly / pdbx_struct_assembly_gen / pdbx_struct_conn_angle / pdbx_struct_sheet_hbond / pdbx_validate_close_contact / pdbx_validate_polymer_linkage / pdbx_validate_rmsd_bond / pdbx_validate_symm_contact / struct_asym / struct_conn / struct_ref / struct_ref_seq / struct_sheet / struct_sheet_order / struct_sheet_range / struct_site / struct_site_gen
Item: _atom_site.B_iso_or_equiv / _atom_site.Cartn_x ..._atom_site.B_iso_or_equiv / _atom_site.Cartn_x / _atom_site.Cartn_y / _atom_site.Cartn_z / _atom_site.auth_asym_id / _atom_site.auth_atom_id / _atom_site.auth_comp_id / _atom_site.auth_seq_id / _atom_site.group_PDB / _atom_site.label_asym_id / _atom_site.label_atom_id / _atom_site.label_comp_id / _atom_site.label_entity_id / _atom_site.label_seq_id / _atom_site.type_symbol / _chem_comp.formula / _chem_comp.formula_weight / _chem_comp.id / _chem_comp.mon_nstd_flag / _chem_comp.name / _chem_comp.pdbx_synonyms / _chem_comp.type / _entity.formula_weight / _entity.pdbx_description / _entity.type / _pdbx_distant_solvent_atoms.auth_asym_id / _pdbx_distant_solvent_atoms.auth_seq_id / _pdbx_distant_solvent_atoms.neighbor_ligand_distance / _pdbx_distant_solvent_atoms.neighbor_macromolecule_distance / _pdbx_struct_assembly.oligomeric_count / _pdbx_struct_assembly.oligomeric_details / _pdbx_struct_assembly_gen.asym_id_list / _pdbx_struct_conn_angle.ptnr2_auth_seq_id / _pdbx_struct_conn_angle.ptnr2_label_asym_id / _pdbx_validate_close_contact.auth_seq_id_1 / _pdbx_validate_close_contact.auth_seq_id_2 / _pdbx_validate_symm_contact.auth_seq_id_1 / _pdbx_validate_symm_contact.auth_seq_id_2 |
|---|
| 改定 2.1 | 2019年10月9日 | Group: Data collection / Derived calculations / カテゴリ: pdbx_struct_conn_angle / struct_conn |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード Ca2+ complex / synaptic remodeling / Hydrolase-inhibitor complex
Ca2+ complex / synaptic remodeling / Hydrolase-inhibitor complex 機能・相同性情報
機能・相同性情報 kallikrein 8 / Formation of the cornified envelope / regulation of synapse organization / keratinocyte proliferation /
kallikrein 8 / Formation of the cornified envelope / regulation of synapse organization / keratinocyte proliferation /  serine protease inhibitor complex / neuron projection morphogenesis /
serine protease inhibitor complex / neuron projection morphogenesis /  記憶 / response to wounding / serine-type endopeptidase activity /
記憶 / response to wounding / serine-type endopeptidase activity /  タンパク質分解 ...
タンパク質分解 ... kallikrein 8 / Formation of the cornified envelope / regulation of synapse organization / keratinocyte proliferation /
kallikrein 8 / Formation of the cornified envelope / regulation of synapse organization / keratinocyte proliferation /  serine protease inhibitor complex / neuron projection morphogenesis /
serine protease inhibitor complex / neuron projection morphogenesis /  記憶 / response to wounding / serine-type endopeptidase activity /
記憶 / response to wounding / serine-type endopeptidase activity /  タンパク質分解 /
タンパク質分解 /  extracellular space / extracellular region /
extracellular space / extracellular region /  細胞質
細胞質
 Homo sapiens (ヒト)
Homo sapiens (ヒト) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 2.1 Å
分子置換 / 解像度: 2.1 Å  データ登録者
データ登録者 オーストリア, 1件
オーストリア, 1件  引用
引用 ジャーナル: Sci Rep / 年: 2018
ジャーナル: Sci Rep / 年: 2018 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 5ms4.cif.gz
5ms4.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb5ms4.ent.gz
pdb5ms4.ent.gz PDB形式
PDB形式 5ms4.json.gz
5ms4.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/ms/5ms4
https://data.pdbj.org/pub/pdb/validation_reports/ms/5ms4 ftp://data.pdbj.org/pub/pdb/validation_reports/ms/5ms4
ftp://data.pdbj.org/pub/pdb/validation_reports/ms/5ms4 リンク
リンク 集合体
集合体




 要素
要素 カリクレイン / hK8 / Neuropsin / NP / Ovasin / Serine protease 19 / Serine protease TADG-14 / Tumor-associated ...hK8 / Neuropsin / NP / Ovasin / Serine protease 19 / Serine protease TADG-14 / Tumor-associated differentially expressed gene 14 protein
カリクレイン / hK8 / Neuropsin / NP / Ovasin / Serine protease 19 / Serine protease TADG-14 / Tumor-associated ...hK8 / Neuropsin / NP / Ovasin / Serine protease 19 / Serine protease TADG-14 / Tumor-associated differentially expressed gene 14 protein
 Homo sapiens (ヒト) / 遺伝子: KLK8, NRPN, PRSS19, TADG14, UNQ283/PRO322 / 発現宿主:
Homo sapiens (ヒト) / 遺伝子: KLK8, NRPN, PRSS19, TADG14, UNQ283/PRO322 / 発現宿主: 
 Escherichia coli (大腸菌) / 参照: UniProt: O60259,
Escherichia coli (大腸菌) / 参照: UniProt: O60259,  kallikrein 8
kallikrein 8 ロイペプチン /
ロイペプチン /  ロイペプチン
ロイペプチン Tert-ブチルアルコール
Tert-ブチルアルコール 水
水 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製
 シンクロトロン / サイト:
シンクロトロン / サイト:  SLS
SLS  / ビームライン: X10SA / 波長: 1 Å
/ ビームライン: X10SA / 波長: 1 Å : 1 Å / 相対比: 1
: 1 Å / 相対比: 1 
 分子置換
分子置換 解析
解析 :
:  分子置換 / 解像度: 2.1→65.53 Å / 交差検証法: FREE R-VALUE / σ(F): 2.38 / 位相誤差: 28.91
分子置換 / 解像度: 2.1→65.53 Å / 交差検証法: FREE R-VALUE / σ(F): 2.38 / 位相誤差: 28.91  ムービー
ムービー コントローラー
コントローラー










 PDBj
PDBj