| 登録情報 | データベース: PDB / ID: 5jel
|
|---|
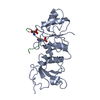
| タイトル | Phosphorylated TRIF in complex with IRF-3 |
|---|
 要素 要素 | - Interferon regulatory factor 3
 IRF3 IRF3 - Phosphorylated TRIF peptide
|
|---|
 キーワード キーワード |  IMMUNE SYSTEM (免疫系) / IMMUNE SYSTEM (免疫系) /  Innate Immunity (自然免疫系) / Innate Immunity (自然免疫系) /  Signaling Signaling |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
TICAM1 deficiency - HSE / TRAF3 deficiency - HSE / IRF3 mediated activation of type 1 IFN / MDA-5 signaling pathway / ripoptosome / MyD88-independent TLR4 cascade / macrophage apoptotic process / Toll Like Receptor 3 (TLR3) Cascade / TRIF-mediated programmed cell death / positive regulation of natural killer cell activation ...TICAM1 deficiency - HSE / TRAF3 deficiency - HSE / IRF3 mediated activation of type 1 IFN / MDA-5 signaling pathway / ripoptosome / MyD88-independent TLR4 cascade / macrophage apoptotic process / Toll Like Receptor 3 (TLR3) Cascade / TRIF-mediated programmed cell death / positive regulation of natural killer cell activation / TLR3-mediated TICAM1-dependent programmed cell death / cellular response to oxidised low-density lipoprotein particle stimulus / programmed necrotic cell death / positive regulation of myeloid dendritic cell cytokine production / Caspase activation via Death Receptors in the presence of ligand / toll-like receptor 3 signaling pathway / TRIF-dependent toll-like receptor signaling pathway / signal transduction involved in regulation of gene expression / RIP-mediated NFkB activation via ZBP1 / macrophage activation involved in immune response / IRF3-mediated induction of type I IFN / LRR FLII-interacting protein 1 (LRRFIP1) activates type I IFN production / positive regulation of cytokine production involved in inflammatory response / positive regulation of type I interferon-mediated signaling pathway / cGAS/STING signaling pathway / positive regulation of macrophage cytokine production /  転写 (生物学) / toll-like receptor 4 signaling pathway / TRAF6 mediated IRF7 activation / 転写 (生物学) / toll-like receptor 4 signaling pathway / TRAF6 mediated IRF7 activation /  Toll様受容体 / DNA-binding transcription activator activity / Toll様受容体 / DNA-binding transcription activator activity /  immune system process / type I interferon-mediated signaling pathway / cellular response to exogenous dsRNA / response to exogenous dsRNA / cytoplasmic pattern recognition receptor signaling pathway / B cell proliferation / antiviral innate immune response / immune system process / type I interferon-mediated signaling pathway / cellular response to exogenous dsRNA / response to exogenous dsRNA / cytoplasmic pattern recognition receptor signaling pathway / B cell proliferation / antiviral innate immune response /  オートファゴソーム / positive regulation of interferon-alpha production / regulation of protein-containing complex assembly / positive regulation of type I interferon production / positive regulation of autophagy / signaling adaptor activity / positive regulation of B cell proliferation / positive regulation of chemokine production / lipopolysaccharide-mediated signaling pathway / IRAK2 mediated activation of TAK1 complex upon TLR7/8 or 9 stimulation / TICAM1,TRAF6-dependent induction of TAK1 complex / TICAM1-dependent activation of IRF3/IRF7 / nitric oxide biosynthetic process / TRAF6-mediated induction of TAK1 complex within TLR4 complex / Regulation of innate immune responses to cytosolic DNA / positive regulation of interferon-beta production / TICAM1, RIP1-mediated IKK complex recruitment / Activation of IRF3, IRF7 mediated by TBK1, IKKε (IKBKE) / IKK complex recruitment mediated by RIP1 / Evasion by RSV of host interferon responses / positive regulation of protein ubiquitination / Negative regulators of DDX58/IFIH1 signaling / promoter-specific chromatin binding / apoptotic signaling pathway / DDX58/IFIH1-mediated induction of interferon-alpha/beta / ISG15 antiviral mechanism / cellular response to virus / DNA-binding transcription repressor activity, RNA polymerase II-specific / positive regulation of interleukin-6 production / SARS-CoV-1 activates/modulates innate immune responses / positive regulation of nitric oxide biosynthetic process / positive regulation of tumor necrosis factor production / Interferon gamma signaling / Interferon alpha/beta signaling / sequence-specific double-stranded DNA binding / TRAF3-dependent IRF activation pathway / オートファゴソーム / positive regulation of interferon-alpha production / regulation of protein-containing complex assembly / positive regulation of type I interferon production / positive regulation of autophagy / signaling adaptor activity / positive regulation of B cell proliferation / positive regulation of chemokine production / lipopolysaccharide-mediated signaling pathway / IRAK2 mediated activation of TAK1 complex upon TLR7/8 or 9 stimulation / TICAM1,TRAF6-dependent induction of TAK1 complex / TICAM1-dependent activation of IRF3/IRF7 / nitric oxide biosynthetic process / TRAF6-mediated induction of TAK1 complex within TLR4 complex / Regulation of innate immune responses to cytosolic DNA / positive regulation of interferon-beta production / TICAM1, RIP1-mediated IKK complex recruitment / Activation of IRF3, IRF7 mediated by TBK1, IKKε (IKBKE) / IKK complex recruitment mediated by RIP1 / Evasion by RSV of host interferon responses / positive regulation of protein ubiquitination / Negative regulators of DDX58/IFIH1 signaling / promoter-specific chromatin binding / apoptotic signaling pathway / DDX58/IFIH1-mediated induction of interferon-alpha/beta / ISG15 antiviral mechanism / cellular response to virus / DNA-binding transcription repressor activity, RNA polymerase II-specific / positive regulation of interleukin-6 production / SARS-CoV-1 activates/modulates innate immune responses / positive regulation of nitric oxide biosynthetic process / positive regulation of tumor necrosis factor production / Interferon gamma signaling / Interferon alpha/beta signaling / sequence-specific double-stranded DNA binding / TRAF3-dependent IRF activation pathway /  regulation of inflammatory response / DNA-binding transcription activator activity, RNA polymerase II-specific / regulation of apoptotic process / positive regulation of canonical NF-kappaB signal transduction / defense response to virus / cellular response to lipopolysaccharide / sequence-specific DNA binding / molecular adaptor activity / regulation of inflammatory response / DNA-binding transcription activator activity, RNA polymerase II-specific / regulation of apoptotic process / positive regulation of canonical NF-kappaB signal transduction / defense response to virus / cellular response to lipopolysaccharide / sequence-specific DNA binding / molecular adaptor activity /  エンドソーム / endosome membrane / DNA-binding transcription factor activity, RNA polymerase II-specific / エンドソーム / endosome membrane / DNA-binding transcription factor activity, RNA polymerase II-specific /  エンドソーム / エンドソーム /  炎症 / RNA polymerase II cis-regulatory region sequence-specific DNA binding / DNA-binding transcription factor activity / protein domain specific binding / 炎症 / RNA polymerase II cis-regulatory region sequence-specific DNA binding / DNA-binding transcription factor activity / protein domain specific binding /  自然免疫系 / apoptotic process / DNA damage response / 自然免疫系 / apoptotic process / DNA damage response /  クロマチン / positive regulation of gene expression / regulation of transcription by RNA polymerase II / クロマチン / positive regulation of gene expression / regulation of transcription by RNA polymerase II /  protein kinase binding / SARS-CoV-2 activates/modulates innate and adaptive immune responses / protein homodimerization activity / positive regulation of transcription by RNA polymerase II類似検索 - 分子機能 protein kinase binding / SARS-CoV-2 activates/modulates innate and adaptive immune responses / protein homodimerization activity / positive regulation of transcription by RNA polymerase II類似検索 - 分子機能 TIR domain-containing adapter molecule 1 /  TRIF, N-terminal / TRIF, N-terminal /  : / TRIF N-terminal domain / RHIM domain / RIP homotypic interaction motif / Tumour Suppressor Smad4 - #10 / : / TRIF N-terminal domain / RHIM domain / RIP homotypic interaction motif / Tumour Suppressor Smad4 - #10 /  Interferon regulatory factor-3 / Interferon-regulatory factor 3 / Interferon-regulatory factor 3 ...TIR domain-containing adapter molecule 1 / Interferon regulatory factor-3 / Interferon-regulatory factor 3 / Interferon-regulatory factor 3 ...TIR domain-containing adapter molecule 1 /  TRIF, N-terminal / TRIF, N-terminal /  : / TRIF N-terminal domain / RHIM domain / RIP homotypic interaction motif / Tumour Suppressor Smad4 - #10 / : / TRIF N-terminal domain / RHIM domain / RIP homotypic interaction motif / Tumour Suppressor Smad4 - #10 /  Interferon regulatory factor-3 / Interferon-regulatory factor 3 / Interferon-regulatory factor 3 / Interferon regulatory factor-3 / Interferon-regulatory factor 3 / Interferon-regulatory factor 3 /  Interferon regulatory factor, conserved site / IRF tryptophan pentad repeat DNA-binding domain signature. / Interferon regulatory factor transcription factor / Interferon regulatory factor, conserved site / IRF tryptophan pentad repeat DNA-binding domain signature. / Interferon regulatory factor transcription factor /  interferon regulatory factor / IRF tryptophan pentad repeat DNA-binding domain profile. / Interferon regulatory factor DNA-binding domain / SMAD-like domain superfamily / Tumour Suppressor Smad4 / TIR domain profile. / Toll/interleukin-1 receptor homology (TIR) domain / Toll/interleukin-1 receptor homology (TIR) domain superfamily / SMAD/FHA domain superfamily / Winged helix DNA-binding domain superfamily / Winged helix-like DNA-binding domain superfamily / interferon regulatory factor / IRF tryptophan pentad repeat DNA-binding domain profile. / Interferon regulatory factor DNA-binding domain / SMAD-like domain superfamily / Tumour Suppressor Smad4 / TIR domain profile. / Toll/interleukin-1 receptor homology (TIR) domain / Toll/interleukin-1 receptor homology (TIR) domain superfamily / SMAD/FHA domain superfamily / Winged helix DNA-binding domain superfamily / Winged helix-like DNA-binding domain superfamily /  サンドイッチ / Mainly Beta類似検索 - ドメイン・相同性 サンドイッチ / Mainly Beta類似検索 - ドメイン・相同性  Interferon regulatory factor 3 / TIR domain-containing adapter molecule 1類似検索 - 構成要素 Interferon regulatory factor 3 / TIR domain-containing adapter molecule 1類似検索 - 構成要素 |
|---|
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / 解像度: 1.6 Å シンクロトロン / 解像度: 1.6 Å |
|---|
 データ登録者 データ登録者 | Zhao, B. / Li, P. |
|---|
 引用 引用 |  ジャーナル: Proc.Natl.Acad.Sci.USA / 年: 2016 ジャーナル: Proc.Natl.Acad.Sci.USA / 年: 2016
タイトル: Structural basis for concerted recruitment and activation of IRF-3 by innate immune adaptor proteins.
著者: Zhao, B. / Shu, C. / Gao, X. / Sankaran, B. / Du, F. / Shelton, C.L. / Herr, A.B. / Ji, J.Y. / Li, P. |
|---|
| 履歴 | | 登録 | 2016年4月18日 | 登録サイト: RCSB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2016年6月15日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2016年6月29日 | Group: Database references |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード IMMUNE SYSTEM (免疫系) /
IMMUNE SYSTEM (免疫系) /  Innate Immunity (自然免疫系) /
Innate Immunity (自然免疫系) /  Signaling
Signaling 機能・相同性情報
機能・相同性情報 転写 (生物学) / toll-like receptor 4 signaling pathway / TRAF6 mediated IRF7 activation /
転写 (生物学) / toll-like receptor 4 signaling pathway / TRAF6 mediated IRF7 activation /  Toll様受容体 / DNA-binding transcription activator activity /
Toll様受容体 / DNA-binding transcription activator activity /  immune system process / type I interferon-mediated signaling pathway / cellular response to exogenous dsRNA / response to exogenous dsRNA / cytoplasmic pattern recognition receptor signaling pathway / B cell proliferation / antiviral innate immune response /
immune system process / type I interferon-mediated signaling pathway / cellular response to exogenous dsRNA / response to exogenous dsRNA / cytoplasmic pattern recognition receptor signaling pathway / B cell proliferation / antiviral innate immune response /  オートファゴソーム / positive regulation of interferon-alpha production / regulation of protein-containing complex assembly / positive regulation of type I interferon production / positive regulation of autophagy / signaling adaptor activity / positive regulation of B cell proliferation / positive regulation of chemokine production / lipopolysaccharide-mediated signaling pathway / IRAK2 mediated activation of TAK1 complex upon TLR7/8 or 9 stimulation / TICAM1,TRAF6-dependent induction of TAK1 complex / TICAM1-dependent activation of IRF3/IRF7 / nitric oxide biosynthetic process / TRAF6-mediated induction of TAK1 complex within TLR4 complex / Regulation of innate immune responses to cytosolic DNA / positive regulation of interferon-beta production / TICAM1, RIP1-mediated IKK complex recruitment / Activation of IRF3, IRF7 mediated by TBK1, IKKε (IKBKE) / IKK complex recruitment mediated by RIP1 / Evasion by RSV of host interferon responses / positive regulation of protein ubiquitination / Negative regulators of DDX58/IFIH1 signaling / promoter-specific chromatin binding / apoptotic signaling pathway / DDX58/IFIH1-mediated induction of interferon-alpha/beta / ISG15 antiviral mechanism / cellular response to virus / DNA-binding transcription repressor activity, RNA polymerase II-specific / positive regulation of interleukin-6 production / SARS-CoV-1 activates/modulates innate immune responses / positive regulation of nitric oxide biosynthetic process / positive regulation of tumor necrosis factor production / Interferon gamma signaling / Interferon alpha/beta signaling / sequence-specific double-stranded DNA binding / TRAF3-dependent IRF activation pathway /
オートファゴソーム / positive regulation of interferon-alpha production / regulation of protein-containing complex assembly / positive regulation of type I interferon production / positive regulation of autophagy / signaling adaptor activity / positive regulation of B cell proliferation / positive regulation of chemokine production / lipopolysaccharide-mediated signaling pathway / IRAK2 mediated activation of TAK1 complex upon TLR7/8 or 9 stimulation / TICAM1,TRAF6-dependent induction of TAK1 complex / TICAM1-dependent activation of IRF3/IRF7 / nitric oxide biosynthetic process / TRAF6-mediated induction of TAK1 complex within TLR4 complex / Regulation of innate immune responses to cytosolic DNA / positive regulation of interferon-beta production / TICAM1, RIP1-mediated IKK complex recruitment / Activation of IRF3, IRF7 mediated by TBK1, IKKε (IKBKE) / IKK complex recruitment mediated by RIP1 / Evasion by RSV of host interferon responses / positive regulation of protein ubiquitination / Negative regulators of DDX58/IFIH1 signaling / promoter-specific chromatin binding / apoptotic signaling pathway / DDX58/IFIH1-mediated induction of interferon-alpha/beta / ISG15 antiviral mechanism / cellular response to virus / DNA-binding transcription repressor activity, RNA polymerase II-specific / positive regulation of interleukin-6 production / SARS-CoV-1 activates/modulates innate immune responses / positive regulation of nitric oxide biosynthetic process / positive regulation of tumor necrosis factor production / Interferon gamma signaling / Interferon alpha/beta signaling / sequence-specific double-stranded DNA binding / TRAF3-dependent IRF activation pathway /  regulation of inflammatory response / DNA-binding transcription activator activity, RNA polymerase II-specific / regulation of apoptotic process / positive regulation of canonical NF-kappaB signal transduction / defense response to virus / cellular response to lipopolysaccharide / sequence-specific DNA binding / molecular adaptor activity /
regulation of inflammatory response / DNA-binding transcription activator activity, RNA polymerase II-specific / regulation of apoptotic process / positive regulation of canonical NF-kappaB signal transduction / defense response to virus / cellular response to lipopolysaccharide / sequence-specific DNA binding / molecular adaptor activity /  エンドソーム / endosome membrane / DNA-binding transcription factor activity, RNA polymerase II-specific /
エンドソーム / endosome membrane / DNA-binding transcription factor activity, RNA polymerase II-specific /  エンドソーム /
エンドソーム /  炎症 / RNA polymerase II cis-regulatory region sequence-specific DNA binding / DNA-binding transcription factor activity / protein domain specific binding /
炎症 / RNA polymerase II cis-regulatory region sequence-specific DNA binding / DNA-binding transcription factor activity / protein domain specific binding /  自然免疫系 / apoptotic process / DNA damage response /
自然免疫系 / apoptotic process / DNA damage response /  クロマチン / positive regulation of gene expression / regulation of transcription by RNA polymerase II /
クロマチン / positive regulation of gene expression / regulation of transcription by RNA polymerase II /  protein kinase binding / SARS-CoV-2 activates/modulates innate and adaptive immune responses / protein homodimerization activity / positive regulation of transcription by RNA polymerase II
protein kinase binding / SARS-CoV-2 activates/modulates innate and adaptive immune responses / protein homodimerization activity / positive regulation of transcription by RNA polymerase II
 Homo sapiens (ヒト)
Homo sapiens (ヒト) X線回折 /
X線回折 /  シンクロトロン / 解像度: 1.6 Å
シンクロトロン / 解像度: 1.6 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Proc.Natl.Acad.Sci.USA / 年: 2016
ジャーナル: Proc.Natl.Acad.Sci.USA / 年: 2016 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 5jel.cif.gz
5jel.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb5jel.ent.gz
pdb5jel.ent.gz PDB形式
PDB形式 5jel.json.gz
5jel.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/je/5jel
https://data.pdbj.org/pub/pdb/validation_reports/je/5jel ftp://data.pdbj.org/pub/pdb/validation_reports/je/5jel
ftp://data.pdbj.org/pub/pdb/validation_reports/je/5jel リンク
リンク 集合体
集合体

 要素
要素 IRF3 / IRF-3
IRF3 / IRF-3
 Homo sapiens (ヒト) / 遺伝子: IRF3 / 発現宿主:
Homo sapiens (ヒト) / 遺伝子: IRF3 / 発現宿主: 
 Escherichia coli (大腸菌) / 参照: UniProt: Q14653
Escherichia coli (大腸菌) / 参照: UniProt: Q14653
 Homo sapiens (ヒト) / 発現宿主:
Homo sapiens (ヒト) / 発現宿主: 
 Escherichia coli (大腸菌) / 参照: UniProt: Q8IUC6*PLUS
Escherichia coli (大腸菌) / 参照: UniProt: Q8IUC6*PLUS 水
水 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製
 シンクロトロン / サイト:
シンクロトロン / サイト:  ALS
ALS  / ビームライン: 5.0.1 / 波長: 1 Å
/ ビームライン: 5.0.1 / 波長: 1 Å : 1 Å / 相対比: 1
: 1 Å / 相対比: 1  解析
解析 ムービー
ムービー コントローラー
コントローラー

















 PDBj
PDBj







