登録情報 データベース : PDB / ID : 5iefタイトル Murine endoplasmic reticulum alpha-glucosidase II with N-butyl-1-deoxynojirimycin Glucosidase 2 subunit beta Neutral alpha-glucosidase AB キーワード / / 機能・相同性 分子機能 ドメイン・相同性 構成要素
/ / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / 生物種 Mus musculus (ハツカネズミ)手法 / / / 解像度 : 2.38 Å データ登録者 Caputo, A.T. / Roversi, P. / Alonzi, D.S. / Kiappes, J.L. / Zitzmann, N. 資金援助 組織 認可番号 国 Wellcome Trust 097300/Z/11/Z
ジャーナル : Proc.Natl.Acad.Sci.USA / 年 : 2016タイトル : Structures of mammalian ER alpha-glucosidase II capture the binding modes of broad-spectrum iminosugar antivirals.著者 : Caputo, A.T. / Alonzi, D.S. / Marti, L. / Reca, I.B. / Kiappes, J.L. / Struwe, W.B. / Cross, A. / Basu, S. / Lowe, E.D. / Darlot, B. / Santino, A. / Roversi, P. / Zitzmann, N. 履歴 登録 2016年2月25日 登録サイト / 処理サイト 改定 1.0 2016年7月27日 Provider / タイプ 改定 1.1 2016年8月3日 Group 改定 1.2 2016年8月24日 Group 改定 2.0 2020年7月29日 Group Advisory / Atomic model ... Advisory / Atomic model / Data collection / Derived calculations / Refinement description / Structure summary カテゴリ atom_site / chem_comp ... atom_site / chem_comp / entity / pdbx_branch_scheme / pdbx_chem_comp_identifier / pdbx_entity_branch / pdbx_entity_branch_descriptor / pdbx_entity_branch_link / pdbx_entity_branch_list / pdbx_entity_nonpoly / pdbx_nonpoly_scheme / pdbx_struct_assembly_gen / pdbx_struct_conn_angle / pdbx_unobs_or_zero_occ_atoms / software / struct_asym / struct_conn / struct_site / struct_site_gen Item _atom_site.auth_asym_id / _atom_site.auth_seq_id ... _atom_site.auth_asym_id / _atom_site.auth_seq_id / _atom_site.label_asym_id / _chem_comp.name / _chem_comp.type / _entity.formula_weight / _entity.pdbx_description / _entity.pdbx_number_of_molecules / _entity.type / _pdbx_struct_assembly_gen.asym_id_list / _pdbx_struct_conn_angle.ptnr2_label_asym_id / _pdbx_unobs_or_zero_occ_atoms.label_asym_id / _software.name / _struct_conn.conn_type_id / _struct_conn.id / _struct_conn.pdbx_dist_value / _struct_conn.pdbx_leaving_atom_flag / _struct_conn.pdbx_role / _struct_conn.ptnr1_auth_asym_id / _struct_conn.ptnr1_auth_comp_id / _struct_conn.ptnr1_auth_seq_id / _struct_conn.ptnr1_label_asym_id / _struct_conn.ptnr1_label_atom_id / _struct_conn.ptnr1_label_comp_id / _struct_conn.ptnr1_label_seq_id / _struct_conn.ptnr2_auth_asym_id / _struct_conn.ptnr2_auth_comp_id / _struct_conn.ptnr2_auth_seq_id / _struct_conn.ptnr2_label_asym_id / _struct_conn.ptnr2_label_atom_id / _struct_conn.ptnr2_label_comp_id 解説 / Provider / タイプ 改定 2.1 2024年1月10日 Group Data collection / Database references ... Data collection / Database references / Refinement description / Structure summary カテゴリ chem_comp / chem_comp_atom ... chem_comp / chem_comp_atom / chem_comp_bond / database_2 / pdbx_initial_refinement_model Item / _database_2.pdbx_DOI / _database_2.pdbx_database_accession
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード HYDROLASE (加水分解酵素) / Enzyme Glycosyl hydrolase GH31 Quality control exoglycosidase / NB-DNJ
HYDROLASE (加水分解酵素) / Enzyme Glycosyl hydrolase GH31 Quality control exoglycosidase / NB-DNJ 機能・相同性情報
機能・相同性情報 glucan 1,3-alpha-glucosidase activity /
glucan 1,3-alpha-glucosidase activity /  glucosidase II complex /
glucosidase II complex /  alpha-glucosidase activity /
alpha-glucosidase activity /  glucosidase activity / Regulation of Insulin-like Growth Factor (IGF) transport and uptake by Insulin-like Growth Factor Binding Proteins (IGFBPs) /
glucosidase activity / Regulation of Insulin-like Growth Factor (IGF) transport and uptake by Insulin-like Growth Factor Binding Proteins (IGFBPs) /  Post-translational protein phosphorylation / N-glycan processing / liver development /
Post-translational protein phosphorylation / N-glycan processing / liver development /  メラノソーム ...mannosyl-oligosaccharide alpha-1,3-glucosidase /
メラノソーム ...mannosyl-oligosaccharide alpha-1,3-glucosidase /  glucan 1,3-alpha-glucosidase activity /
glucan 1,3-alpha-glucosidase activity /  glucosidase II complex /
glucosidase II complex /  alpha-glucosidase activity /
alpha-glucosidase activity /  glucosidase activity / Regulation of Insulin-like Growth Factor (IGF) transport and uptake by Insulin-like Growth Factor Binding Proteins (IGFBPs) /
glucosidase activity / Regulation of Insulin-like Growth Factor (IGF) transport and uptake by Insulin-like Growth Factor Binding Proteins (IGFBPs) /  Post-translational protein phosphorylation / N-glycan processing / liver development /
Post-translational protein phosphorylation / N-glycan processing / liver development /  メラノソーム / negative regulation of neuron projection development /
メラノソーム / negative regulation of neuron projection development /  carbohydrate binding / in utero embryonic development / carbohydrate metabolic process / intracellular membrane-bounded organelle /
carbohydrate binding / in utero embryonic development / carbohydrate metabolic process / intracellular membrane-bounded organelle /  calcium ion binding / protein-containing complex binding /
calcium ion binding / protein-containing complex binding /  ゴルジ体 /
ゴルジ体 /  小胞体 /
小胞体 /  RNA binding
RNA binding
 Mus musculus (ハツカネズミ)
Mus musculus (ハツカネズミ) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  フーリエ合成 / 解像度: 2.38 Å
フーリエ合成 / 解像度: 2.38 Å  データ登録者
データ登録者 英国, 1件
英国, 1件  引用
引用 ジャーナル: Proc.Natl.Acad.Sci.USA / 年: 2016
ジャーナル: Proc.Natl.Acad.Sci.USA / 年: 2016 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 5ief.cif.gz
5ief.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb5ief.ent.gz
pdb5ief.ent.gz PDB形式
PDB形式 5ief.json.gz
5ief.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/ie/5ief
https://data.pdbj.org/pub/pdb/validation_reports/ie/5ief ftp://data.pdbj.org/pub/pdb/validation_reports/ie/5ief
ftp://data.pdbj.org/pub/pdb/validation_reports/ie/5ief リンク
リンク 集合体
集合体
 要素
要素
 Mus musculus (ハツカネズミ) / 遺伝子: Ganab, G2an, Kiaa0088 / プラスミド: pOPINGS / Cell (発現宿主): ENDOTHELIAL / 細胞株 (発現宿主): HEK 293-F / 器官 (発現宿主): KIDNEY / 発現宿主:
Mus musculus (ハツカネズミ) / 遺伝子: Ganab, G2an, Kiaa0088 / プラスミド: pOPINGS / Cell (発現宿主): ENDOTHELIAL / 細胞株 (発現宿主): HEK 293-F / 器官 (発現宿主): KIDNEY / 発現宿主: 
 Homo sapiens (ヒト) / 参照: UniProt: Q8BHN3, EC: 3.2.1.84
Homo sapiens (ヒト) / 参照: UniProt: Q8BHN3, EC: 3.2.1.84 Glucosidases / 80K-H protein / Glucosidase II subunit beta / Protein kinase C substrate 60.1 kDa protein heavy chain / PKCSH
Glucosidases / 80K-H protein / Glucosidase II subunit beta / Protein kinase C substrate 60.1 kDa protein heavy chain / PKCSH
 Mus musculus (ハツカネズミ) / 遺伝子: Prkcsh / プラスミド: pOPING / Cell (発現宿主): ENDOTHELIAL / 細胞株 (発現宿主): HEK 293-F / 器官 (発現宿主): KIDNEY / 発現宿主:
Mus musculus (ハツカネズミ) / 遺伝子: Prkcsh / プラスミド: pOPING / Cell (発現宿主): ENDOTHELIAL / 細胞株 (発現宿主): HEK 293-F / 器官 (発現宿主): KIDNEY / 発現宿主: 
 Homo sapiens (ヒト) / 参照: UniProt: O08795
Homo sapiens (ヒト) / 参照: UniProt: O08795 オリゴ糖 / 分子量: 424.401 Da / 分子数: 1 / 由来タイプ: 組換発現
オリゴ糖 / 分子量: 424.401 Da / 分子数: 1 / 由来タイプ: 組換発現

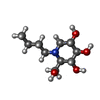








 ギ酸
ギ酸 酢酸塩
酢酸塩 ミグルスタット
ミグルスタット ポリエチレングリコール
ポリエチレングリコール 水
水 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製
 シンクロトロン / サイト:
シンクロトロン / サイト:  ESRF
ESRF  / ビームライン: BM14 / 波長: 0.9788 Å
/ ビームライン: BM14 / 波長: 0.9788 Å : 0.9788 Å / 相対比: 1
: 0.9788 Å / 相対比: 1  解析
解析 :
:  フーリエ合成
フーリエ合成 ムービー
ムービー コントローラー
コントローラー




















 PDBj
PDBj











