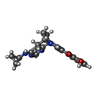
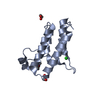
登録情報 データベース : PDB / ID : 5dkhタイトル Crystal structure of the bromodomain of human BRM (SMARCA2) in complex with a hydroxyphenyl propenone inhibitor 17 Probable global transcription activator SNF2L2 キーワード / / / 機能・相同性 分子機能 ドメイン・相同性 構成要素
/ / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / 生物種 Homo sapiens (ヒト)手法 / / / 解像度 : 1.7 Å データ登録者 Tallant, C. / Owen, D.R. / Taylor, A. / Fedorov, O. / Savitsky, P. / Siejka, P. / Srikannathasan, V. / Nowak, R. / von Delft, F. / Arrowsmith, C.H. ...Tallant, C. / Owen, D.R. / Taylor, A. / Fedorov, O. / Savitsky, P. / Siejka, P. / Srikannathasan, V. / Nowak, R. / von Delft, F. / Arrowsmith, C.H. / Edwards, A.M. / Bountra, C. / Knapp, S. ジャーナル : To Be Published タイトル : Crystal structure of the bromodomain of human BRM (SMARCA2) in complex with a hydroxyphenyl propenone inhibitor 17著者 : Tallant, C. / Owen, D.R. / Taylor, A. / Fedorov, O. / Savitsky, P. / Siejka, P. / Srikannathasan, V. / Nowak, R. / von Delft, F. / Arrowsmith, C.H. / Edwards, A.M. / Bountra, C. / Knapp, S. 履歴 登録 2015年9月3日 登録サイト / 処理サイト 改定 1.0 2016年9月14日 Provider / タイプ 改定 1.1 2024年1月10日 Group Data collection / Database references ... Data collection / Database references / Derived calculations / Refinement description カテゴリ chem_comp_atom / chem_comp_bond ... chem_comp_atom / chem_comp_bond / database_2 / pdbx_initial_refinement_model / pdbx_struct_conn_angle / struct_conn Item _database_2.pdbx_DOI / _database_2.pdbx_database_accession ... _database_2.pdbx_DOI / _database_2.pdbx_database_accession / _pdbx_struct_conn_angle.ptnr1_auth_asym_id / _pdbx_struct_conn_angle.ptnr1_auth_comp_id / _pdbx_struct_conn_angle.ptnr1_auth_seq_id / _pdbx_struct_conn_angle.ptnr1_label_asym_id / _pdbx_struct_conn_angle.ptnr1_label_atom_id / _pdbx_struct_conn_angle.ptnr1_label_comp_id / _pdbx_struct_conn_angle.ptnr1_label_seq_id / _pdbx_struct_conn_angle.ptnr1_symmetry / _pdbx_struct_conn_angle.ptnr2_auth_asym_id / _pdbx_struct_conn_angle.ptnr2_auth_seq_id / _pdbx_struct_conn_angle.ptnr2_label_asym_id / _pdbx_struct_conn_angle.ptnr2_symmetry / _pdbx_struct_conn_angle.ptnr3_auth_asym_id / _pdbx_struct_conn_angle.ptnr3_auth_comp_id / _pdbx_struct_conn_angle.ptnr3_auth_seq_id / _pdbx_struct_conn_angle.ptnr3_label_asym_id / _pdbx_struct_conn_angle.ptnr3_label_atom_id / _pdbx_struct_conn_angle.ptnr3_label_comp_id / _pdbx_struct_conn_angle.ptnr3_label_seq_id / _pdbx_struct_conn_angle.ptnr3_symmetry / _pdbx_struct_conn_angle.value / _struct_conn.pdbx_dist_value / _struct_conn.ptnr1_auth_asym_id / _struct_conn.ptnr1_auth_comp_id / _struct_conn.ptnr1_auth_seq_id / _struct_conn.ptnr1_label_asym_id / _struct_conn.ptnr1_label_atom_id / _struct_conn.ptnr1_label_comp_id / _struct_conn.ptnr1_label_seq_id / _struct_conn.ptnr2_auth_asym_id / _struct_conn.ptnr2_auth_comp_id / _struct_conn.ptnr2_auth_seq_id / _struct_conn.ptnr2_label_asym_id / _struct_conn.ptnr2_label_atom_id / _struct_conn.ptnr2_label_comp_id / _struct_conn.ptnr2_symmetry
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード TRANSCRIPTION (転写 (生物学)) / SWI-SNF complex /
TRANSCRIPTION (転写 (生物学)) / SWI-SNF complex /  chromatin remodeling (クロマチンリモデリング) / Brg associated factors (BAF)
chromatin remodeling (クロマチンリモデリング) / Brg associated factors (BAF) 機能・相同性情報
機能・相同性情報 brahma complex /
brahma complex /  nBAF complex / GBAF complex / regulation of G0 to G1 transition / regulation of nucleotide-excision repair / ATP-dependent chromatin remodeler activity / regulation of mitotic metaphase/anaphase transition /
nBAF complex / GBAF complex / regulation of G0 to G1 transition / regulation of nucleotide-excision repair / ATP-dependent chromatin remodeler activity / regulation of mitotic metaphase/anaphase transition /  SWI/SNF complex ...bBAF complex / npBAF complex /
SWI/SNF complex ...bBAF complex / npBAF complex /  brahma complex /
brahma complex /  nBAF complex / GBAF complex / regulation of G0 to G1 transition / regulation of nucleotide-excision repair / ATP-dependent chromatin remodeler activity / regulation of mitotic metaphase/anaphase transition /
nBAF complex / GBAF complex / regulation of G0 to G1 transition / regulation of nucleotide-excision repair / ATP-dependent chromatin remodeler activity / regulation of mitotic metaphase/anaphase transition /  SWI/SNF complex / positive regulation of double-strand break repair / positive regulation of T cell differentiation / intermediate filament cytoskeleton / positive regulation of stem cell population maintenance / RUNX1 interacts with co-factors whose precise effect on RUNX1 targets is not known / regulation of G1/S transition of mitotic cell cycle / negative regulation of cell differentiation / spermatid development / positive regulation of myoblast differentiation / ATP-dependent activity, acting on DNA /
SWI/SNF complex / positive regulation of double-strand break repair / positive regulation of T cell differentiation / intermediate filament cytoskeleton / positive regulation of stem cell population maintenance / RUNX1 interacts with co-factors whose precise effect on RUNX1 targets is not known / regulation of G1/S transition of mitotic cell cycle / negative regulation of cell differentiation / spermatid development / positive regulation of myoblast differentiation / ATP-dependent activity, acting on DNA /  helicase activity / positive regulation of cell differentiation /
helicase activity / positive regulation of cell differentiation /  加水分解酵素; 酸無水物に作用; 酸無水物に作用・細胞または細胞小器官の運動に関与 / negative regulation of cell growth / RMTs methylate histone arginines /
加水分解酵素; 酸無水物に作用; 酸無水物に作用・細胞または細胞小器官の運動に関与 / negative regulation of cell growth / RMTs methylate histone arginines /  nervous system development /
nervous system development /  histone binding /
histone binding /  transcription coactivator activity /
transcription coactivator activity /  hydrolase activity / transcription cis-regulatory region binding /
hydrolase activity / transcription cis-regulatory region binding /  クロマチンリモデリング / negative regulation of cell population proliferation / intracellular membrane-bounded organelle / negative regulation of DNA-templated transcription /
クロマチンリモデリング / negative regulation of cell population proliferation / intracellular membrane-bounded organelle / negative regulation of DNA-templated transcription /  chromatin binding /
chromatin binding /  クロマチン / positive regulation of cell population proliferation / regulation of DNA-templated transcription / regulation of transcription by RNA polymerase II / positive regulation of DNA-templated transcription / negative regulation of transcription by RNA polymerase II / positive regulation of transcription by RNA polymerase II /
クロマチン / positive regulation of cell population proliferation / regulation of DNA-templated transcription / regulation of transcription by RNA polymerase II / positive regulation of DNA-templated transcription / negative regulation of transcription by RNA polymerase II / positive regulation of transcription by RNA polymerase II /  DNA binding /
DNA binding /  核質 /
核質 /  ATP binding /
ATP binding /  細胞核
細胞核
 Homo sapiens (ヒト)
Homo sapiens (ヒト) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 1.7 Å
分子置換 / 解像度: 1.7 Å  データ登録者
データ登録者 引用
引用 ジャーナル: To Be Published
ジャーナル: To Be Published 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 5dkh.cif.gz
5dkh.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb5dkh.ent.gz
pdb5dkh.ent.gz PDB形式
PDB形式 5dkh.json.gz
5dkh.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/dk/5dkh
https://data.pdbj.org/pub/pdb/validation_reports/dk/5dkh ftp://data.pdbj.org/pub/pdb/validation_reports/dk/5dkh
ftp://data.pdbj.org/pub/pdb/validation_reports/dk/5dkh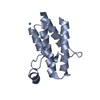
 リンク
リンク 集合体
集合体



 要素
要素
 Homo sapiens (ヒト) / 遺伝子: SMARCA2, BAF190B, BRM, SNF2A, SNF2L2 / プラスミド: pNIC28-Bsa4 / 発現宿主:
Homo sapiens (ヒト) / 遺伝子: SMARCA2, BAF190B, BRM, SNF2A, SNF2L2 / プラスミド: pNIC28-Bsa4 / 発現宿主: 
 Escherichia coli (大腸菌)
Escherichia coli (大腸菌) 加水分解酵素; 酸無水物に作用; 酸無水物に作用・細胞または細胞小器官の運動に関与
加水分解酵素; 酸無水物に作用; 酸無水物に作用・細胞または細胞小器官の運動に関与 エチレングリコール
エチレングリコール 水
水 X線回折
X線回折 試料調製
試料調製
 シンクロトロン / サイト:
シンクロトロン / サイト:  Diamond
Diamond  / ビームライン: I02 / 波長: 0.9795 Å
/ ビームライン: I02 / 波長: 0.9795 Å : 0.9795 Å / 相対比: 1
: 0.9795 Å / 相対比: 1  解析
解析 :
:  分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー











 PDBj
PDBj