+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 5d67 | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | Crystal structure of an EF-Hand calcium binding domain of CAP-Binding Protein Complex-Interacting Protein 1 (EFCAB6) from Homo sapiens at 2.00 A resolution | ||||||
 要素 要素 | EF-hand calcium-binding domain-containing protein 6 | ||||||
 キーワード キーワード | METAL BINDING PROTEIN /  Structural Genomics (構造ゲノミクス) / Joint Center for Structural Genomics / JCSG / Structural Genomics (構造ゲノミクス) / Joint Center for Structural Genomics / JCSG /  Protein Structure Initiative / PSI-BIOLOGY / Partnership for Nuclear Receptor Signaling Code Biology / NHRS Protein Structure Initiative / PSI-BIOLOGY / Partnership for Nuclear Receptor Signaling Code Biology / NHRS | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 | ||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  単波長異常分散 / 解像度: 2 Å 単波長異常分散 / 解像度: 2 Å | ||||||
 データ登録者 データ登録者 | Joint Center for Structural Genomics (JCSG) / Partnership for Nuclear Receptor Signaling Code Biology / Partnership for Nuclear Receptor Signaling Code Biology (NHRS) | ||||||
 引用 引用 |  ジャーナル: To be published ジャーナル: To be publishedタイトル: Crystal structure of an EF-Hand calcium binding domain of CAP-Binding Protein Complex-Interacting Protein 1 (EFCAB6) from Homo sapiens at 2.00 A resolution 著者: Joint Center for Structural Genomics (JCSG) / Partnership for Nuclear Receptor Signaling Code Biology | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  5d67.cif.gz 5d67.cif.gz | 164.7 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb5d67.ent.gz pdb5d67.ent.gz | 133.4 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  5d67.json.gz 5d67.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/d6/5d67 https://data.pdbj.org/pub/pdb/validation_reports/d6/5d67 ftp://data.pdbj.org/pub/pdb/validation_reports/d6/5d67 ftp://data.pdbj.org/pub/pdb/validation_reports/d6/5d67 | HTTPS FTP |
|---|
-関連構造データ
| 類似構造データ | |
|---|---|
| その他のデータベース |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 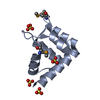
| ||||||||
| 2 | 
| ||||||||
| 3 | 
| ||||||||
| 4 | 
| ||||||||
| 単位格子 |
|
- 要素
要素
| #1: タンパク質 | 分子量: 11778.729 Da / 分子数: 4 / 断片: UNP residues 1160-1260 / 変異: D1218G / 由来タイプ: 組換発現 / 由来: (組換発現)   Homo sapiens (ヒト) / 遺伝子: EFCAB6, DJBP, KIAA1672 / プラスミド: SpeedET / 発現宿主: Homo sapiens (ヒト) / 遺伝子: EFCAB6, DJBP, KIAA1672 / プラスミド: SpeedET / 発現宿主:   Escherichia Coli (大腸菌) / 株 (発現宿主): PB1 / 参照: UniProt: Q5THR3 Escherichia Coli (大腸菌) / 株 (発現宿主): PB1 / 参照: UniProt: Q5THR3#2: 化合物 | ChemComp-SO4 /  硫酸塩 硫酸塩#3: 水 | ChemComp-HOH / |  水 水 |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.15 Å3/Da / 溶媒含有率: 42.91 % |
|---|---|
結晶化 | 温度: 277 K / 手法: 蒸気拡散法, シッティングドロップ法 / 詳細: 30% polyethylene glycol 4000, 0.2M ammonium sulfate |
-データ収集
| 回折 | 平均測定温度: 100 K | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  SSRL SSRL  / ビームライン: BL14-1 / 波長: 0.97917 Å / ビームライン: BL14-1 / 波長: 0.97917 Å | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 検出器 | タイプ: MARMOSAIC 325 mm CCD / 検出器: CCD / 日付: 2015年3月8日 詳細: Vertical focusing mirror; double crystal Si(111) monochromator | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 放射 | モノクロメーター: double crystal Si(111) / プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 放射波長 | 波長 : 0.97917 Å / 相対比: 1 : 0.97917 Å / 相対比: 1 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 反射 | 解像度: 2→27.118 Å / Num. obs: 26723 / % possible obs: 98.1 % / Observed criterion σ(I): -3 / 冗長度: 3.787 % / Biso Wilson estimate: 26.457 Å2 / Rmerge F obs: 0.996 / Rmerge(I) obs: 0.112 / Rrim(I) all: 0.13 / Net I/σ(I): 10.59 / Num. measured all: 197737 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 反射 シェル |
|
-位相決定
位相決定 | 手法:  単波長異常分散 単波長異常分散 |
|---|
- 解析
解析
| ソフトウェア |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法 : :  単波長異常分散 / 解像度: 2→27.118 Å / Cor.coef. Fo:Fc: 0.953 / Cor.coef. Fo:Fc free: 0.9373 / Occupancy max: 1 / Occupancy min: 0.37 / 交差検証法: THROUGHOUT / σ(F): 0 単波長異常分散 / 解像度: 2→27.118 Å / Cor.coef. Fo:Fc: 0.953 / Cor.coef. Fo:Fc free: 0.9373 / Occupancy max: 1 / Occupancy min: 0.37 / 交差検証法: THROUGHOUT / σ(F): 0 詳細: 1. ATOM RECORDS CONTAIN SUM OF TLS AND RESIDUAL B FACTORS. 2. ANISOU RECORDS CONTAIN SUM OF TLS AND RESIDUAL U FACTORS. 3. A MET-INHIBITION PROTOCOL WAS USED FOR SELENOMETHIONINE ...詳細: 1. ATOM RECORDS CONTAIN SUM OF TLS AND RESIDUAL B FACTORS. 2. ANISOU RECORDS CONTAIN SUM OF TLS AND RESIDUAL U FACTORS. 3. A MET-INHIBITION PROTOCOL WAS USED FOR SELENOMETHIONINE INCORPORATION DURING PROTEIN EXPRESSION. THE OCCUPANCY OF THE SE ATOMS IN THE MSE RESIDUES WAS REDUCED TO 0.75 FOR THE REDUCED SCATTERING POWER DUE TO PARTIAL S-MET INCORPORATION. 4. THE SAD PHASES WERE USED AS RESTRAINTS DURING REFINEMENT
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso max: 161.22 Å2 / Biso mean: 35.3461 Å2 / Biso min: 10.12 Å2
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine analyze | Luzzati coordinate error obs: 0.209 Å | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 2→27.118 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル | 解像度: 2→2.08 Å / Total num. of bins used: 13
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLS | 手法: refined / Refine-ID: X-RAY DIFFRACTION
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLSグループ |
|
 ムービー
ムービー コントローラー
コントローラー












 PDBj
PDBj




