+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 4wyt | ||||||
|---|---|---|---|---|---|---|---|
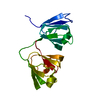
| タイトル | Crystal Structure of Scribble PDZ34 tandem at 2.6 Angstroms | ||||||
 要素 要素 | Protein scribble homolog | ||||||
 キーワード キーワード |  STRUCTURAL PROTEIN (タンパク質) / PDZ tandem STRUCTURAL PROTEIN (タンパク質) / PDZ tandem | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 extrinsic component of postsynaptic density membrane / neurotransmitter receptor transport postsynaptic membrane to endosome / establishment of T cell polarity / apoptotic process involved in morphogenesis / establishment of apical/basal cell polarity / cochlear nucleus development / synaptic vesicle targeting / Scrib-APC-beta-catenin complex / astrocyte cell migration / polarized epithelial cell differentiation ... extrinsic component of postsynaptic density membrane / neurotransmitter receptor transport postsynaptic membrane to endosome / establishment of T cell polarity / apoptotic process involved in morphogenesis / establishment of apical/basal cell polarity / cochlear nucleus development / synaptic vesicle targeting / Scrib-APC-beta-catenin complex / astrocyte cell migration / polarized epithelial cell differentiation ... extrinsic component of postsynaptic density membrane / neurotransmitter receptor transport postsynaptic membrane to endosome / establishment of T cell polarity / apoptotic process involved in morphogenesis / establishment of apical/basal cell polarity / cochlear nucleus development / synaptic vesicle targeting / Scrib-APC-beta-catenin complex / astrocyte cell migration / polarized epithelial cell differentiation / myelin sheath abaxonal region / neurotransmitter receptor transport, endosome to postsynaptic membrane / protein localization to adherens junction / cell-cell contact zone / mammary gland duct morphogenesis / post-anal tail morphogenesis / activation of GTPase activity / establishment or maintenance of epithelial cell apical/basal polarity / regulation of postsynaptic neurotransmitter receptor internalization / auditory receptor cell stereocilium organization / RND2 GTPase cycle / RND3 GTPase cycle / positive regulation of receptor recycling / positive chemotaxis / RHOJ GTPase cycle / RHOQ GTPase cycle / receptor clustering / negative regulation of activated T cell proliferation / CDC42 GTPase cycle / synaptic vesicle endocytosis / extrinsic component of postsynaptic density membrane / neurotransmitter receptor transport postsynaptic membrane to endosome / establishment of T cell polarity / apoptotic process involved in morphogenesis / establishment of apical/basal cell polarity / cochlear nucleus development / synaptic vesicle targeting / Scrib-APC-beta-catenin complex / astrocyte cell migration / polarized epithelial cell differentiation / myelin sheath abaxonal region / neurotransmitter receptor transport, endosome to postsynaptic membrane / protein localization to adherens junction / cell-cell contact zone / mammary gland duct morphogenesis / post-anal tail morphogenesis / activation of GTPase activity / establishment or maintenance of epithelial cell apical/basal polarity / regulation of postsynaptic neurotransmitter receptor internalization / auditory receptor cell stereocilium organization / RND2 GTPase cycle / RND3 GTPase cycle / positive regulation of receptor recycling / positive chemotaxis / RHOJ GTPase cycle / RHOQ GTPase cycle / receptor clustering / negative regulation of activated T cell proliferation / CDC42 GTPase cycle / synaptic vesicle endocytosis /  免疫シナプス / negative regulation of mitotic cell cycle / signaling adaptor activity / Asymmetric localization of PCP proteins / neural tube closure / 免疫シナプス / negative regulation of mitotic cell cycle / signaling adaptor activity / Asymmetric localization of PCP proteins / neural tube closure /  接着結合 / 接着結合 /  wound healing / wound healing /  細胞接着 / positive regulation of type II interferon production / cell-cell junction / 細胞接着 / positive regulation of type II interferon production / cell-cell junction /  遊走 / presynapse / 遊走 / presynapse /  細胞結合 / 細胞結合 /  lamellipodium / basolateral plasma membrane / cell population proliferation / lamellipodium / basolateral plasma membrane / cell population proliferation /  postsynaptic density / postsynaptic density /  cadherin binding / positive regulation of apoptotic process / glutamatergic synapse / extracellular exosome / cadherin binding / positive regulation of apoptotic process / glutamatergic synapse / extracellular exosome /  核質 / 核質 /  細胞膜 細胞膜類似検索 - 分子機能 | ||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2.6 Å 分子置換 / 解像度: 2.6 Å | ||||||
 データ登録者 データ登録者 | Ren, J.Q. / Feng, W. | ||||||
| 資金援助 |  中国, 1件 中国, 1件
| ||||||
 引用 引用 |  ジャーナル: Biochem.J. / 年: 2015 ジャーナル: Biochem.J. / 年: 2015タイトル: Interdomain interface-mediated target recognition by the Scribble PDZ34 supramodule. 著者: Ren, J. / Feng, L. / Bai, Y. / Pei, H. / Yuan, Z. / Feng, W. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  4wyt.cif.gz 4wyt.cif.gz | 90.5 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb4wyt.ent.gz pdb4wyt.ent.gz | 67.4 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  4wyt.json.gz 4wyt.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/wy/4wyt https://data.pdbj.org/pub/pdb/validation_reports/wy/4wyt ftp://data.pdbj.org/pub/pdb/validation_reports/wy/4wyt ftp://data.pdbj.org/pub/pdb/validation_reports/wy/4wyt | HTTPS FTP |
|---|
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| 単位格子 |
| ||||||||
| 詳細 | There are 2 biological units in the asymmetric unit (chains A & D and chains B & C) |
- 要素
要素
| #1: タンパク質 | 分子量: 22172.270 Da / 分子数: 1 / 断片: PDZ3/PDZ4 tandem (UNP RESIDUES 992-1203) / 由来タイプ: 組換発現 / 由来: (組換発現)   Homo sapiens (ヒト) / 遺伝子: SCRIB, CRIB1, KIAA0147, LAP4, SCRB1, VARTUL / プラスミド: pET32a / 発現宿主: Homo sapiens (ヒト) / 遺伝子: SCRIB, CRIB1, KIAA0147, LAP4, SCRB1, VARTUL / プラスミド: pET32a / 発現宿主:   Escherichia coli (大腸菌) / 株 (発現宿主): BL21(DE3) / 参照: UniProt: Q14160 Escherichia coli (大腸菌) / 株 (発現宿主): BL21(DE3) / 参照: UniProt: Q14160 | ||
|---|---|---|---|
| #2: 化合物 |  塩化物 塩化物#3: 水 | ChemComp-HOH / |  水 水 |
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.55 Å3/Da / 溶媒含有率: 51.79 % |
|---|---|
結晶化 | 温度: 298 K / 手法: 蒸気拡散法, シッティングドロップ法 / pH: 7.5 / 詳細: 3.0 M sodium chloride, 0.1M Tris-Hcl |
-データ収集
| 回折 | 平均測定温度: 100 K | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  SSRF SSRF  / ビームライン: BL17U / 波長: 0.9792 Å / ビームライン: BL17U / 波長: 0.9792 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 検出器 | タイプ: MARMOSAIC 225 mm CCD / 検出器: CCD / 日付: 2010年11月11日 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 放射波長 | 波長 : 0.9792 Å / 相対比: 1 : 0.9792 Å / 相対比: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 反射 | 解像度: 2.6→49.483 Å / Num. all: 7301 / Num. obs: 7301 / % possible obs: 100 % / 冗長度: 6 % / Biso Wilson estimate: 40.95 Å2 / Rpim(I) all: 0.055 / Rrim(I) all: 0.136 / Rsym value: 0.124 / Net I/av σ(I): 4.79 / Net I/σ(I): 10.2 / Num. measured all: 44038 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 反射 シェル | Diffraction-ID: 1 / Rejects: 0
|
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法 : :  分子置換 分子置換開始モデル: 1TP3 解像度: 2.6→43.658 Å / SU ML: 0.4 / 交差検証法: FREE R-VALUE / σ(F): 1.34 / 位相誤差: 29.35 / 立体化学のターゲット値: ML
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | 減衰半径: 0.9 Å / VDWプローブ半径: 1.11 Å / 溶媒モデル: FLAT BULK SOLVENT MODEL | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso max: 145.97 Å2 / Biso mean: 47.6748 Å2 / Biso min: 21.76 Å2 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: final / 解像度: 2.6→43.658 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル | Refine-ID: X-RAY DIFFRACTION / Total num. of bins used: 2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLS | 手法: refined / Refine-ID: X-RAY DIFFRACTION
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLSグループ |
|
 ムービー
ムービー コントローラー
コントローラー














 PDBj
PDBj











