登録情報 データベース : PDB / ID : 4rojタイトル Crystal Structure of the VAV2 SH2 domain in complex with TXNIP phosphorylated peptide Guanine nucleotide exchange factor VAV2 Thioredoxin-interacting protein キーワード / / / / 機能・相同性 分子機能 ドメイン・相同性 構成要素
/ / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / 生物種 Homo sapiens (ヒト)手法 / / 解像度 : 1.95 Å データ登録者 Liu, Y. / Tempel, W. / Bountra, C. / Arrowsmith, C.H. / Edwards, A.M. / Min, J. / Structural Genomics Consortium (SGC) ジャーナル : TO BE PUBLISHED タイトル : Crystal Structure of the VAV2 SH2 domain in complex with TXNIP phosphorylated peptide著者 : Liu, Y. / Tempel, W. / Bountra, C. / Arrowsmith, C.H. / Edwards, A.M. / Min, J. / Structural Genomics Consortium (SGC) 履歴 登録 2014年10月28日 登録サイト / 処理サイト 改定 1.0 2014年12月10日 Provider / タイプ 改定 1.1 2023年9月20日 Group Data collection / Database references ... Data collection / Database references / Derived calculations / Refinement description カテゴリ chem_comp_atom / chem_comp_bond ... chem_comp_atom / chem_comp_bond / database_2 / pdbx_initial_refinement_model / struct_conn / struct_ref_seq_dif Item _database_2.pdbx_DOI / _database_2.pdbx_database_accession ... _database_2.pdbx_DOI / _database_2.pdbx_database_accession / _struct_conn.pdbx_dist_value / _struct_conn.pdbx_leaving_atom_flag / _struct_conn.ptnr1_auth_asym_id / _struct_conn.ptnr1_auth_seq_id / _struct_conn.ptnr1_label_asym_id / _struct_conn.ptnr1_label_seq_id / _struct_conn.ptnr1_symmetry / _struct_conn.ptnr2_auth_asym_id / _struct_conn.ptnr2_auth_seq_id / _struct_conn.ptnr2_label_asym_id / _struct_conn.ptnr2_label_seq_id / _struct_conn.ptnr2_symmetry / _struct_ref_seq_dif.details 改定 1.2 2023年12月6日 Group / カテゴリ / chem_comp_bond / Item / _chem_comp_bond.atom_id_2
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード LIGASE (リガーゼ) /
LIGASE (リガーゼ) /  structural genomics (構造ゲノミクス) /
structural genomics (構造ゲノミクス) /  SH2 domain (SH2ドメイン) /
SH2 domain (SH2ドメイン) /  Structural Genomics Consortium / SGC
Structural Genomics Consortium / SGC 機能・相同性情報
機能・相同性情報 enzyme inhibitor activity / regulation of small GTPase mediated signal transduction / Regulation of FOXO transcriptional activity by acetylation /
enzyme inhibitor activity / regulation of small GTPase mediated signal transduction / Regulation of FOXO transcriptional activity by acetylation /  lamellipodium assembly /
lamellipodium assembly /  regulation of cell size /
regulation of cell size /  epidermal growth factor receptor binding ...cellular response to tumor cell / cellular response to oxidised low-density lipoprotein particle stimulus / negative regulation of cell division / Azathioprine ADME /
epidermal growth factor receptor binding ...cellular response to tumor cell / cellular response to oxidised low-density lipoprotein particle stimulus / negative regulation of cell division / Azathioprine ADME /  enzyme inhibitor activity / regulation of small GTPase mediated signal transduction / Regulation of FOXO transcriptional activity by acetylation /
enzyme inhibitor activity / regulation of small GTPase mediated signal transduction / Regulation of FOXO transcriptional activity by acetylation /  lamellipodium assembly /
lamellipodium assembly /  regulation of cell size /
regulation of cell size /  epidermal growth factor receptor binding / small GTPase-mediated signal transduction / RHOB GTPase cycle / NRAGE signals death through JNK /
epidermal growth factor receptor binding / small GTPase-mediated signal transduction / RHOB GTPase cycle / NRAGE signals death through JNK /  regulation of GTPase activity / Fc-gamma receptor signaling pathway involved in phagocytosis / RHOC GTPase cycle / Fc-epsilon receptor signaling pathway / CDC42 GTPase cycle / platelet-derived growth factor receptor signaling pathway / The NLRP3 inflammasome / RHOG GTPase cycle / EPH-ephrin mediated repulsion of cells / RHOA GTPase cycle / RAC2 GTPase cycle / RAC3 GTPase cycle / vascular endothelial growth factor receptor signaling pathway / response to glucose /
regulation of GTPase activity / Fc-gamma receptor signaling pathway involved in phagocytosis / RHOC GTPase cycle / Fc-epsilon receptor signaling pathway / CDC42 GTPase cycle / platelet-derived growth factor receptor signaling pathway / The NLRP3 inflammasome / RHOG GTPase cycle / EPH-ephrin mediated repulsion of cells / RHOA GTPase cycle / RAC2 GTPase cycle / RAC3 GTPase cycle / vascular endothelial growth factor receptor signaling pathway / response to glucose /  Purinergic signaling in leishmaniasis infection / response to mechanical stimulus / keratinocyte differentiation / GPVI-mediated activation cascade / RAC1 GTPase cycle / phosphotyrosine residue binding / FCERI mediated Ca+2 mobilization / guanyl-nucleotide exchange factor activity / VEGFR2 mediated vascular permeability / Signal transduction by L1 / response to progesterone / FCERI mediated MAPK activation / FCGR3A-mediated phagocytosis / response to hydrogen peroxide / Cytoprotection by HMOX1 / Regulation of actin dynamics for phagocytic cup formation /
Purinergic signaling in leishmaniasis infection / response to mechanical stimulus / keratinocyte differentiation / GPVI-mediated activation cascade / RAC1 GTPase cycle / phosphotyrosine residue binding / FCERI mediated Ca+2 mobilization / guanyl-nucleotide exchange factor activity / VEGFR2 mediated vascular permeability / Signal transduction by L1 / response to progesterone / FCERI mediated MAPK activation / FCGR3A-mediated phagocytosis / response to hydrogen peroxide / Cytoprotection by HMOX1 / Regulation of actin dynamics for phagocytic cup formation /  凝固・線溶系 / VEGFA-VEGFR2 Pathway / response to calcium ion / protein import into nucleus / G alpha (12/13) signalling events /
凝固・線溶系 / VEGFA-VEGFR2 Pathway / response to calcium ion / protein import into nucleus / G alpha (12/13) signalling events /  遊走 / cellular response to xenobiotic stimulus /
遊走 / cellular response to xenobiotic stimulus /  protein transport / DAP12 signaling / response to estradiol / regulation of cell population proliferation /
protein transport / DAP12 signaling / response to estradiol / regulation of cell population proliferation /  血管新生 / positive regulation of phosphatidylinositol 3-kinase/protein kinase B signal transduction / response to xenobiotic stimulus /
血管新生 / positive regulation of phosphatidylinositol 3-kinase/protein kinase B signal transduction / response to xenobiotic stimulus /  炎症 / positive regulation of apoptotic process /
炎症 / positive regulation of apoptotic process /  細胞周期 /
細胞周期 /  ubiquitin protein ligase binding / negative regulation of transcription by RNA polymerase II /
ubiquitin protein ligase binding / negative regulation of transcription by RNA polymerase II /  シグナル伝達 /
シグナル伝達 /  metal ion binding /
metal ion binding /  細胞膜 /
細胞膜 /  細胞質基質 /
細胞質基質 /  細胞質
細胞質
 Homo sapiens (ヒト)
Homo sapiens (ヒト) X線回折 /
X線回折 /  分子置換 / 解像度: 1.95 Å
分子置換 / 解像度: 1.95 Å  データ登録者
データ登録者 引用
引用 ジャーナル: TO BE PUBLISHED
ジャーナル: TO BE PUBLISHED 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 4roj.cif.gz
4roj.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb4roj.ent.gz
pdb4roj.ent.gz PDB形式
PDB形式 4roj.json.gz
4roj.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/ro/4roj
https://data.pdbj.org/pub/pdb/validation_reports/ro/4roj ftp://data.pdbj.org/pub/pdb/validation_reports/ro/4roj
ftp://data.pdbj.org/pub/pdb/validation_reports/ro/4roj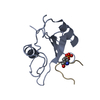
 リンク
リンク 集合体
集合体

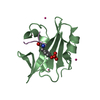

 要素
要素
 Homo sapiens (ヒト) / 遺伝子: VAV2 / プラスミド: pET28-MHL / 発現宿主:
Homo sapiens (ヒト) / 遺伝子: VAV2 / プラスミド: pET28-MHL / 発現宿主: 
 Escherichia coli (大腸菌) / 株 (発現宿主): BL21(DE3)-V2R-pRARE2 / 参照: UniProt: P52735
Escherichia coli (大腸菌) / 株 (発現宿主): BL21(DE3)-V2R-pRARE2 / 参照: UniProt: P52735
 Homo sapiens (ヒト) / 参照: UniProt: Q9H3M7
Homo sapiens (ヒト) / 参照: UniProt: Q9H3M7 水
水 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製
 回転陽極 / タイプ: RIGAKU FR-E SUPERBRIGHT / 波長: 1.5418 Å
回転陽極 / タイプ: RIGAKU FR-E SUPERBRIGHT / 波長: 1.5418 Å : 1.5418 Å / 相対比: 1
: 1.5418 Å / 相対比: 1 
 分子置換
分子置換 解析
解析 :
:  分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー













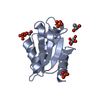


 PDBj
PDBj

























